MÉTODO DE DETECCIÓN DE SECUENCIAS DE ÁCIDO NUCLEICO Y SUSTRATO PARA DETECCIÓN DE SECUENCIAS DE ÁCIDO NUCLEICO.
Método de detección de secuencias de ácido nucleico para detectar la presencia de una secuencia de ácido nucleico diana particular (10) que va a detectarse en una muestra,
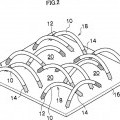
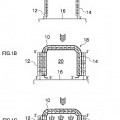
comprendiendo el método: una etapa de inmovilización de anclar cadenas de oligonucleótidos (12, 14) sobre un sustrato (16) mediante la inmovilización de uno de los extremos 5' y 3' de las mismas sobre el sustrato; una etapa de formación de espacio reticulado de llevar a cabo, basándose en un conjunto de las cadenas de oligonucleótidos (12, 14), la etapa de inmovilización y unir las cadenas de oligonucleótidos a sus respectivas partes complementarias de la secuencia de ácido nucleico diana (10) para formar una estructura entrecruzada en múltiples conjuntos para formar un espacio finamente reticulado (20) en el sustrato, en la que una pluralidad de las estructuras entrecruzadas se entrelazan entre sí; una etapa de captura de capturar un ligando (22) por el espacio reticulado (20) a través de adsorción física; una etapa de coloración de hacer que el ligando capturado (22) se coloree con una sustancia activa reactiva con el ligando; y una etapa de detección de detectar una señal de color derivada de la coloración para detectar la secuencia de ácido nucleico diana (10)
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E08019285.
Solicitante: HITACHI PLANT TECHNOLOGIES, LTD.
Nacionalidad solicitante: Japón.
Dirección: 5-2 HIGASHI-IKEBUKURO 4-CHOME TOSHIMA-KU TOKYO JAPON.
Inventor/es: Ikuta,Hajime, Takemura,Kiyokazu.
Fecha de Publicación: .
Fecha Solicitud PCT: 4 de Noviembre de 2008.
Clasificación Internacional de Patentes:
- C12Q1/68B10A
- C12Q1/68B2D
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
PDF original: ES-2356299_T3.pdf


Fragmento de la descripción:
ANTECEDENTES DE LA INVENCIÓN
Campo de la invención
La presente invención se refiere a un método de detección de secuencias de ácido nucleico. 5 Particularmente, la presente invención se refiere a una técnica para detectar de manera altamente sensible, convenientemente y a bajo coste, la presencia de una secuencia de ácido nucleico diana particular que va a detectarse en una muestra.
Descripción de la técnica relacionada
Un método convencional para detectar una secuencia de ácido nucleico específica para una muestra como 10 diana implica: unir la secuencia de ácido nucleico diana a cadenas de oligonucleótidos inmovilizadas en un sustrato y unir una cadena de oligonucleótidos diferente marcada con una sustancia fluorescente a la secuencia de ácido nucleico diana unida; y detectar la presencia de la secuencia de ácido nucleico particular en la muestra detectando/amplificando una señal de luz reflejada del marcador de sustancia fluorescente mediante irradiación con luz (por ejemplo, haz de láser) a una longitud de onda particular. Otro método de detección implica: unir la secuencia 15 de ácido nucleico diana a cadenas de oligonucleótidos inmovilizadas en un sustrato; llevar a cabo una reacción de elongación usando las cadenas de oligonucleótidos como cebadores y la secuencia de ácido nucleico diana unida como molde, en el que se marcan las sustancias de base incorporadas durante la reacción con una sustancia fluorescente; y detectar/amplificar una señal de luz reflejada del marcador de sustancia fluorescente mediante irradiación con luz (por ejemplo, haz de láser) a una longitud de onda particular. 20
Sin embargo, estos métodos de detección deben detectar una sustancia fluorescente y por tanto requieren un aparato de detección especial que emite luz a una longitud de onda particular. Un aparato de detección de este tipo es costoso y tiene un uso limitado para ciertos institutos o universidades de investigación en las presentes circunstancias. Por tanto, estos enfoques no pueden adoptarse, por ejemplo, para detectar convenientemente microorganismos patógenos tales como norovirus o Cryptosporidium en agua (muestra) en sitios de tratamiento de 25 agua, debido a procedimientos complicados y aparatos de análisis costosos.
Frente a este telón de fondo, por ejemplo, el método MPEX (extensión con múltiples cebadores) descrito en K. Kinoshita et al., Multiple primer extension by DNA polimerase on a novel plastic DNA array coated with a biocompatible polimer, Nucleic Acid Research, vol. 35, n.º 1, 2007, págs. e3, y solicitudes de patentes japonesas abiertas a consulta por el público n.os 2006-174788 y 2007-222010 se ha desarrollado como un método para detectar 30 convenientemente una secuencia de ácido nucleico particular en detección (por ejemplo, sitios de tratamiento de agua) a bajo coste.
Este método MPEX es un método para pruebas genéticas que usa una reacción de elongación de ADN catalizada por una enzima (ADN polimerasa) en un sustrato y también se usa en análisis de mutación génica, análisis de SNP (Polimorfismo de un solo nucleótido) e identificación de microorganismos. Este método usa, como 35 cebadores, cadenas de oligonucleótidos inmovilizadas en un sustrato que tiene, en la superficie del mismo, una sustancia polimérica que contiene una primera unidad que tiene un grupo inducido a partir de éster fosfórico que constituye el resto hidrófilo de fosfolípido y una segunda unidad que tiene un grupo éster activo. Implica: tras la adición de una secuencia de ácido nucleico diana, desnaturalizar por calor el ADN bicatenario en cadenas sencillas en el sustrato y llevar a cabo la reacción de elongación de ADN catalizada por ADN polimerasa usando la cadena 40 sencilla como molde para incorporar bases, todas o algunas de las cuales están modificadas con un ligando, en el producto de reacción de elongación (ADN amplificado); y finalmente añadir al mismo una sustancia activa reactiva con el ligando para su reacción. Por tanto, el método MPEX consigue una detección génica altamente sensible.
El documento EP-A-1 634 963 se refiere a amplificación y secuenciación de ácidos nucleicos mediante amplificación en puente, y a aparatos y a equipos útiles para amplificación y secuenciación de alto rendimiento a 45 gran escala de ácidos nucleicos. El método comprende las etapas de: (1) formar al menos un molde de ácido nucleico que comprende el ácido nucleico que va a amplificarse, en el que dicho ácido nucleico contiene en el extremo 5' una secuencia de oligonucleótidos Y y en el extremo 3' una secuencia de oligonucleótidos Z y, además, porta en el extremo 5' un medio para unir el ácido nucleico a un soporte sólido; (2) mezclar dicho molde de ácido nucleico con un cebador de colonia X, que puede hibridar con la secuencia Z y porta en el extremo 5' un medio para 50 unir el cebador de colonia al soporte sólido, en presencia de un soporte sólido de manera que los extremos 5' tanto del molde de ácido nucleico como del cebador de colonia se unen al soporte sólido; (3) llevar a cabo una o más reacciones de amplificación de ácido nucleico en el molde unido, de manera que se generen colonias de ácido nucleico.
El documento WO 02/46456 A da a conocer otro método para amplificar ácidos nucleicos mediante 55 amplificación en puente que se realiza en condiciones isotérmicas.
El documento WO 01/62982 A describe un método de amplificación en puente de múltiples fases que emplea un producto de amplificación bicatenario formado durante una primera amplificación en puente para iniciar una fase posterior de amplificación en puente.
El documento WO 03/076902 A se refiere a un método de detección de moléculas de ácido nucleico diana en una muestra. El método implica proporcionar una pluralidad de grupos diferentes de dos o más conductores 5 eléctricos separados eléctricamente con sondas de captura, tales como oligonucleótidos, unidas a los conductores de manera que exista un espacio vacío entre las sondas de captura en los conductores separados eléctricamente. Se ponen en contacto las sondas de captura con una muestra en condiciones que permiten que cualquiera de las moléculas de ácido nucleico diana presentes en la muestra hibride con las sondas de captura y conecte de ese modo las sondas de captura en los conductores separados. Entonces, se detecta la presencia de la molécula de 10 ácido nucleico diana determinando si se conduce electricidad entre los conductores separados.
Collins M.L. et al., Nucleic Acid Research, 1997, vol. 25, n.º 1, páginas 2979-2984 describe un ensayo de amplificación de ADN ramificado para cuantificar moléculas diana de ácido nucleico. Se inmovilizan sondas de captura en un soporte sólido y se usan oligonucleótidos extensores de captura para capturar la molécula diana para el soporte sólido hibridando tanto con las sondas de captura como con la molécula diana. Posteriormente, se marca 15 la molécula diana uniendo oligonucleótidos específicos para la molécula diana denominados extensores de marcador. Para detectar la molécula diana, se unen moléculas amplificadoras de ADN ramificado, que a su vez unen muchas sondas de fosfatasa alcalina, a los extensores de marcador que se hibridan con la molécula diana.
SUMARIO DE LA INVENCIÓN
Sin embargo, los colorantes de tinción son inferiores en sensibilidad de amplificación de señal de detección 20 con respecto a las sustancias fluorescentes. Por tanto, el método MPEX no detecta de manera altamente sensible, por ejemplo, microorganismos patógenos tales como norovirus o Cryptosporidium, que están presentes sólo en un orden muy bajo en el agua (muestra) y no pueden hacerse proliferar mediante cultivo.
El método MPEX requiere, para una detección altamente sensible, aumentar la cantidad de marcadores y requiere, para este fin, la etapa de aumentar la concentración de una secuencia de ácido nucleico diana mediante 25 PCR o similares. Por tanto, este método lleva desventajosamente un tiempo prolongado para obtener resultados.
Por tanto, el método MPEX debe mejorarse adicionalmente para detectar microorganismos patógenos tales como norovirus o Cryptosporidium, que están presentes sólo en un orden muy bajo en el agua y no pueden hacerse proliferar mediante cultivo.
Alternativamente, incluso el uso de sustancias fluorescentes da como resultado una sensibilidad de 30 detección inestable para una cantidad extremadamente pequeña de un gen particular que va a detectarse, como en los microorganismos... [Seguir leyendo]
Reivindicaciones:
1. Método de detección de secuencias de ácido nucleico para detectar la presencia de una secuencia de ácido nucleico diana particular (10) que va a detectarse en una muestra, comprendiendo el método:
una etapa de inmovilización de anclar cadenas de oligonucleótidos (12, 14) sobre un sustrato (16) mediante la inmovilización de uno de los extremos 5' y 3' de las mismas sobre el sustrato; 5
una etapa de formación de espacio reticulado de llevar a cabo, basándose en un conjunto de las cadenas de oligonucleótidos (12, 14), la etapa de inmovilización y unir las cadenas de oligonucleótidos a sus respectivas partes complementarias de la secuencia de ácido nucleico diana (10) para formar una estructura entrecruzada en múltiples conjuntos para formar un espacio finamente reticulado (20) en el sustrato, en la que una pluralidad de las estructuras entrecruzadas se entrelazan entre sí; 10
una etapa de captura de capturar un ligando (22) por el espacio reticulado (20) a través de adsorción física;
una etapa de coloración de hacer que el ligando capturado (22) se coloree con una sustancia activa reactiva con el ligando; y
una etapa de detección de detectar una señal de color derivada de la coloración para detectar la secuencia de ácido nucleico diana (10). 15
2. Método de detección de secuencias de ácido nucleico para detectar la presencia de una secuencia de ácido nucleico diana particular (10) que va a detectarse en una muestra según la reivindicación 1, comprendiendo el método:
añadir, a un sistema de reacción, la muestra que contiene la secuencia de ácido nucleico diana (10) así como un ligando (22) como un constituyente de una sustancia cromógena y fijar la temperatura del sistema 20 de reacción a una temperatura a la que se desnaturaliza por calor la secuencia de ácido nucleico diana en cadenas sencillas antes de la etapa de formación de espacio reticulado; y
fijar la temperatura del sistema de reacción a una temperatura de hibridación a la que se unen de manera complementaria las cadenas de oligonucleótidos (12, 14) a la secuencia de ácido nucleico diana (10) en la etapa de formación de espacio reticulado y la etapa de captura; 25
añadir, sobre el sustrato (16), una sustancia activa como otro constituyente de la sustancia cromógena para su reacción con el ligando (22) en la etapa de coloración; y
detectar una señal de color derivada de la coloración mediante la sustancia cromógena en la etapa de detección.
3. Método de detección de secuencias de ácido nucleico para detectar la presencia de una secuencia de 30 ácido nucleico diana particular (19) que va a detectarse en una muestra, comprendiendo el método:
una etapa de diseño de diseñar de antemano cadenas de oligonucleótidos primera y segunda (24, 26) de manera que la segunda cadena de oligonucleótidos tenga una secuencia de ácido nucleico complementaria a un producto de reacción de elongación disociado de un dúplex formado a través de la reacción de elongación usando la primera cadena de oligonucleótidos como cebador y la secuencia de ácido nucleico 35 diana (19) como molde tras la unión complementaria de la secuencia de ácido nucleico diana y la primera cadena de oligonucleótidos;
una etapa de inmovilización de anclar las cadenas de oligonucleótidos primera y segunda (24, 26) sobre un sustrato (16) inmovilizando los extremos 5' de las mismas sobre el sustrato;
una primera etapa de reacción de elongación de unir de manera complementaria la primera cadena de 40 oligonucleótidos a la secuencia de ácido nucleico diana (19) y llevar a cabo la primera reacción de elongación usando la primera cadena de oligonucleótidos como cebador y la secuencia de ácido nucleico diana como molde;
una primera etapa de disociación de disociar, de la secuencia de ácido nucleico diana (19), el primer producto de reacción de elongación extendido en la primera etapa de reacción de elongación; 45
una primera etapa de entrecruzamiento de unir el primer producto de reacción de elongación disociado a la segunda cadena de oligonucleótidos para formar una estructura entrecruzada entre las cadenas de oligonucleótidos primera y segunda (24, 26);
una segunda etapa de reacción de elongación de llevar a cabo la segunda reacción de elongación usando la segunda cadena de oligonucleótidos como cebador y el primer producto de reacción de elongación como 50 molde;
una segunda etapa de disociación de disociar del mismo el segundo producto de reacción de elongación extendido en la segunda etapa de reacción de elongación;
una segunda etapa de entrecruzamiento de llevar a cabo la primera etapa de reacción de elongación con respecto a la segunda etapa de disociación en una pluralidad de las cadenas de oligonucleótidos primera y segunda (24, 26) y unir una primera cadena de oligonucleótidos diferente de la primera cadena de oligonucleótidos al segundo producto de reacción de elongación disociado para formar una estructura entrecruzada entre las cadenas de oligonucleótidos segunda y primera; 5
una etapa de entrecruzamiento repetitivo de repetir las etapas de entrecruzamiento primera y segunda entre una pluralidad de las cadenas de oligonucleótidos primera y segunda (24, 26);
una etapa de formación de espacio reticulado de formar un espacio finamente reticulado (20) mediante la etapa de entrecruzamiento repetitivo, en la que una pluralidad de las estructuras entrecruzadas se entrelazan entre sí; 10
una etapa de captura de capturar un ligando por el espacio reticulado (20) a través de adsorción física;
una etapa de coloración de hacer que el ligando capturado se coloree con una sustancia activa reactiva con el ligando (22); y
una etapa de detección de detectar una señal de color derivada de la coloración para detectar la secuencia de ácido nucleico diana (19). 15
4. Método de detección de secuencias de ácido nucleico según la reivindicación 3, que comprende además:
una tercera etapa de reacción de elongación de unir el segundo producto de reacción de elongación disociado en la segunda etapa de disociación a una tercera cadena de oligonucleótidos diseñada de manera que la tercera cadena de oligonucleótidos tenga una secuencia de ácido nucleico complementaria al segundo producto de reacción de elongación y llevar a cabo la tercera reacción de elongación usando la 20 tercera cadena de oligonucleótidos como cebador y el segundo producto de reacción de elongación como molde;
una tercera etapa de disociación de disociar, del segundo producto de reacción de elongación, el tercer producto de reacción de elongación extendido en la tercera etapa de reacción de elongación;
una tercera etapa de entrecruzamiento de unir una primera cadena de oligonucleótidos diferente de la 25 primera cadena de oligonucleótidos al tercer producto de reacción de elongación disociado para formar una estructura entrecruzada entre las cadenas de oligonucleótidos tercera y primera; y
una etapa de entrecruzamiento repetitivo de repetir las etapas de entrecruzamiento primera, segunda y tercera entre una pluralidad de las cadenas de oligonucleótidos primera, segunda y tercera.
5. Método de detección de secuencias de ácido nucleico según la reivindicación 3 ó 4, que comprende 30 además una etapa de hibridación de fijar la temperatura tras la etapa de entrecruzamiento repetitivo a una temperatura de hibridación para las cadenas de oligonucleótidos.
6. Método de detección de secuencias de ácido nucleico para detectar la presencia de una secuencia de ácido nucleico diana particular que va a detectarse en una muestra según la reivindicación 3, comprendiendo el método: 35
mezclar la muestra que contiene la secuencia de ácido nucleico diana con un ligando (22) como un constituyente de una sustancia cromógena para preparar una disolución de mezcla;
añadir la disolución de mezcla a un sistema de reacción en el sustrato (16) y fijar la temperatura del sistema de reacción a una temperatura de desnaturalización por calor a la que se desnaturaliza por calor la secuencia de ácido nucleico diana tras la etapa de inmovilización y antes de la etapa de formación de 40 espacio reticulado;
fijar la temperatura del sistema de reacción a una primera temperatura de reacción de elongación a la que se une de manera complementaria la primera cadena de oligonucleótidos a la secuencia de ácido nucleico diana (19) para la primera reacción de elongación en la primera etapa de disociación;
fijar la temperatura del sistema de reacción a una primera temperatura de disociación a la que se disocia el 45 primer producto de reacción de elongación del primer dúplex formado a través de la primera reacción de elongación;
fijar la temperatura del sistema de reacción a una segunda temperatura de reacción de elongación a la que el primer producto de reacción de elongación disociado se une de manera complementaria a la segunda cadena de oligonucleótidos para la segunda reacción de elongación en la segunda etapa de disociación; 50
fijar la temperatura del sistema de reacción a una segunda temperatura de disociación a la que el segundo producto de reacción de elongación se disocia del segundo dúplex formado a través de la segunda reacción de elongación;
fijar la temperatura del sistema de reacción a una temperatura a la que el segundo producto de reacción de elongación disociado se une de manera complementaria a la primera cadena de oligonucleótidos en la segunda etapa de entrecruzamiento;
añadir, sobre el sustrato (16), una sustancia activa como otro constituyente de la sustancia cromógena y fijar la temperatura a una temperatura para su reacción con el ligando (22); y 5
detectar una señal de color derivada de la coloración mediante la sustancia cromógena.
7. Método de detección de secuencias de ácido nucleico según una cualquiera de las reivindicaciones 1 a 6, en el que el ligando (22) es un elemento seleccionado del grupo que consiste en biotina, avidina, antígenos, hapteno y oligonucleótidos.
8. Método de detección de secuencias de ácido nucleico según una cualquiera de las reivindicaciones 1 a 7, 10 en el que la sustancia activa es un elemento seleccionado del grupo que consiste en receptores marcados con enzima, receptores marcados con sustancia fluorescente y sustratos.
9. Método de detección de secuencias de ácido nucleico según una cualquiera de las reivindicaciones 1 a 8, en el que el sustrato (16) tiene, en la superficie del mismo, una capa que comprende un polímero hidrófilo así como un grupo funcional reactivo con un grupo amino. 15
Patentes similares o relacionadas:
METODO IN VITRO PARA EL PRONOSTICO O PREDICCION DE LA RESPUESTA POR PARTE DE PACIENTES CON ARTRITIS REUMATOIDE AL TRATAMIENTO CON AGENTES QUE RECONOCEN EL RECEPTOR DE MEMBRANA CD20 DE LOS LINFOCITOS B, del 28 de Noviembre de 2011, de FUNDACIO INSTITUT DE RECERCA DE L'HOSPITAL UNIVERSITARI VALL D'HEBRON: Método in vitro para el pronóstico o predicción de la respuesta por parte de pacientes con artritis reumatoide al tratamiento con agentes que reconocen el receptor […]
TECNOLOGIA DE PROCESADORES GÉNICOS PARA DETERMINAR GENES DE MEMORIA, del 21 de Julio de 2011, de COLD SPRING HARBOR LABORATORY F. Hoffmann-La Roche AG EMORY UNIVERSITY: Un procedimiento que identifica un gen o genes candidatos implicados en la memoria dependiente de la transcripción de aprendizaje asociativo que comprende […]
 MÉTODO DE GUÍA DE ONDAS ÓPTICA PARA DETECTAR ACONTECIMIENTOS DE UNIÓN ESPECÍFICA MEDIANTE DIFUSIÓN DE LA LUZ, del 11 de Mayo de 2011, de ABBOTT LABORATORIES: Un procedimiento para determinar la secuencia de nucleótidos de segmento de ácido nucleico desconocido o para distinguir dos secuencias de nucleótidos estrechamente […]
MÉTODO DE GUÍA DE ONDAS ÓPTICA PARA DETECTAR ACONTECIMIENTOS DE UNIÓN ESPECÍFICA MEDIANTE DIFUSIÓN DE LA LUZ, del 11 de Mayo de 2011, de ABBOTT LABORATORIES: Un procedimiento para determinar la secuencia de nucleótidos de segmento de ácido nucleico desconocido o para distinguir dos secuencias de nucleótidos estrechamente […]
GENES DE CONTROL PARA LA NORMALIZACIÓN DE DATOS DE ANÁLISIS DE LA EXPRESIÓN GÉNICA, del 6 de Mayo de 2011, de SIRS-LAB GMBH: Conjunto de genes de control para la normalización de datos de análisis de la expresión génica de muestras de sangre de un paciente, en el que el conjunto […]
 APARATO Y PROCEDIMIENTO PARA LA SECUENCIACION DE ACIDOS NUCLEICOS, del 7 de Diciembre de 2010, de 454 LIFE SCIENCES CORPORATION: Matriz para secuenciar un ácido nucleico, que comprende: una oblea cavitada de fibras ópticas formadas a partir de un haz de una pluralidad de fibras ópticas […]
APARATO Y PROCEDIMIENTO PARA LA SECUENCIACION DE ACIDOS NUCLEICOS, del 7 de Diciembre de 2010, de 454 LIFE SCIENCES CORPORATION: Matriz para secuenciar un ácido nucleico, que comprende: una oblea cavitada de fibras ópticas formadas a partir de un haz de una pluralidad de fibras ópticas […]
METODOS Y PRODUCTOS PARA GENOTIPADO IN VITRO, del 30 de Septiembre de 2010, de PROGENIKA BIOPHARMA, S.A.: El método de genotipado se basa en la combinación de un diseño experimental de DNA-chips de genotipado y en el desarrollo de un sistema secuencial de procesamiento e interpretación […]
SISTEMA DE CLASIFICACION E INMOVILIZACION PARA ACIDOS NUCLEICOS UTILIZANDO SISTEMAS DE FIJACION SINTETICOS, del 14 de Septiembre de 2010, de NANOGEN RECOGNOMICS GMBH: Un método para modificar enzimáticamente un ácido nucleico diana, por reacción de ligación independiente del molde, o ligación de extremos romos o por PCR, […]
 METODOS Y COMPOSICIONES PARA LA DETECCION Y EL ANALISIS DE ACIDOS NUCLEICOS POR AMPLIFICACION DE SEÑAL, del 24 de Junio de 2010, de NATIONAL RESEARCH COUNCIL OF CANADA: Un sistema de detección de ácido nucleico que comprende:
una sonda oligonucleotídica aniónica marcada con un fluoróforo; y
un politiofeno catiónico crómico por afinidad […]
METODOS Y COMPOSICIONES PARA LA DETECCION Y EL ANALISIS DE ACIDOS NUCLEICOS POR AMPLIFICACION DE SEÑAL, del 24 de Junio de 2010, de NATIONAL RESEARCH COUNCIL OF CANADA: Un sistema de detección de ácido nucleico que comprende:
una sonda oligonucleotídica aniónica marcada con un fluoróforo; y
un politiofeno catiónico crómico por afinidad […]