UN MÉTODO PARA EL DESARROLLO MOLECULAR IN VITRO DE UNA FUNCIÓN PROTEICA.
Un método para generar una secuencia de polinucleótidos o población de secuencias desde las secuencias de polinucleótido de filamento simple codificando una o más proteinas causales,
el método comprende los pasos de: a) suministrar una primera población de moléculas de polinucleótidos de filamento simple, la primera y segunda poblaciones juntas constituyen los filamentos positivos y negativos de la secuencia de polinucleótidos progenitores; b) realizar una reacción para la asimilación de las primera y segunda poblaciones de moléculas de polinucleótidos de filamento simple con una exonucleasa para generar las correspondientes poblaciones de fragmentos de polinucleótidos de filamento simple; c) contactar dichos fragmentos de polinucleótidos generados desde los filamentos positivos con fragmentos generados desde los filamentos negativos; y d) amplificar los fragmentos que templan a cada otro para generar al menos una secuencia de polinucleótidos codificando una o más proteínas causales que tienen características alteradas como se comparó con una o más proteinas causales codificadas por dichos polinucleótidos progenitores. en donde, en el paso (b), al menos un parámetro de la reacción usada para asimilación de la primera población de moléculas de polinucleótidos de filamento simple es diferente del parámetro(s) equivalente(s) usado(s) en la reacción para la asimilación de la segunda población de moléculas de polinucleótidos de filamento simple.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2003/002102.
Solicitante: ALLIGATOR BIOSCIENCE AB.
Nacionalidad solicitante: Suecia.
Dirección: SCHEELEVAGEN 19A 223 70 LUND SUECIA.
Inventor/es: CARLSSON, ROLAND, FUREBRING, CHRISTINA, MALMBORG HAGER, ANN-CHRISTIN, BORREBAECK, CARL, ARNE, KRISTER.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- A61K38/00 NECESIDADES CORRIENTES DE LA VIDA. › A61 CIENCIAS MEDICAS O VETERINARIAS; HIGIENE. › A61K PREPARACIONES DE USO MEDICO, DENTAL O PARA EL ASEO (dispositivos o métodos especialmente concebidos para conferir a los productos farmacéuticos una forma física o de administración particular A61J 3/00; aspectos químicos o utilización de substancias químicas para, la desodorización del aire, la desinfección o la esterilización, vendas, apósitos, almohadillas absorbentes o de los artículos para su realización A61L; composiciones a base de jabón C11D). › Preparaciones medicinales que contienen péptidos (péptidos que contienen ciclos beta-lactama A61K 31/00; dipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina 2,5-dionas, A61K 31/00; péptidos basados en la ergolina A61K 31/48; que contienen compuestos macromoleculares que tienen unidades aminoácido repartidas estadísticamente A61K 31/74; preparaciones medicinales que contienen antígenos o anticuerpos A61K 39/00; preparaciones medicinales caracterizadas por los ingredientes no activos, p. ej. péptidos como soportes de fármacos, A61K 47/00).
- A61K48/00 A61K […] › Preparaciones medicinales que contienen material genético que se introduce en las células del cuerpo vivo para tratar enfermedades genéticas; Terapia génica.
- C07K14/00 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados.
- C12N15/09 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Tecnología del ADN recombinante.
- C12N15/10 C12N 15/00 […] › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12P21/02 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 21/00 Preparación de péptidos o de proteínas (proteína monocelular C12N 1/00). › que tienen una secuencia conocida de varios aminoácidos, p. ej. glutation.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
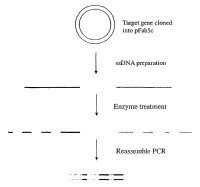
Fragmento de la descripción:
La presente invención describe un método para el desarrollo molecular in vitro de una función proteica, en particular barajando segmentos de flilamentos simples de ADN obtenidos por el uso de una nucleasa.
La función proteica puede ser modificada y perfeccionada in vitro por una variedad de métodos, que incluyen la clonación combinatoria de mutagénesis controlada en un sitio (Alber et al., Nature, 5; 330 (6143) :41-46, 1987) (Huse et al., Science, 246:1275-1281, 1989; Marks et al., Biotechnology, 10: 779-783, 1992) y mutagénesis aleatoria combinada con sistemas apropiados de selección (Barbas et al., PNAS. USA, 89: 4457-4461, 1992) .
El método de mutagénesis aleatoria junto con la selección ha sido usado en un número de casos para perfeccionar la función proteica y existen dos estrategias diferentes. Primeramente, la aleatorización de la secuencia genética completa en combinación con la selección de una variante proteica (mutante) con las características deseadas, seguida por un nuevo ciclo de mutagénesis aleatoria y selección. Este método puede entonces ser repetido hasta que una variante proteica es encontrada la cual es considerada óptima (Schier R. et al.,
J. Mol. Biol. 1996 263 (4) : 551-567) . Aquí, la ruta tradicional para introducir mutaciones es por medio de la tendencia al error PCR (Leung et al., Technique, 1: 11-15, 1989) con un índice de mutación de aproximadamente 0.7 %. En segundo lugar, las regiones definidas del gen puede ser mutagenizada con degradaciones de bases, que permiten índices de mutación de hasta el 100 % (Griffiths et al., EMBO. J, 13: 3245-3260, 1994; Yang et al., J. Mol.
Biol. 254: 392-403, 1995) . A más alto índice de mutación usado, más limitada la región del gen que puede ser sometida a mutaciones.
La mutación aleatoria ha sido usada extensivamente en el campo de la ingeniería de anticuerpos. Los genes anticuerpos formados in vivo pueden ser clonados in v itro (Larrick et al., Biochem. Biophys. Res. Commun. 160: 1250-1256, 1989) y las combinaciones aleatorias de los genes que codifican la variable de genes pesados y ligeros pueden ser sometidas a la selección (Marks et al ., Biotechnology, 10: 779-783, 1992) . Los fragmentos de anticuerpos funcionales seleccionados pueden ser además perfeccionados por el uso de mutagénesis aleatoria y ciclos adicionales de selección (Schier R. et al., J. Mol. Biol. 1996 263 (4) : 551-567) .
La estrategia de la mutagénesis aleatoria es seguida por la selección. Las variantes con características interesantes pueden ser seleccionadas y las regiones de ADN mutadas de diferentes variantes, cada cual con características interesantes, son combinadas dentro de una secuencia de codificación (Yang et al., J. Mol. Biol. 254: 392-403, 1995) . Este es un proceso secuencial de múltiples pasos, y los efectos sinergéticos potenciales de las diferentes mutaciones pueden perderse, ya que ellos no están sometidos a la selección en combinación. Así, estas dos estrategias no incluyen la mutagénesis simultánea de regiones definidas y la selección de una combinación de estas regiones.
Otro proceso implica el pareo combinatorio de genes los cuales pueden ser usados para perfeccionar, por ejemplo. la afinidad de anticuerpo (Marks et al., Biotechnology, 10: 779-783, 1992) . Aquí, las tres regiones CDR en cada gen variable están fijas y esta tecnología no permite barajar los segmentos del gen individual en el gen por el dominio variable, por ejemplo, incluyendo las regiones CDR, entre clones.
El concepto de barajar el ADN (Stemmer, Nature 370: 389-391, 1994) utiliza la fragmentación aleatoria del ADN y el ensamblaje de los fragmentos en una secuencia de codificación funcional. En este proceso, es posible introducir secuencias de ADN sintetizadas químicamente y de esta manera la variación objetivo para definir los lugares en el gen cuya secuencia ADN es conocida (Crameri et al., Biotechniques, 18: 194-196, 1995) . Stemmer y colaboradores desarrollaron este método in vitro que se asemeja al proceso de evolución normal de la proteína en la naturaleza. El barajar el ADN genera diversidad por recombinación, combinando mutaciones útiles de genes individuales. Ha sido utilizado exitosamente para la evolución artificial de diferentes proteínas, por ejemplo, enzimas y citoquinas (Chang et al. Nature Biotech 17, 793-797, 1999; Zhang et al. Proc. Natl. Acad. Sci. USA 94, 4504-4509, 1997; Christians et al. Nature Biotech. 17, 259-264, 1999) . Los genes son fragmentados aleatoriamente usando ADNasa I y entonces reensamblados por recombinación con cada otro. El material de partida puede ser o un gen simple (mutado primero aleatoriamente usando la tendencia al error (PCR) o secuencias homólogas que surgen naturalmente (también llamadas barajeo de familia) . La ADNasa I hidroliza el ADN preferentemente en los sitios adyacentes a los nucleótidos pirimidina, por lo tanto es una selección adecuada para la fragmentación aleatoria del ADN. Sin embargo, la actividad es dependiente de los iones Mg o Mn, los iones Mg restringen el tamaño del fragmento a 50 bp, mientras que los iones Mn darán tamaños de fragmentos menores que 50 bp. Por lo tanto, para obtener todos los tamaños posibles para la recombinación el gen en cuestión necesita ser tratado al menos dos veces con ADNasa I en presencia de cualquiera de los dos iones diferentes, seguida por la remoción de esos mismos iones.
Un método adicional para generar una colección de ADN recombinante, usando fragmentos de ADN de cadena sencilla unidireccional, se describe en WO 02/38757.
En teoría, es posible barajar ADN entre cualquiera de los clones. Sin embargo, si el gen barajado resultante debe ser funcional con respecto a expresión y actividad, los clones a ser barajados tienen que ser preferiblemente relacionados o hasta idénticos, con la excepción de un bajo nivel de mutaciones aleatorias. Barajar el ADN entre clones genéticamente diferentes producirá generalmente genes no funcionales. Sin embargo, ha sido comprobado por la metodología ITCHY que las colecciones de fusión inter-especie pueden ser creadas entre fragmentos de la E. coli y los genes humanos transformilasa ribonucleótido glicinamida, los cuales tienen solamente el 50 % de identidad en el nivel de ADN (Ostermeier et al., Nat Biotechnol 17, 1205-9, 1999) .
Una recombinación exitosa de dos genes diferentes requiere la formación de moléculas heterodobles. En algunos casos de familias barajadas casi solamente forman homo-dúplex como resultado de una recombinación de baja frecuencia. Este problema puede ser dirigido por el uso de ADN de cadena sencilla asimilado por ADNasa I (Kikuchi et al. Gene 243, 133-137 2000) .
El ADN de filamento simple puede ser obtenido usando métodos conocidos en la técnica. Por ejemplo, las bases biotinilatadas pueden ser usadas en las reacciones PCR en combinación con, por ejemplo, Dynabeads (Dynal, Norway) or AffiniTip Streptavidin Capture Micro-columns (Genosys Biotechnologies Inc., The Woodlands, USA) . Alternativamente, el ADN de filamento simple puede ser obtenido por la utilización de bacteriófagos que son capaces de empacar el ADN de cadena sencilla (Viruses and Related Entities in Modem Microbiology, Principles and Applications pp.171-192, Ed. E.A. Birge, Wm. C. Brown Publishers1992; Sambrook et al. Molecular Cloning, A laborator y manual 2nd edition. Cold Spring Habor Laborator y Press, 1989) . En adición, pueden ser usados los métodos asimétricos de PCR (ver Ejemplo 1) .
La selección de enzimas con propiedades alteradas y perfeccionadas está a menudo basada en la función real de la enzima. Por ejemplo, el incremento de la termo estabilidad de una enzima puede ser seleccionada por medio de la incubación de colonias transformadas a temperaturas que causan inactivación de tipos de enzimas descontroladas. En adición, la actividad perfeccionada de -glucosidasa puede ser identificada por el uso de PNPG como el sustrato (Arrizubieta et al J Biol Chem Jun 27, 2000) .
La selección de proteínas funcionales desde las colecciones moleculares ha sido revolucionada por el desarrollo de la tecnología de exposición de bacteriofagia (Parmley et al., Gene, 73: 305-391 1988; McCafferty et al., Nature, 348: 552-554, 1990; Barbas et al., PNAS. USA, 88: 7978-7982, 1991) . Aquí, el fenotipo (proteína) está directamente vinculado a su correspondiente genotipo (DNA) y esto permite controlar la clonación del material genético, el cual puede entonces ser sometido a modificaciones adicionales para perfeccionar la...
Reivindicaciones:
1. Un método para generar una secuencia o población de secuencias de polinucleótidos a partir de secuencias progenitoras de polinucleótidos de cadena sencilla codificando uno o más motivos proteicos, el método comprendiendo los pasos de:
a) suministrar una primera población de moléculas de polinucleótidos de cadena sencilla y una segunda población de moléculas de polinucleótidos de cadena sencilla, la primera y segunda poblaciones juntas constituyendo cadenas positivas y negativas de la secuencia de polinucleótidos progenitores;
b) realizar una reacción para la asimilación de las primera y segunda poblaciones de moléculas de polinucleótidos de cadena sencilla con una exonucleasa para generar las correspondientes poblaciones de fragmentos de polinucleótidos de cadena sencilla;
c) contactar dichos fragmentos de polinucleótidos generados desde las cadenas positivas con fragmentos generados desde las cadenas negativas; y
d) amplificar los fragmentos que se hibridan entre sí para generar al menos una secuencia de polinucleótidos codificando uno o más motivos proteicos que tienen características alteradas comparados con uno o más motivos proteicos codificados por dichos polinucleótidos progenitores.
en donde, en el paso (b) , al menos un parámetro de la reacción usada para asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es diferente del parámetro (s) equivalente (s) usado (s) en la reacción para la asimilación de la segunda población de moléculas de polinucleótidos de cadena sencilla.
2. Un método de acuerdo a la reinvindicación 1, en el que el parámetro de la reacción es seleccionado de un tipo de exonucleasa, una concentración de exonucleasa, un volumen de reacción, duración de la reacción de asimilación, la temperatura de la mezcla de reacción, el pH de la mezcla de reacción, la longitud de las secuencias de polinucleótidos de cadena sencilla progenitoras, la cantidad de moléculas de polinucleótidos de cadena sencilla y la composición tampón de la mezcla de reacción.
3 Un método de acuerdo a la reivindicación 1 o 2, en el que la exonucleasa usada para la asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es diferente de la exonucleasa usada para la asimilación de la segunda población de moléculas de polinucleótidos de cadena sencilla.
4 Un método de acuerdo a la reinvindicación 3, en el que la exonucleasa usada para la asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es una exonucleasa 3’ y la exonucleasa usada para la asimilación de la segunda población de moléculas de polinucléótidos de cadena sencilla es una exonucleasa 5’.
Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la concentración de exonucleasa usada para la asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es diferente de la concentración de exonucleasa usada para la asimilación de la segunda población de moléculas de polinucleótidos de cadena sencilla.
6 Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que el volumen de reacción usado para la asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es diferente del volumen de reacción usado para la segunda población de moléculas de polinucleótidos de cadena sencilla.
7 Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la duración de la reacción de asimilación usada para la asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es diferente de la duración de la reacción de asimilación usada para la asimilación de la segunda población de moléculas de polinucleótidos de cadena sencilla.
8 Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la temperatura de la mezcla de la reacción usada para la asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es diferente de la temperatura de la mezcla de la reacción usada para la asimilación de la segunda población de moléculas de polinucleótidos de cadena sencilla.
9. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que el pH de la mezcla de la reacción usada para la asimilación de la primera población de moléculas de polinucleótidos de cadena sencilla es diferente del pH de la mezcla de la reacción usada para la asimilación de la segunda población de moléculas de polinucleótidos de cadena sencilla.
10. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la longitud de los polinucleótidos en la primera población de moléculas de polinucleótidos de cadena sencilla es diferente de la longitud de los polinucleótidos en la segunda población de moléculas de polinucleótidos de cadena sencilla.
11. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la composición tampón de la mezcla de reacción usada para la asimilación de la primera población de moléculas de polinucleótidos
de cadena sencilla es diferente de la composición tampón de la mezcla de reacción usada para la asimilación de la segunda población de moléculas de polinucleótidos de cadena sencilla.
12. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la cantidad de moléculas de polinucleótidos de cadena sencilla en la primera población de moléculas de polinucleótidos de cadena sencilla es diferente de la cantidad de moléculas de polinucleótidos de cadena sencilla en la segunda población de moléculas de polinucleótidos de cadena sencilla.
13. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la primera población de moléculas de polinucleótidos de cadena sencilla constituye las cadenas positivas de las secuencias de polinucleótidos progenitoras y la segunda población de moléculas de polinucleótidos de cadena sencilla constituye las cadenas negativas de las secuencias de polinucleótidos progenitoras.
14. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que las moléculas de polinucleótido del paso (a) son moléculas de ADN.
15. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que el paso c) además comprende añadir secuencias iniciadoras que hibridan a los terminales 3’ y/o 5’ de al menos uno de los polinucleótidos progenitores bajo condiciones de hibridación.
16. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que la exonucleasa usada para asimilar la primera y/o la segunda población de moléculas de polinucleótidos de cadena sencilla es seleccionada del grupo que consiste de BAL 31, exonucleasa I, exonucleasa V, exonucleasa VII, exonucleasa T7gene 6, exonucleasa lambda bacterófaga y exonucleasa Rec Jf.
17. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que una secuencia
o secuencias de polinucleótidos progenitores ha (n) sido sometida (s) a mutagénesis.
18. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que una o ambas poblaciones de fragmentos generados en el paso b) es (son) sometida (s) a mutagénesis.
19. Un método de acuerdo a la reivindicación 17 o 18 donde la mutagénesis es PCR propensa a error.
20. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes, en el que el paso b) es realizado para generar poblaciones de fragmentos de cadena sencilla de diversas longitudes.
21. Un método de acuerdo a la reinvindicación 20 donde el paso b) es controlado para generar fragmentos de cadena sencilla que tienen una longitud promedio de más que aproximadamente 50 nucleótidos.
22. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes que además comprende el paso de expresar in vitro al menos una secuencia de polinucleóticos generada en el paso d) para producir el polipéptido codificado.
23. Un método de acuerdo a la reinvindicación 22 que además comprende el paso de prueba del polipéptido codificado para las carácterísticas deseadas.
24. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes en el que la secuencia de polinucleótidos progenitora codifica un anticuerpo o fragmento del mismo.
25. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes en el que la secuencia de polinucleótidos progenitora codifica una enzima.
26. Un método de acuerdo a una cualquiera de las reinvindicaciones precedentes en el que la secuencia de polinucleótidos progenitora codifica un antígeno.
27. Un método para hacer un polipéptido que tiene propiedades deseadas, el método comprendiendo los pasos siguientes:
(a) generar formas variantes de un polinucleótido progenitor usando un método de acuerdo a una cualquiera de las reivindicaciones 1 a 26;
(b) expresar los polinucleótidos variantes producidos en el paso (a) para producir polipéptidos variantes;
(c) filtrar los polopéptidos variantes para las propiedades deseadas; y
(d) seleccionar un polopéptido que tiene las propiedades deseadas de los polipéptidos variantes.
28. Un método de acuerdo a una cualquiera de las reivindicaciones 1 a 26 el cual comprende, después de la identificación de un polinucleótido y/o un polipéptido codificado con las carácterísticas deseadas, añadir dicho polinucleótido y/o polipéptido codificado a un portador aceptable farmacéuticamente.
Patentes similares o relacionadas:
Composiciones útiles en el tratamiento de la deficiencia de ornitina transcarbamilasa (OTC), del 8 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Un vector vírico recombinante que comprende una secuencia de ácido nucleico que codifica la proteína ornitina transcarbamilasa humana (hOTC) y secuencias […]
Terapia génica para la diabetes, del 8 de Julio de 2020, de UCL Business Ltd: Una molécula de ácido nucleico que comprende una secuencia de nucleótidos que codifica una proteína preproinsulina funcional en donde la secuencia de nucleótidos tiene al menos […]
Vacuna de ADN que contiene un epítopo específico de VEGF y/o un epítopo específico de angiopoyetina-2, del 1 de Julio de 2020, de OSAKA UNIVERSITY: Un vector de expresión que codifica un polipéptido del antígeno del núcleo del virus de la hepatitis B quimérico con una inserción para uso en el tratamiento o la profilaxis […]
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Ácido nucleico antisentido, del 24 de Junio de 2020, de NIPPON SHINYAKU CO., LTD.: Un oligómero antisentido de 14 a 32 bases de longitud, que comprende dos unidades de oligómeros conectadas seleccionadas del grupo que consiste […]
Plekhg5 como diana farmacéutica para trastornos neurológicos, del 15 de Junio de 2020, de CONSEJO SUPERIOR DE INVESTIGACIONES CIENTIFICAS (CSIC): Plekhg5 como diana farmacéutica para trastornos neurológicos. La invención hace referencia al uso del gen Plekhg5 como diana farmacológica para el cribado, […]
Vectores de AAV dirigidos a oligodendrocitos, del 10 de Junio de 2020, de THE UNIVERSITY OF NORTH CAROLINA AT CHAPEL HILL: Un ácido nucleico que codifica una cápside de AAV, comprendiendo el ácido nucleico una secuencia codificante de la cápside de AAV que es al menos el 96 % idéntica […]
Método para activar células T auxiliares, del 10 de Junio de 2020, de OTSUKA PHARMACEUTICAL CO., LTD.: Una composición para su uso en el tratamiento o prevención del cáncer mediante la activación de células T auxiliares en un sujeto, en donde dicha composición […]