OLIGONUCLEÓTIDOS PARA DIAGNÓSTICOS DE CÁNCER DE MAMA.
Un método para preparar un patrón estándar de transcritos génicos característico del cáncer de mama en un organismo,
que comprende al menos las operaciones de: a) aislar mRNA de las células de una muestra de uno o más organismos que tienen cáncer de mama; b) hibridar el mRNA de la operación (a) con un conjunto de sondas oligonucleotídicas específicas para dicho cáncer de mama en un organismo y una muestra del mismo que corresponda al organismo y una muestra del mismo bajo investigación, en que dicho conjunto consiste en menos de 500 oligonucleótidos y comprende todos los oligonucleótidos enumerados en la Tabla 2 ó 3, en que cada oligonucleótido puede ser sustituido por un oligonucleótido que es una parte de dicho oligonucleótido de la Tabla 2 ó 3 o por un oligonucleótido con una secuencia complementaria; y c) evaluar la cantidad de mRNA que se hibrida con cada una de dichas sondas para producir un patrón característico que refleje el nivel de expresión génica de los genes a los que se unen dichos oligonucleótidos, en la muestra con cáncer de mama
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2005/002180.
Solicitante: DIAGENIC AS.
Nacionalidad solicitante: Noruega.
Dirección: OSTENSJOVEIEN 158 0661 OSLO NORUEGA.
Inventor/es: SHARMA, PRAVEEN, LONNEBORG,ANDERS.
Fecha de Publicación: .
Fecha Solicitud PCT: 2 de Junio de 2005.
Clasificación Internacional de Patentes:
- C12Q1/68M6B
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
PDF original: ES-2358910_T3.pdf



Fragmento de la descripción:
El presente invento se refiere a sondas oligonucleotídicas para uso en la evaluación de los niveles de transcritos génicos en una célula, que pueden ser utilizadas en técnicas analíticas, particularmente en técnicas diagnósticas. Convenientemente, las sondas se proporcionan en forma de kit. Se pueden utilizar diferentes conjuntos de sondas en técnicas para preparar patrones de expresión génica e identificar, diagnosticar o controlar cánceres de mama.
La identificación de métodos rápidos y sencillos para el análisis de muestras para, por ejemplo, aplicaciones diagnósticas sigue siendo el objetivo de muchos investigadores. Los usuarios finales buscan métodos que sean eficaces en cuanto al costo, produzcan resultados estadísticamente significativos y puedan ser rutinariamente implementados sin necesidad de individuos muy especializados.
El análisis de la expresión génica dentro de las células ha sido utilizado para obtener información sobre el estado de esas células e, importantemente, el estado del individuo del que proceden las células. La expresión relativa de diversos genes en una célula ha sido identificada como un reflejo de un estado particular de un organismo. Por ejemplo, se sabe que las células cancerosas presentan una expresión alterada de diversas proteínas y, por lo tanto, se pueden usar los transcritos o las proteínas expresadas como marcadores de ese estado morboso.
De esta manera, se puede analizar el tejido de una biopsia en cuanto a la presencia de esos marcadores y se pueden identificar células procedentes del sitio de la enfermedad en otros tejidos o fluidos del organismo por la presencia de los marcadores. Además, se pueden liberar productos de la expresión alterada en la corriente sanguínea y se pueden analizar esos productos. Además, las células que han entrado en contacto con las células de la enfermedad pueden verse afectadas por su contacto directo con esas células para dar lugar a una expresión génica alterada, y su expresión o los productos de su expresión pueden ser similarmente analizados.
Sin embargo, hay ciertas limitaciones con estos métodos. Por ejemplo, el uso de marcadores tumorales específicos para identificar el cáncer adolece de una diversidad de defectos, tales como falta de especificidad o sensibilidad, asociación del marcador con estados morbosos además de con el tipo específico de cáncer, y dificultad para la detección en individuos asintomáticos.
Más recientemente, además del análisis de una o dos proteínas o transcritos marcadores, se han analizado patrones de expresión génica. La mayoría del trabajo, que acarrea el análisis de la expresión génica a gran escala con implicaciones en el diagnóstico de la enfermedad, ha requerido muestras clínicas procedentes de células o tejidos enfermos. Por ejemplo, en varias publicaciones recientes, que demuestran que se pueden usar datos de expresión génica para distinguir entre tipos de cáncer similares, se han utilizado muestras clínicas procedentes de células o tejidos enfermos (Alon et al., 1999, PNAS 96, páginas 6745-6750; Golub et al., 1999, Science 286, páginas 531-537; Alizadeh et al., 2000, Nature 403, páginas 503-511; Bittner et al., 2000, Nature 406, páginas 536-540).
Sin embargo, estos métodos se han basado en el análisis de una muestra que contiene células enfermas o productos de esas células o células que han entrado en contacto con células de la enfermedad. El análisis de dichas muestras depende del conocimiento de la presencia de una enfermedad y su localización, lo que puede ser difícil en pacientes asintomáticos. Además, no siempre se pueden tomar muestras del sitio de la enfermedad; por ejemplo, en enfermedades del cerebro.
En un hallazgo de gran significación, los presentes inventores identificaron el potencial previamente no explotado de todas las células de un organismo para obtener información relativa al estado del organismo del cual procedían las células. En el Documento WO98/49342 se describe el análisis de la expresión génica de células alejadas del sitio de la enfermedad, por ejemplo, de sangre periférica recogida lejos de un sitio canceroso. En el Documento WO 2004/046382 se describen sondas específicas para el diagnóstico del cáncer de mama y la enfermedad de Alzheimer y se discuten protocolos para identificar otras sondas apropiadas para ese fin y para diagnosticar otras enfermedades.
Este hallazgo se basa en la premisa de que las diferentes partes del cuerpo de un organismo existen en interacción dinámica entre sí. Cuando una enfermedad afecta a una parte del cuerpo, otras partes del cuerpo resultan también afectadas. La interacción es el resultado de un amplio espectro de señales bioquímicas que se liberan de la zona enferma, para afectar a otras zonas del cuerpo. Aunque la naturaleza de los cambios bioquímicos y fisiológicos inducidos por las señales liberadas puede variar en las diferentes partes del cuerpo, los cambios pueden ser medidos a nivel de la expresión génica y ser utilizados con fines diagnósticos.
El estado fisiológico de una célula en un organismo viene determinado por el patrón con que se expresan genes en ella. El patrón depende de los estímulos biológicos internos y externos a los que está expuesta dicha célula, y cualquier cambio en el grado o la naturaleza de estos estímulos puede conducir a un cambio en el patrón con que se expresan los diferentes genes en la célula. Existe el conocimiento creciente de que, analizando los cambios sistémicos en patrones de expresión génica en células de muestras biológicas, es posible obtener información sobre el tipo y la naturaleza de los estímulos biológicos que están actuando sobre ellas. De este modo, por ejemplo, controlando la expresión de un gran número de genes en células de una muestra de ensayo, es posible determinar si sus genes se expresan con un patrón característico de una enfermedad, un estado o una fase particular de los mismos. Por lo tanto la medición de cambios en las actividades génicas de células, por ejemplo, de tejidos o fluidos corporales, está surgiendo como una potente herramienta para el diagnóstico de enfermedades.
**(Ver fórmula)**
Dichos métodos presentan varias ventajas. A menudo, la obtención de muestras clínicas de ciertas zonas del cuerpo que está enfermo puede ser difícil y puede implicar invasiones indeseables del cuerpo; por ejemplo, a menudo se utiliza la biopsia para obtener muestras de cáncer. En ciertos casos, tal como en la enfermedad de Alzheimer, la muestra del cerebro enfermo sólo se puede obtener post mórtem. Además, las muestras tisulares que se obtienen son a menudo heterogéneas y pueden contener una mezcla de células tanto enfermas como no enfermas, lo que hace que el análisis de los datos sobre expresión génica generados sea complejo y difícil.
Se ha sugerido que una colección de tejidos tumorales que parecen ser patogenéticamente homogéneos con respecto a aspectos morfológicos del tumor bien puede ser muy heterogénea a nivel molecular (Alizadeh, 2000, supra) y, en realidad, podría contener tumores que representaran enfermedades esencialmente diferentes (Alizadeh, 2000, supra; Golub, 1999, supra). Con el fin de identificar una enfermedad, un estado o una fase de los mismos, es muy deseable cualquier método que no requiera que las muestras clínicas procedan directamente de células o tejidos enfermos ya que, de una región fácilmente accesible del organismo, se pueden obtener muestras clínicas que representan una mezcla homogénea de tipos celulares.
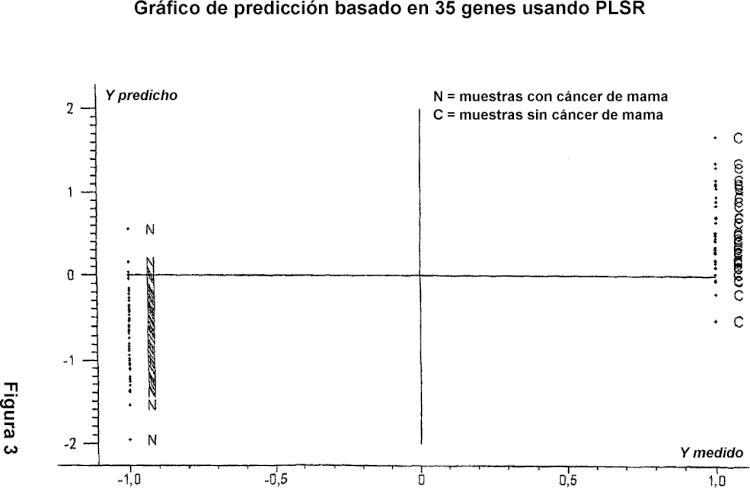
Hemos identificado ahora una familia de secuencias que permite la obtención de un conjunto de sondas de sorprendente utilidad para identificar el cáncer de mama. De este modo, describimos ahora familias de genes cuya expresión está alterada en las células de muestras sanguíneas procedentes de pacientes con cáncer de mama, que se pueden usar para generar sondas para uso en métodos para identificar, diagnosticar o controlar el cáncer de mama o fases del mismo.
En el trabajo que condujo a este invento, los inventores examinaron el nivel de expresión de un gran número de genes en pacientes con cáncer con respecto al nivel en pacientes normales. No sólo se halló que había un gran número de genes que presentaban una expresión alterada sino que, además, se halló que aquellos que presentaban una expresión alterada caían dentro de familias discretas de genes en virtud de su función. Como tales, estos genes proporcionan una colección a partir de la cual se pueden generar las correspondientes sondas que pueden ser utilizadas... [Seguir leyendo]
Reivindicaciones:
1. Un método para preparar un patrón estándar de transcritos génicos característico del cáncer de mama en un organismo, que comprende al menos las operaciones de:
a) aislar mRNA de las células de una muestra de uno o más organismos que tienen cáncer de mama;
b) hibridar el mRNA de la operación (a) con un conjunto de sondas oligonucleotídicas específicas para dicho cáncer de mama en un organismo y una muestra del mismo que corresponda al organismo y una muestra del mismo bajo investigación, en que dicho conjunto consiste en menos de 500 oligonucleótidos y comprende todos los oligonucleótidos enumerados en la Tabla 2 ó 3, en que cada oligonucleótido puede ser sustituido por un oligonucleótido que es una parte de dicho oligonucleótido de la Tabla 2 ó 3 o por un oligonucleótido con una secuencia complementaria; y
c) evaluar la cantidad de mRNA que se hibrida con cada una de dichas sondas para producir un patrón característico que refleje el nivel de expresión génica de los genes a los que se unen dichos oligonucleótidos, en la muestra con cáncer de mama.
2. Un método para preparar un patrón de transcritos génicos de ensayo, que comprende al menos las operaciones de:
a) aislar mRNA de las células de una muestra de dicho organismo de ensayo;
b) hibridar el mRNA de la operación (a) con un conjunto de oligonucleótidos como los definidos en la Reivindicación 1, específicos para el cáncer de mama en un organismo y una muestra del mismo que corresponda al organismo y una muestra del mismo bajo investigación; y
c) evaluar la cantidad de mRNA que se hibrida con cada una de dichas sondas para producir dicho patrón que refleja el nivel de expresión génica de los genes a los que se unen dichos oligonucleótidos, en dicha muestra de ensayo.
3. Un método para diagnosticar o identificar o controlar el cáncer de mama en un organismo, que comprende las operaciones de:
a) aislar mRNA de las células de una muestra de dicho organismo;
b) hibridar el mRNA de la operación (a) con un conjunto de oligonucleótidos como los definidos en la Reivindicación 1, específicos para cáncer de mama en un organismo y una muestra del mismo que corresponde al organismo y una muestra del mismo bajo investigación;
c) evaluar la cantidad de mRNA que se hibrida con cada una de dichas sondas para producir un patrón característico que refleje el nivel de expresión génica de los genes a los que se unen dichos oligonucleótidos, en dicha muestra; y
d) comparar dicho patrón con un patrón diagnóstico estándar preparado de acuerdo con el método de la Reivindicación 1, utilizando una muestra de un organismo que corresponde al organismo y una muestra bajo investigación para determinar la presencia de cáncer de mama en el organismo bajo investigación.
4. Un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 3, en que dicho mRNA aislado es inversa-mente transcrito a cDNA y dicho cDNA sustituye a dicho mRNA en las operaciones b) y c) de dicho método.
5. Un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 4, en que dicho conjunto comprende además todos los oligonucleótidos enumerados en la Tabla 4, en que cada oligonucleótido puede ser sustituido por un oligonucleótido que es una parte de dicho oligonucleótido de la Tabla 4 o por un oligonucleótido con una secuencia complementaria.
6. Un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 5, en que dicho conjunto de sondas está inmovilizado sobre uno o más soportes sólidos.
7. Un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 6, en que dichas células no son células de la enfermedad, no han entrado en contacto con células de la enfermedad y no proceden del sitio de la enfermedad.
8. Un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 7, en que dicha muestra ha sido obtenida de un sitio alejado del sitio de la enfermedad.
9. Un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 8, en que dicha muestra es tejido, fluido corporal o desecho corporal.
10. Un método como el reivindicado en la Reivindicación 9, en que dicha muestra es sangre periférica.
11. Un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 10, en que dicho organismo es un mamífero.
12. Un método como el reivindicado en la Reivindicación 11, en que dicho mamífero es un ser humano.
13. Un conjunto de menos de 500 sondas oligonucleotídicas como el definido en cualquiera de las Reivindicaciones 1 a
**(Ver fórmula)**
12.
14. Un kit para llevar a cabo un método como el reivindicado en cualquiera de las Reivindicaciones 1 a 12, que comprende un conjunto de sondas oligonucleotídicas como el definido en la Reivindicación 13, inmovilizado sobre uno o más soportes sólidos.
15. Un kit como el reivindicado en la Reivindicación 14, que comprende además una información añadida que detalla cómo se debería llevar el método a cabo.
16. El uso de un conjunto de sondas oligonucleotídicas o un kit como los definidos en cualquiera de las Reivindicaciones
10 13 a 15 para determinar el patrón de expresión génica de una célula, patrón que refleja el nivel de expresión génica de genes a los que se unen dichas sondas oligonucleotídicas para el diagnóstico del cáncer de mama, que comprende al menos las operaciones de
a) aislar mRNA de dicha célula;
b) hibridar el mRNA de la operación (a) con un conjunto de sondas oligonucleotídicas o un kit como los defini15 dos en cualquiera de las 1. a 15; y
c) evaluar la cantidad de mRNA que se hibrida con cada una de dichas sondas para producir dicho patrón.
17. Un uso como el reivindicado en la Reivindicación 16, en que dicho mRNA aislado es inversamente transcrito a cDNA y dicho cDNA sustituye a dicho mRNA en las operaciones b) y c) de dicho método.
Patentes similares o relacionadas:
MARCADORES PARA LA SELECCIÓN DE TERAPIAS PERSONALIZADAS PARA EL TRATAMIENTO DEL CÁNCER, del 7 de Febrero de 2012, de FUNDACION INSTITUT DE RECERCA DE L'HOSPITAL UNIVERSITARI VALL D'HEBRON: La invención se relaciona con la identificación de los niveles de expresión de aprataxina (APTX) como marcador de respuesta a terapias basadas en inhibidores de topoisomerasa […]
MÉTODOS PARA SELECCIONAR REGÍMENES DE TRATAMIENTO Y PREDECIR RESULTADOS EN PACIENTES CON CÁNCER DE MAMA, del 6 de Febrero de 2012, de AMERICAN DIAGNOSTICA INC: Un método para seleccionar de dos o más regímenes de tratamiento, un régimen de tratamiento que tiene el mayor beneficio esperado para un […]
 METODO Y KIT PARA EL PRONOSTICO DEL LINFOMA DE CELULAS DEL MANTO, del 23 de Enero de 2012, de HOSPITAL CLINIC DE BARCELONA UNIVERSIDAD DE BARCELONA FUNDACIO CLINIC PER A LA RECERCA BIOMEDICA INSTITUT D'INVESTIGATIONS BIOMEDIQUES AUGUST PI I SUNYER (IDIBAPS): Método y kit para el pronóstico del linfoma de células del manto.
El método y el kit son útiles como herramientas para clasificar un paciente diagnosticado con linfoma […]
METODO Y KIT PARA EL PRONOSTICO DEL LINFOMA DE CELULAS DEL MANTO, del 23 de Enero de 2012, de HOSPITAL CLINIC DE BARCELONA UNIVERSIDAD DE BARCELONA FUNDACIO CLINIC PER A LA RECERCA BIOMEDICA INSTITUT D'INVESTIGATIONS BIOMEDIQUES AUGUST PI I SUNYER (IDIBAPS): Método y kit para el pronóstico del linfoma de células del manto.
El método y el kit son útiles como herramientas para clasificar un paciente diagnosticado con linfoma […]
PROCEDIMIENTOS Y COMPOSICIONES PARA EL DIAGNÓSTICO Y TRATAMIENTO DEL CÁNCER DE PULMÓN UTILIZANDO EL GEN DE PDGFRA, KIT O KDR COMO MARCADOR GENÉTICO, del 17 de Enero de 2012, de GENENTECH, INC.: Procedimiento para diagnosticar la presencia de un cáncer de pulmón en un mamífero, comprendiendo el procedimiento la detección de si el gen de PDGFRA está […]
MÉTODOS Y SONDAS PARA LA DETECCIÓN DEL CÁNCER, del 12 de Enero de 2012, de VYSIS, INC. MAYO FOUNDATION FOR MEDICAL EDUCATION AND RESEARCH: Un conjunto de sondas cromosómicas que comprende la siguiente combinación de cuatro sondas: una sonda específica del locus 5p15, una sonda específica del locus 8q24, […]
COMPOSICIÓN Y PROCEDIMIENTO PARA LA DETECCIÓN, DIAGNÓSTICO Y TERAPÍA DE NEOPLASIAS HEMATOLÓGICAS, del 22 de Diciembre de 2011, de CORIXA CORPORATION: Uso de una cantidad eficaz de un anticuerpo monoclonal aislado que se une específicamente a un polipéptido que comprende la secuencia expuesta en SEC ID Nº: 4 […]
MÉTODO NORMALIZADO Y OPTIMIZADO DE REACCIÓN EN CADENA DE LA POLIMERASA DE TRANSCRIPTASA INVERSA CUANTITATIVA EN TIEMPO REAL PARA LA DETECCIÓN DE MRD EN LEUCEMIA, del 14 de Diciembre de 2011, de UNIVERSITE DE LA MEDITERRANEE: Conjunto de ácidos nucleicos ABL para RQ-PCR que comprende una sonda nucleotídica que tiene la secuencia SEC ID N.º 2 y cebadores directo e inverso que tienen las secuencias […]
BIOMARCADORES Y PROCEDIMIENTOS PARA DETERMINAR LA SENSIBILIDAD A MODULADORES DEL RECEPTOR DEL FACTOR DE CRECIMIENTO EPIDÉRMICO, del 12 de Diciembre de 2011, de BRISTOL-MYERS SQUIBB COMPANY: Un procedimiento in vitro para predecir la probabilidad de que un mamífero responda terapéuticamente a un procedimiento de tratamiento del […]