ANÁLISIS CROMOSÓMICO DE CARIOTIPADO MOLECULAR.
Un procedimiento de cariotipado de una célula diana humana para detectar desequilibrio cromosómico en la misma,
comprendiendo el procedimiento: (a) examinar los SNP bialélicos adyacentes y cercanos a lo largo del cromosoma de la célula diana (b) comparar los resultados de (a) con el haplotipo de SNP de los cromosomas paterno y materno para montar un haplotipo teórico de los cromosomas de la célula diana de origen paterno y de origen materno (c) evaluar el haplotipo de SNP teórico de los cromosomas de la célula diana de origen paterno y de origen materno para detectar aneuploidía del cromosoma en la célula diana, en el que el haplotipo de SNP teórico de los cromosomas de la célula diana de origen paterno y de origen materno se monta en la etapa (b) usando: (i) alelos de SNP informativos que identifican positivamente a partir de cuál de los cuatro cromosomas paternos y maternos se ha originado un cromosoma en la célula diana, o identifican positivamente a partir de qué cromosoma paterno y de qué cromosoma materno se ha originado un par de cromosomas en la célula diana, y opcionalmente (ii) alelos de SNP semiinformativos que identifican positivamente a partir de cuál de las dos posibles combinaciones de las cuatro combinaciones de pares posibles de cromosomas paternos y maternos, se ha originado un par de cromosomas en la célula diana
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2006/004221.
Solicitante: BlueGnome Limited.
Nacionalidad solicitante: Reino Unido.
Dirección: Unit 5 Breaks House Mill Court Great Shelford, Cambridgeshire CB2 5LD REINO UNIDO.
Inventor/es: HANDYSIDE,Alan.
Fecha de Publicación: .
Fecha Solicitud PCT: 13 de Noviembre de 2006.
Clasificación Internacional de Patentes:
- C12Q1/68B6
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
PDF original: ES-2360085_T3.pdf
Fragmento de la descripción:
Campo técnico
La presente invención se refiere en general a procedimientos y materiales para usar para detectar anomalías del número de cromosomas enteros o regiones de cromosomas (aneuploidía). Tiene utilidad particular para el diagnóstico prenatal, antes de que el embarazo esté establecido en gametos y células tomadas de embriones tempranos, o más tarde en el embarazo en muestras de células de la placenta o el feto.
Antecedentes de la invención
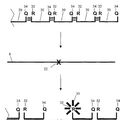
En la meiosis normal las células precursoras del esperma o del óvulo deben multiplicarse y después reducir el número de cromosomas a un grupo de la mitad en cada gameto en dos divisiones meióticas especializadas. Durante las primeras etapas de la meiosis después de la replicación del ADN, las 4 cromátidas duplicadas de una pareja homóloga se alinean cercanas a lo largo de su longitud y pueden intercambiar segmentos, dando como resultado cromosomas no recombinantes (sin intercambio) y recombinantes, generando variación genética. Por lo tanto, los gametos resultantes, contienen cada uno un solo cromosoma que es un recombinante de ambos cromosomas homólogos o no es recombinante y es idéntico a uno de los cromosomas parentales. Esto se muestra en la figura 1(a).
La aneuploidía se define como un número anómalo de cromosomas enteros o partes de cromosomas que produce un desequilibrio genético que puede ser letal en las primeras etapas de desarrollo, producir aborto espontáneo en el embarazo posterior o dar como resultado un embarazo viable pero anómalo. Las aneuploidías más frecuentes y clínicamente significativas implican cromosomas solos (estrictamente “aneusomía”) en las que hay tres (“trisomía”) o sólo uno (“monosomía”) en lugar del par normal de cromosomas.
Al principio del desarrollo, la aneuploidía puede surgir por la segregación cromosómica anómala después de replicación y división celular (1) durante las dos divisiones meióticas que normalmente dan un haploide, la mitad del grupo, en cada gameto antes de la fertilización, o (2) durante las primeras divisiones de la etapa de escisión del embrión fertilizado. La figura 1(b) muestra los efectos de la no disyunción, es decir el fallo de los cromosomas replicados en la separación durante la división, lo cual es una causa común de la segregación de cromosomas anómala, durante las dos divisiones meióticas. El objetivo del cariotipado molecular es identificar anomalías cromosómicas numéricas o estructurales y en particular identificar cualquier desequilibrio en lugar de las dos copias normales de un cromosoma o segmento del cromosoma.
Actualmente hay dos procedimientos básicos para el cariotipado molecular:
El primero es usar marcadores genéticos moleculares, a menudo repeticiones cortas en tándem (STR) altamente polimorfas, para cada uno de los cromosomas parentales. Cuando hay una repetición diferente en cada uno de los cromosomas parentales el marcador de STR es completamente informativo, es decir, es capaz de identificar la presencia de cada cromosoma (en esta posición). Mediante el uso de una serie de marcadores de STR se puede mejorar la confianza en el procedimiento. Un ejemplo del uso de las STR en el análisis de trisomía lo dan Findlay y col. (1998) Journal of Assisted Reproduction and Genetics Vol 15, No 5: 1998: 266-275. Findlay y col. (1998) Journal of Assisted Reproduction and Genetics Vol 15, No 5: 1998: 266-275.
Slater y col. (Am. J. Human Genetics: 77, 709-726, 2005) describen un procedimiento de identificación de anomalías cromosómicas usando SNP. Usan la cuantificación de los SNP frente a una referencia como medida de anomalías.
El segundo procedimiento, de hibridación genómica comparativa (CGH), implica el marcaje fluorescente de ADN genómico de ensayo y normal, y la comparación de las diferencias cuantitativas en las secuencias específicas de cromosomas mediante hibridación de los cromosomas de la metafase o clones de ADN en una micromatriz (matriz CGH). En general, el ADN de ensayo y de referencia se marcan con diferentes fluorocromos y se hibridan en la micromatriz de ADN con una serie de ADN diana clonados, por ejemplo, clones de BAC, derivados de regiones cromosómicas particulares. Dichos “chips” se diseñan para el diagnóstico prenatal y otras aplicaciones de diagnóstico por inducción de BAC informativos, por ejemplo, para los síndromes de eliminación comunes así como aneuploidía y traslocación desequilibradas.
Dada la importancia del cariotipado, puede verse que procedimientos y métodos nuevos relacionados con el cariotipado molecular proporcionarían una contribución a la técnica.
Descripción de la invención
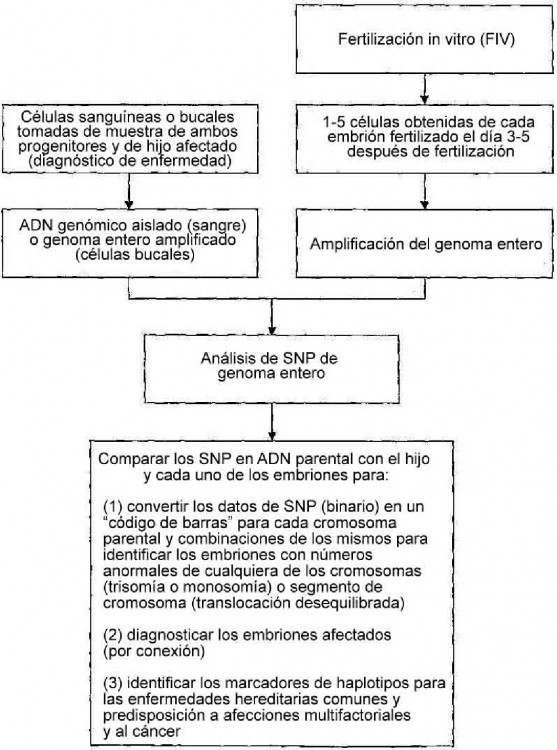
La presente invención describe que el análisis cromosómico por cariotipado molecular (por ejemplo para la detección de trisomía) se puede llevar a cabo usando un análisis de marcadores bialélicos del genoma completo (p. ej., polimorfismos de nucleótido único (SNP) bialélicos) que están distribuidos por el genoma, y que se pueden detectar fácilmente usando tecnologías existentes.
Este descubrimiento es inesperado por varias razones, principalmente porque a priori se supondría que un marcador bialélico (que proporciona solo información binaria en una posición dada en el cromosoma) no podría identificar positivamente la presencia de tres o más cromosomas diferentes.
No obstante, como se describe a continuación, la invención proporciona que este análisis de alta densidad de SNP adyacentes cercanos es capaz de identificar positivamente, entre otros, la presencia de dos cromosomas derivados de un progenitor y que basado en suposiciones bien establecidas sobre la frecuencia y espaciado de sucesos de recombinación entre cromosomas parentales durante la meiosis, esto permitirá la detección precisa de trisomía.
Además, se identifica en cada caso el origen parental del error, lo cual no es posible mediante algunos procedimientos de cariotipado.
Por lo tanto, los procedimientos de la invención no dependen de la cuantificación de las secuencias específicas de cromosomas, como se usa en algunos procedimientos disponibles actualmente, sino que más bien compara los haplotipos de la muestra de ensayo con los haplotipos conocidos de los progenitores. Cuando se combina con los procedimientos existentes para la amplificación del genoma entero, los procedimientos de la invención son particularmente útiles cuando sólo hay un número relativamente pequeño de células de muestra disponible para el análisis.
Por lo tanto, en un aspecto la invención proporciona un procedimiento de cariotipado de una célula diana para detectar desequilibrio cromosómico en la misma, comprendiendo el procedimiento:
(a) examinar SNP bialélicos adyacentes cercanos a lo largo del cromosoma de la célula diana
(b) comparar los resultados de (a) con el haplotipo del SNP de los cromosomas paterno y materno para montar un haplotipo teórico de los cromosomas de la célula diana de origen paterno y de origen materno
(c) evaluar el haplotipo del SNP teórico de los cromosomas de la célula diana de origen paterno y de origen materno para detectar aneuploidía del cromosoma en la célula diana
Como se expone a continuación, el procedimiento también se puede usar para evaluar la recombinación cromosómica, cuando se desee hacerlo.
La célula diana será una de una especie diploide, de reproducción sexual, con un genoma en el que los SNP bialélicos aparecen con una densidad suficiente para proporcionar un haplotipo teórico. Preferiblemente, la célula será una célula aviar, de reptil o mamífera. Más preferiblemente la célula es una célula de mamífero humano o no humano. El mamífero no humano puede ser, por ejemplo, un primate.
En una realización, la célula es humana, y se evalúan al menos 2, 3, 4, 5, 6 o todos los cromosomas humanos seleccionados del siguiente grupo: X, Y, 22, 21, 18, 16 y 13. Los desequilibrios en cualquiera de estos cromosomas se pueden asociar con embarazos viables pero anómalos.
Preferiblemente, se evalúan un total de al menos 10, 15 ó 20 cromosomas. En una realización, se evalúa el genoma entero de la célula diana (p. ej., los 24 cromosomas humanos).
Como se discute a continuación, los SNP se pueden examinar usando técnicas convencionales. A este le puede preceder una o más etapas de amplificación convencionales. Preferiblemente, la frecuencia del alelo menos frecuente de los marcadores... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento de cariotipado de una célula diana humana para detectar desequilibrio cromosómico en la misma, comprendiendo el procedimiento:
(a) examinar los SNP bialélicos adyacentes y cercanos a lo largo del cromosoma de la célula diana
(b) comparar los resultados de (a) con el haplotipo de SNP de los cromosomas paterno y materno para montar un haplotipo teórico de los cromosomas de la célula diana de origen paterno y de origen materno
(c) evaluar el haplotipo de SNP teórico de los cromosomas de la célula diana de origen paterno y de origen materno para detectar aneuploidía del cromosoma en la célula diana, en el que el haplotipo de SNP teórico de los cromosomas de la célula diana de origen paterno y de origen materno se monta en la etapa (b) usando:
(i) alelos de SNP informativos que identifican positivamente a partir de cuál de los cuatro cromosomas paternos y maternos se ha originado un cromosoma en la célula diana, o identifican positivamente a partir de qué cromosoma paterno y de qué cromosoma materno se ha originado un par de cromosomas en la célula diana, y opcionalmente
(ii) alelos de SNP semiinformativos que identifican positivamente a partir de cuál de las dos posibles combinaciones de las cuatro combinaciones de pares posibles de cromosomas paternos y maternos, se ha originado un par de cromosomas en la célula diana.
2. Un procedimiento según la reivindicación 1, en el que:
(i) se examinan al menos 2, 3, 4, 5, 6 ó 7 de los cromosomas seleccionados del grupo que consiste en: X, Y, 22, 21, 18, 16 y 13, o
(ii) se examinan los 24 cromosomas.
3. Un procedimiento según la reivindicación 1 o reivindicación 2, en el que la aneuploidía del cromosoma en la célula diana se detecta en la etapa (c) por:
(c1) evaluación del haplotipo de SNP teórico de los cromosomas de la célula diana de origen paterno y de origen materno y de esta forma asignación de cada cromosoma como recombinante o no recombinante y/o presente en 0, 1 o más copias, (c2) deducción de la aneuploidía del cromosoma en la célula diana cuando la etapa (c1) indica un desequilibrio de cromosomas de origen paterno y de origen materno.
4. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que un cromosoma en la célula diana se identifica como no recombinante cuando los resultados de su haplotipo de SNP teórico están de acuerdo con:
(i) sus alelos de SNP son idénticos a los alelos de SNP de uno de los dos cromosomas paternos o uno de los dos cromosomas maternos a lo largo de la longitud del cromosoma, y
(ii) una ausencia de los alelos de SNP de la alternativa de los dos cromosomas paternos o maternos,
y en el que un cromosoma en la célula diana se identifica como recombinante cuando los resultados de su haplotipo de SNP teórico corresponden a los alelos de SNP de ambos de los dos cromosomas paternos o los dos cromosomas maternos en uno o más segmentos alternantes de acuerdo con la recombinación normal entre los dos cromosomas.
5. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que el haplotipo de SNP teórico de los cromosomas de la célula diana indica la presencia de ambos de los dos cromosomas paternos o los dos cromosomas maternos con un patrón y/o frecuencia de acuerdo con la recombinación normal entre los dos cromosomas, y se deduce que la célula es trisómica para todo o parte del cromosoma o segmento de cromosoma relevante.
6. Un procedimiento según la reivindicación 5, en el que la coherencia con la recombinación normal se evalúa basándose en la probabilidad estadística de la recombinación normal entre alelos de SNP informativos y adyacentes particulares de los dos cromosomas paternos o los dos cromosomas maternos durante la primera división meiótica, y en el que la probabilidad estadística se evalúa basándose en uno o más de los siguientes criterios:
(i) el número medio de sucesos de recombinación para el cromosoma paterno o materno específico,
(ii) distancia entre los sucesos de recombinación aparentes en cada brazo de cromosoma, y su posición relativa unos respecto a otros, el centrómero y el telómero.
7. Un procedimiento según una cualquiera de las reivindicaciones 1 a 4, en el que el haplotipo de SNP teórico de los cromosomas de la célula diana indica
(i) la presencia de un cromosoma de origen paterno y un cromosoma de origen materno, y se deduce que la célula es diploide normal con respecto al cromosoma relevante, o indica
(ii) una ausencia de cualquier cromosoma o segmento de cromosoma de origen paterno y de origen materno, y se deduce que la célula es nulisómica para el cromosoma o segmento de cromosoma relevante, o indica
(iii) una ausencia de un cromosoma o segmento de cromosoma sea de origen paterno o de origen materno pero no ambos, y se deduce que la célula es monosómica para el cromosoma o segmento de cromosoma relevante.
8. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que la distancia media entre los SNP examinados es menor que 0,1, 0,2, 0,3, 0,4 ó 0,5 kb, o menor que 0,1, 0,2, 0,3, 0,4 ó 0,5 cM.
9. Un procedimiento según una cualquiera de las reivindicaciones precedentes, que además comprende la etapa de cuantificación de los alelos de SNP examinados.
10. Un procedimiento según una cualquiera de las reivindicaciones precedentes, que además comprende:
(i) diagnosticar la presencia de una enfermedad genética hereditaria en la célula diana comparando el haplotipo del SNP teórico de la célula diana con los alelos de SNP de los cromosomas paternos y los cromosomas maternos y de uno o más hermanos afectados para diagnosticar la enfermedad en la célula diana por conexión y/o
(ii) diagnosticar la susceptibilidad a una enfermedad común o al cáncer en la célula diana comparando el haplotipo de SNP teórico con un haplotipo que se sabe que está asociado con dicha enfermedad.
11. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que el haplotipo de SNP de cromosomas paternos y maternos se obtiene por el análisis del haplotipo de SNP de células obtenidas de embriones fertilizados hermanos después de fertilización in vitro (FIV) después de la amplificación del genoma entero o de análisis de múltiples gametos haploides parentales individuales después de la amplificación del genoma entero.
12. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que la célula diana se ha obtenido de un embrión de mamífero que opcionalmente es resultado de una FIV y opcionalmente es un embrión preimplantado.
13. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que se proporciona y se ensaya un número igual a, o de al menos 1, 2, 3, 4 ó 5 células diana.
14. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que al examen de SNP le precede la amplificación del genoma entero.
15. Un procedimiento según una cualquiera de las reivindicaciones precedentes, en el que el examen de SNP se lleva a cabo mediante un chip de oligonucleótidos.
16. Un medio usable con ordenador que tiene un código del programa almacenado en el mismo que puede ser leído por el ordenador, para hacer que un ordenador ejecute un procedimiento para determinar la aneuploidía o recombinación cromosómica en una célula diana, cuyo procedimiento comprende:
(a) acceder a una base de datos que comprende los datos de genotipo obtenidos de una pluralidad de locus de SNP bialélicos adyacentes y cercanos presentes en un cromosoma de la célula diana,
(b) acceder a una base de datos que comprende los datos de haplotipo de SNP de los correspondientes cromosomas paternos y maternos,
(c) comparar los datos de SNP de la célula diana de la base de datos de la etapa (a) con los datos de haplotipo de SNP de la base de datos de la etapa (b) para montar un haplotipo teórico de regiones de cromosomas de la célula diana de origen paterno y de origen materno,
(d) evaluar el haplotipo de SNP teórico de los cromosomas de la célula diana de origen paterno y
origen materno para determinar la aneuploidía o recombinación cromosómica del cromosoma en la célula diana.
17. Un medio usable con ordenador según la reivindicación 16, en el que a cada locus de SNP de los SNP “x” de la base de datos en la etapa (b) se le asigna un valor “n” de acuerdo con cuál de las 16 combinaciones de los 4 alelos de SNP parentales está presente en ese locus, y en el que la etapa (c) comprende montar un haplotipo teórico en este locus comparando:
(i) los datos de genotipo para el SNP bialélico en ese locus de la base de datos de la etapa (a) y,
(ii) el valor “n” en ese locus de la base de datos de la etapa (b) con,
(iii) una tabla de origen cromosómico,
y de esta forma asignar el locus de los cromosomas de la célula diana como procedentes de un cromosoma paterno
o materno.
18. Un medio usable con ordenador según la reivindicación 16 ó 17, en el que el haplotipo de SNP teórico de regiones de cromosomas de la célula diana de origen paterno y de origen materno se monta usando un subconjunto de los locus de SNP “x” de la base de datos en la etapa (b), cuyo subconjunto consiste en:
(i) alelos de SNP informativos que identifican positivamente cuál de los 4 cromosomas paternos y maternos ha originado un cromosoma en la célula diana, o identifican positivamente qué cromosoma paterno y qué cromosoma materno han originado un par de cromosomas en la célula diana, y opcionalmente
(ii) alelos de SNP semiinformativos que identifican positivamente cuál de las dos posibles combinaciones de las 4 posibles combinaciones de pares de cromosomas paternos y maternos ha originado un par de cromosomas en la célula diana, en el que se fija un número umbral de SNP informativos y opcionalmente semiinformativos positivos y negativos, y se determina un cariotipo sólo cuando se supera este número.
19. Un medio usable con ordenador según una cualquiera de las reivindicaciones 16 a 18, en el que:
el cromosoma en la célula diana se identifica como no recombinante cuando los resultados de su haplotipo de SNP teórico están de acuerdo con:
(i) sus alelos de SNP son idénticos a los alelos de SNP de uno de los dos cromosomas paternos o uno de los dos cromosomas maternos a lo largo de la longitud del cromosoma, y
(ii) una ausencia de los alelos de SNP de la alternativa de los dos cromosomas paternos o maternos, y en el que el cromosoma en la célula diana se identifica como recombinante
en el que los resultados de su haplotipo de SNP teórico corresponden a los alelos de SNP de ambos de los dos cromosomas paternos o los dos cromosomas maternos en uno o más segmentos alternantes de acuerdo con la recombinación normal entre los dos cromosomas.
20. Un medio usable con ordenador según una cualquiera de las reivindicaciones 16 a 19, en el que:
(i) el cromosoma en la célula diana se identifica como trisómico para el cromosoma cuando el haplotipo de SNP teórico del cromosoma de la célula diana indica la presencia de ambos de los dos cromosomas paternos o los dos cromosomas maternos con un patrón y/o frecuencia no concordante con la recombinación normal entre los dos cromosomas;
(ii) el cromosoma en la célula diana se identifica como nulisómico para el cromosoma cuando el haplotipo de SNP teórico del cromosoma de la célula diana indica una ausencia del cromosoma o un segmento del mismo tanto de origen paterno como de origen materno;
(iii) el cromosoma en la célula diana se identifica como monosómico para el cromosoma cuando el haplotipo de SNP teórico del cromosoma de la célula diana indica una ausencia del cromosoma o un segmento del mismo de origen paterno y de origen materno, pero no de ambos.
21. Un sistema para el cariotipado de una célula diana para detectar desequilibrio cromosómico en la misma, comprendiendo el sistema:
(i) medios para examinar los SNP bialélicos adyacentes y cercanos a lo largo del cromosoma una célula diana, que es un chip de oligonucleótidos,
(ii) un ordenador programado para,
(1) ejecutar un procedimiento para determinar la aneuploidía o recombinación cromosómica en una
célula diana, cuyo procedimiento comprende: 5
(a) acceder a una base de datos que comprende datos de genotipo obtenidos de una pluralidad de locus de SNP bialélicos adyacentes y cercanos presentes en un cromosoma de la célula diana,
(b) acceder a una base de datos que comprende datos de haplotipo de SNP de los 10 correspondientes cromosomas paternos y maternos,
(c) comparar los datos de SNP de la célula diana de la base de datos de la etapa (a) con los datos de haplotipo de SNP de la base de datos de la etapa (b) para montar un haplotipo teórico de regiones de los cromosomas de la célula diana de origen paterno y de origen materno, (d) evaluar el haplotipo de SNP teórico de los cromosomas de la célula diana de origen paterno y de origen materno para determinar aneuploidía o recombinación cromosómica del cromosoma en la célula diana:
o,
(2) programado con un medio usable con ordenador como se reivindica en cualquiera de las 20 reivindicaciones 16 a 20.
Patentes similares o relacionadas:
INFIDELIDAD DE LA TRANSCRIPCIÓN, SU DETECCIÓN Y SUS USOS, del 3 de Febrero de 2012, de TRANSMEDI SA: Un método para detectar la predisposición o la etapa de un cáncer en un sujeto mamífero, comprendiendo dicho método evaluar in vitro o ex vivo la presencia o la […]
 CONJUNTO DE CEBADORES, SONDAS, PROCEDIMIENTO Y KIT PARA EL GENOTIPADODEL POLIMORFISMO GENETICO S19W DEL GEN APO A5, del 30 de Mayo de 2011, de UNIVERSIDAD DE MALAGA: Conjunto de cebadores, sondas, procedimiento y kit para el genotipado del polimorfismo genético S19W del gen APO A5.La presente invención se refiere […]
CONJUNTO DE CEBADORES, SONDAS, PROCEDIMIENTO Y KIT PARA EL GENOTIPADODEL POLIMORFISMO GENETICO S19W DEL GEN APO A5, del 30 de Mayo de 2011, de UNIVERSIDAD DE MALAGA: Conjunto de cebadores, sondas, procedimiento y kit para el genotipado del polimorfismo genético S19W del gen APO A5.La presente invención se refiere […]
 PROCEDIMIENTO PARA EL ANÁLISIS DE ÁCIDOS NUCLEICOS AMPLIFICADOS, del 19 de Mayo de 2011, de SIEMENS AKTIENGESELLSCHAFT: Procedimiento para el análisis de ácidos nucleicos en un dispositivo microfluídico, cuyo procedimiento presenta las etapas siguientes: a) la […]
PROCEDIMIENTO PARA EL ANÁLISIS DE ÁCIDOS NUCLEICOS AMPLIFICADOS, del 19 de Mayo de 2011, de SIEMENS AKTIENGESELLSCHAFT: Procedimiento para el análisis de ácidos nucleicos en un dispositivo microfluídico, cuyo procedimiento presenta las etapas siguientes: a) la […]
 MÉTODO PARA LA DETECCIÓN DE ALELOS MUTANTES COMBINANDO PCR EN TIEMPO REAL Y REMS-PCR, del 17 de Marzo de 2011, de DIASORIN S.P.A: Método para detectar la presencia o la ausencia de una secuencia de ADN mutante en una muestra, comprendiendo el método las etapas de: a) poner en contacto a la muestra […]
MÉTODO PARA LA DETECCIÓN DE ALELOS MUTANTES COMBINANDO PCR EN TIEMPO REAL Y REMS-PCR, del 17 de Marzo de 2011, de DIASORIN S.P.A: Método para detectar la presencia o la ausencia de una secuencia de ADN mutante en una muestra, comprendiendo el método las etapas de: a) poner en contacto a la muestra […]
 MÉTODO PARA LA DETERMINACIÓN DEL GENOTIPO DE ANTÍGENOS DE CÉLULAS DE LA SANGRE Y KIT ADECUADO PARA LA DETERMINACIÓN DEL GENOTIPO DE ANTÍGENOS DE CÉLULAS DE LA SANGRE, del 24 de Febrero de 2011, de STICHTING SANQUIN BLOEDVOORZIENING: Método de determinación del genotipo de antígenos de células de la sangre que comprende someter al ADN de un individuo de una especie de mamífero a […]
MÉTODO PARA LA DETERMINACIÓN DEL GENOTIPO DE ANTÍGENOS DE CÉLULAS DE LA SANGRE Y KIT ADECUADO PARA LA DETERMINACIÓN DEL GENOTIPO DE ANTÍGENOS DE CÉLULAS DE LA SANGRE, del 24 de Febrero de 2011, de STICHTING SANQUIN BLOEDVOORZIENING: Método de determinación del genotipo de antígenos de células de la sangre que comprende someter al ADN de un individuo de una especie de mamífero a […]
 MÉTODO PARA LA DETECCIÓN DE UNA ALTERACIÓN EN UN ÁCIDO NUCLÉICO, del 18 de Febrero de 2011, de GENZYME CORPORATION: Un método para detectar una alteración en un ácido nucleico diana que se sospecha que está en una muestra biológica, comprendiendo el método las etapas […]
MÉTODO PARA LA DETECCIÓN DE UNA ALTERACIÓN EN UN ÁCIDO NUCLÉICO, del 18 de Febrero de 2011, de GENZYME CORPORATION: Un método para detectar una alteración en un ácido nucleico diana que se sospecha que está en una muestra biológica, comprendiendo el método las etapas […]
METODO PARA LA DETECCION DEL RIESGO DE DESARROLLAR VITREORRETINOPATIAPROLIFERANTE (VRP), del 24 de Enero de 2011, de UNIVERSIDADE DE SANTIAGO DE COMPOSTELA UNIVERSIDAD DE VALLADOLID: Método para la detección del riesgo de desarrollar vitreorretinopatía proliferante (VRP).La presente invención se refiere a un método que permite identificar […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]