MÉTODO PARA LA DETERMINACIÓN DEL GENOTIPO DE ANTÍGENOS DE CÉLULAS DE LA SANGRE Y KIT ADECUADO PARA LA DETERMINACIÓN DEL GENOTIPO DE ANTÍGENOS DE CÉLULAS DE LA SANGRE.
Método de determinación del genotipo de antígenos de células de la sangre que comprende someter al ADN de un individuo de una especie de mamífero a una Reacción en Cadena de la Polimerasa (PCR) múltiple para amplificar y marcar de forma detectable una región del locus de,
como mínimo, dos antígenos diferentes de células de la sangre que contiene el sitio de polimorfismo de nucleótido de dicho antígeno de células de la sangre y utilizar los fragmentos de ADN amplificados y marcados de esta manera para determinar el genotipo de cada uno de dichos antígenos de células de la sangre, comprendiendo dicha PCR múltiple el uso de, como mínimo, un par de cebadores quiméricos específicos de antígenos de células de la sangre para que sea determinado el genotipo de cada antígeno de células de la sangre y, como mínimo, un cebador universal marcado de forma detectable, en el que dicho, como mínimo, un cebador universal tiene una secuencia única que no aparece en el ADN de dicha especie de mamíferos, y en el que cada par de cebadores quiméricos comprende un cebador quimérico de la izquierda y un cebador quimérico de la derecha, cada uno de ellos comprende una parte específica del antígeno de célula de la sangre en el extremo 3' y una parte universal en el extremo 5', en el que la secuencia de bases de la parte universal de los cebadores quiméricos se corresponde con la secuencia de bases de dicho, como mínimo, un cebador universal, y en el que dichas partes específicas del antígeno de células de la sangre del par de cebadores quiméricos comprende una región del locus del antígeno de célula de la sangre que contiene el sitio del polimorfismo de nucleótido de dicho antígeno de células de la sangre
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/NL2005/000236.
Solicitante: STICHTING SANQUIN BLOEDVOORZIENING.
Nacionalidad solicitante: Países Bajos.
Dirección: PLESMANLAAN 125 1066 CX AMSTERDAM PAISES BAJOS.
Inventor/es: DEN DUNNEN, JOHANNES, THEODORUS, WIERINGA-JELSMA,HENDRIKA, DE HAAS,MASCHENKA, BEIBOER,SIGRID,HERMA,WILMA.
Fecha de Publicación: .
Fecha Solicitud PCT: 31 de Marzo de 2005.
Clasificación Internacional de Patentes:
- C12Q1/68B6
- C12Q1/68D4
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania.
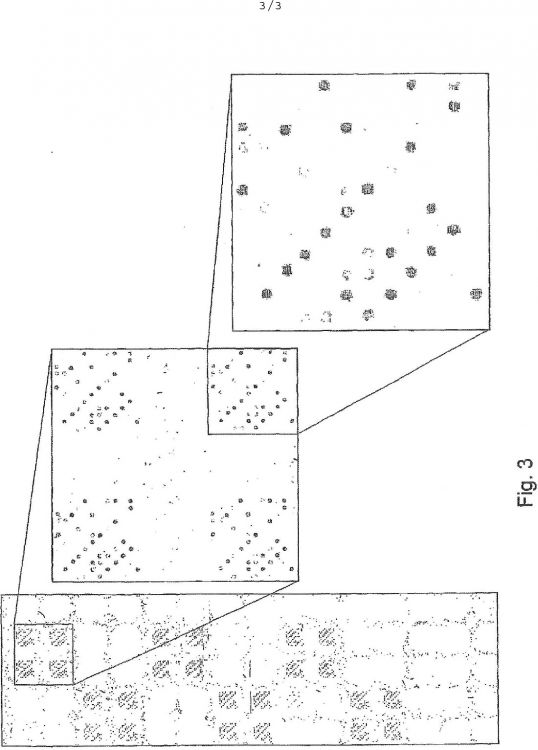
Fragmento de la descripción:
SECTOR DE LA INVENCIÓN
La presente invención se encuentra en el sector de la determinación del genotipo de los antígenos de células de la sangre, más en particular de los antígenos de los glóbulos rojos (antígenos de grupo sanguíneo), de plaquetas de la sangre (antígenos de plaquetas) y de leucocitos (antígenos de leucocitos).
La presente invención da a conocer un método de determinar el genotipo de antígenos de las células sanguíneas y un kit adecuado para determinar el genotipo de los antígenos de las células sanguíneas, y da a conocer los conjuntos de los cebadores y de las sondas útiles para la determinación de los antígenos de las células sanguíneas.
ANTECEDENTES DE LA INVENCIÓN
Cuando la sangre, o una fracción de la sangre, tal como glóbulos rojos (los eritrocitos o las células rojas), las plaquetas (trombocitos) y los glóbulos blancos (leucocitos), derivados de un donante se administran a otra persona (o de forma más general, a otro individuo mamífero), pueden ocurrir reacciones adversas serias cuando la sangre o una fracción de la sangre del donante no coincide correctamente con la sangre del receptor. Se conocen bien las reacciones de transfusión (aglutinación) que ocurren cuando por ejemplo la sangre de un donante del grupo sanguíneo A se da a una persona del grupo sanguíneo B (los antígenos A y B del grupo sanguíneo pertenecen al sistema ABO). Cuando la sangre de un donante Rhesus D (RhD) positivo se da a un paciente de RhD negativo hay una alta probabilidad de formación del aloanticuerpos. Los anticuerpos de RhD llevan a la destrucción rápida de glóbulos rojos RhD-positivos y a las reacciones de transfusión. Además, cuando una mujer con anticuerpos de glóbulos rojos o plaquetas se queda embarazada, estos anticuerpos pueden atravesar la placenta y destruir los glóbulos rojos o las plaquetas del feto. Esto puede llevar a una hemólisis severa resultando en anemia, ictericia (después del nacimiento) y si no se trata puede ser fatal o conducir a daño cerebral. Así, para evitar las reacciones de transfusión, la política actual de transfusión consiste en transfundir sólo los glóbulos rojos que coincidan en ABO y RhD. Para evitar la formación de aloanticuerpos en las mujeres en edad fértil con las posibles complicaciones durante el embarazo, sólo se transfunden glóbulos rojos que coinciden en el tipo ABO, RhD y K1.
Posiblemente, en el futuro, también se den glóbulos rojos que coincidan en RhC y RhE a mujeres en edad fértil.
Existen sin embargo otros antígenos de grupo sanguíneo (antígenos de glóbulos rojos) y estos pueden causar también serios problemas cuando la sangre del donante no coincidente se da a receptores con aloanticuerpos. La determinación del tipo de antígenos específicos de plaquetas es importante para el diagnóstico y la terapia de los pacientes, ya que pueden ocurrir diferentes síndromes de trombocitopenia aloinmune. Si una mujer ha desarrollado anticuerpos de antígeno antiplaquetas (en la mayoría de los casos anticuerpos de Antígeno de Plaqueta Humana (HPA) de tipo 1a), desarrollados principalmente durante la gestación, estos anticuerpos pueden conducir a la destrucción de las plaquetas del feto con una tendencia creciente de la hemorragia en el feto. En un número de casos, esto llevará a una hemorragia intracraneal. Para prevenir la hemorragia, es necesario transfundir plaquetas HPA-1anegativas.
Las plaquetas se transfunden habitualmente sin determinar previamente el tipo de antígeno de plaqueta [antígenos de plaqueta humana (HPA) en humanos] del donante y el receptor. Las transfusiones de sangre o fracciones de sangre no coincidentes pueden provocar la generación de aloanticuerpos, especialmente en pacientes que necesitan transfusiones frecuentes o recurrentes de glóbulos rojos o plaquetas (o leucocitos). Si se forman aloanticuerpos múltiples, o si los aloanticuerpos están dirigidos contra antígenos de alta frecuencia, puede ser un problema encontrar glóbulos rojos o plaquetas compatibles. De acuerdo con un estudio publicado (Seltsam y otros, 2003), el apoyo de transfusión no fue satisfactorio en aproximadamente un tercio de los pacientes hospitalizados con anticuerpos a antígenos de alta frecuencia.
El método clásico de prueba de los antígenos y anticuerpos del grupo sanguíneo es la determinación del fenotipo por la prueba de hemoaglutinación. La técnica de esta prueba serológica es simple y barata, pero sus costes y dificultades se incrementan cuando se necesita realizar múltiples ensayos para una determinación completa del tipo y requiere de la disponibilidad de un gran número de antisueros específicos. Sólo en los Países Bajos, el número total de donantes de sangre está en el orden de magnitud de 500.000 personas y se estima que el número de donantes se incrementa cada año en unos 60.000 nuevos donantes. Para los glóbulos rojos, existen, como mínimo, 29 sistemas de atígenos de grupo sanguíneo (cada uno con un número de alelos diferentes). Además, será relevante determinar los antígenos de alta frecuencia y los antígenos de baja frecuencia. El número de sistemas de antígeno de células de la sangre clínicamente relevantes es de aproximadamente 60. La determinación completa de fenotipos de todos los donantes de sangre es por tanto cara, laboriosa, lenta y no factible debido a la falta de suficientes reactivos de determinación de tipos.
La base molecular de la mayoría de los sistemas de antígenos de células de sangre es conocida. La mayoría de los antígenos de grupos sanguíneos, antígenos de plaquetas y antígenos de neutrófilos son bialélicos y son el resultado de un polimorfismo de nucleótido simple (SNP). Estos SNP se pueden utilizar para la determinación del genotipo. En la literatura científica se han descrito innumerables ensayos basados en ADN para la determinación del genotipo de antígenos de grupos sanguíneos y de plaquetas. Estos incluyen PCR-RFLP, PCR específicas de alelos, PCR específicas de secuencias en forma de ensayos simples o múltiples, PCR cuantitativa en tiempo real, el método de extensión de terminadores fluorescentes de nucleótido único y tecnología de perlas de alto rendimiento (revisado por Reid, 2003). Métodos semiautomatizados que utilizan un espectrofotómetro de masas o un pirosecuenciador se pueden utilizar también para la determinación del genotipo.
Por ejemplo, Randen y otros (2003) dieron a conocer recientemente la determinación del genotipo de los antígenos de plaquetas humanas 1, 2, 3, 4, 5 y Gov (llamado recientemente HPA15, Metcalfe y otros, 2003) a través del análisis de la curva de fusión usando la tecnología de LightCycler. Los sistemas bialélicos HPA-1 a 5 y Gov son los más frecuentemente involucrados en la enfermedad (Berry y otros, 2000), haciéndolos dianas importantes para la determinación del genotipo. La tecnología de LightCycler involucra una amplificación de fragmentos relevantes de fragmentos de ADN del donante por PCR usando un par de cebadores específicos para cada uno de los antígenos de plaquetas mencionados anteriormente. Utilizando sondas de hibridación fluorescentes y el análisis de curvas de fusión fue posible lograr la detección simultánea de ambos alelos de un antígeno de plaqueta en el mismo capilar, sin necesidad de la laboriosa y lenta electroforesis en gel de la anterior metodología de determinación de genotipo.
Sin embargo, tal como en otros sistemas descritos de determinación de genotipos de antígenos de células de la sangre, la tecnología LightCycler no es capaz de realizar la enorme tarea de determinación completa del genotipo de los donantes de sangre, lo que requeriría una metodología que no es apropiada para el cribado de alto rendimiento. La determinación del tipo de 60.000 donantes cada año después de dos donaciones diferentes por 60 antígenos de grupos sanguíneos y de plaquetas implicaría más de 7 millones de ensayos de determinación de tipo por año o aproximadamente 140.000 ensayos de determinación de tipo por semana. Un método apropiado de alto rendimiento, que es rápido y fiable, es altamente deseable para esta prueba. Reacción en cadena de la polimerasa múltiple
La técnica de la Reacción en Cadena de la Polimerasa (PCR) se ha desarrollado mucho desde que se introdujo en los años
80. Chamberlain y otros (1988) dieron a conocer la PCR múltiple como una técnica general para la amplificación de múltiples locus en el ADN (genómico). Aquí, en vez de un par de cebadores para la amplificación de un locus, se adicionan más pares de cebadores...
Reivindicaciones:
1. Método de determinación del genotipo de antígenos de células de la sangre que comprende someter al ADN de un individuo de una especie de mamífero a una Reacción en Cadena de la Polimerasa (PCR) múltiple para amplificar y marcar de forma detectable una región del locus de, como mínimo, dos antígenos diferentes de células de la sangre que contiene el sitio de polimorfismo de nucleótido de dicho antígeno de células de la sangre y utilizar los fragmentos de ADN amplificados y marcados de esta manera para determinar el genotipo de cada uno de dichos antígenos de células de la sangre, comprendiendo dicha PCR múltiple el uso de, como mínimo, un par de cebadores quiméricos específicos de antígenos de células de la sangre para que sea determinado el genotipo de cada antígeno de células de la sangre y, como mínimo, un cebador universal marcado de forma detectable, en el que dicho, como mínimo, un cebador universal tiene una secuencia única que no aparece en el ADN de dicha especie de mamíferos, y en el que cada par de cebadores quiméricos comprende un cebador quimérico de la izquierda y un cebador quimérico de la derecha, cada uno de ellos comprende una parte específica del antígeno de célula de la sangre en el extremo 3' y una parte universal en el extremo 5', en el que la secuencia de bases de la parte universal de los cebadores quiméricos se corresponde con la secuencia de bases de dicho, como mínimo, un cebador universal, y en el que dichas partes específicas del antígeno de células de la sangre del par de cebadores quiméricos comprende una región del locus del antígeno de célula de la sangre que contiene el sitio del polimorfismo de nucleótido de dicho antígeno de células de la sangre.
2. Método, según la reivindicación 1, en el que dicho, como mínimo, un cebador universal marcado de forma detectable se utiliza en una cantidad molar que es, como mínimo, 100 veces, más preferentemente, como mínimo, 200 veces la cantidad molar de cada cebador quimérico.
3. Método, según la reivindicación 1 ó 2, en el que se utiliza un par de cebadores universales marcados de forma detectable con una secuencia única que no aparece en el ADN de dicha especie de mamíferos y en el que para cada par de cebadores quiméricos la secuencia de bases de la parte universal de un miembro del par de
cebadores quiméricos se corresponde con la secuencia de bases de un miembro del par de cebadores universales y la secuencia de bases de la parte universal del otro miembro del par de cebadores quiméricos se corresponde con la secuencia de bases del otro miembro del par de cebadores universales.
4. Método, según la reivindicación 3, en el que uno de dichos cebadores universales tiene la secuencia de bases gccgcgaattcactagtg (SEQ ID No:: 2) y el otro cebador universal tiene la secuencia de bases ggccgcgggaattcgatt (SEQ ID No:: 1).
5. Método, según cualquiera de las reivindicaciones 14, en el que cada cebador universal porta un marcador fluorescente, preferentemente Cy5, en su extremo 5'.
6. Método, según cualquiera de las reivindicaciones 15, en el que dicha especie de mamíferos es humana, dicho ADN es ADN genómico, y dichos antígenos de células de la sangre comprenden, como mínimo, dos, preferentemente todos, los miembros seleccionados del grupo que comprende HPA1, HPA2, HPA3, HPA4, HPA5, Gov, JK1/2, FY1/2, GATAbox, KEL1/2, KEL3/4, RHCEex2/RHex2, RHCEex5/RHex5, RHDψ, RHD, BigC, MN, U y JO.
7. Método, según la reivindicación 6, en el que dichos cebadores quiméricos comprenden, como mínimo, dos, preferentemente substancialmente todos, los pares seleccionados del grupo que comprende:
HPA1-izquierdo gccgcgaattcactagtgcttcaggtcacagcgaggt SEQ ID No: 3 HPA1-derecho ggccgcgggaattcgattgctccaatgtacggggtaaa SEQ ID No: 4 HPA2-izquierdo gccgcgaattcactagtgtgaaaggcaatgagctgaag SEQ ID No: 5 HPA2-derecho ggccgcgggaattcgattagccagactgagcttctcca SEQ ID No: 6 HPA3-izquierdo gccgcgaattcactagtggcctgaccactcctttgc SEQ ID No: 7 HPA3-derecho ggccgcgggaattcgattggaagatctgtctgcgatcc SEQ ID No: 8 HPA4-izquierdo gccgcgaattcactagtgatccgcaggttactggtgag SEQ ID No: 9
HPA4-derecho ggccgcgggaattcgattccatgaaggatgatctgtgg SEQ ID No: 10 HPA5-izquierdo gccgcgaattcactagtgtccaaatgcaagttaaattaccag SEQ ID No: 11 HPA5-derecho ggccgcgggaattcgattacagacgtgctcttggtaggt SEQ ID No: 12 HPA15-izquierdo gccgcgaattcactagtgtgtatcagttcttggttttgtgatg SEQ ID No: 13 HPA15-derecho ggccgcgggaattcgattaaaaccagtagccacccaag SEQ ID No: 14 JK1/2-izquierdo gccgcgaattcactagtggtctttcagccccatttgag SEQ ID No: 15 JK1/2-derecho ggccgcgggaattcgattgttgaaaccccagagtccaa SEQ ID No: 16 FY1/2-izquierdo gccgcgaattcactagtggaattcttcctatggtgtgaatga SEQ ID No: 17 FY1/2-derecho ggccgcgggaattcgattaagaagggcagtgcagagtc SEQ ID No: 18 GATAbox-izquierdo gccgcgaattcactagtgggccctcattagtccttgg SEQ ID No: 19 GATAbox-derecho ggccgcgggaattcgattgaaatgaggggcatagggata SEQ ID No: 20 Fyx-izquierdo gccgcgaattcactagtgtcatgcttttcagacctctcttc SEQ ID No: 175 Fyx-derecho ggccgcgggaattcgattcaagacgggcaccacaat SEQ ID No: 176 KEL1/2-izquierdo gccgcgaattcactagtgaagggaaatggccatactga SEQ ID No: 21 KEL1/2-derecho ggccgcgggaattcgattagctgtgtaagagccgatcc SEQ ID No: 22 KEL3/4-izquierdo gccgcgaattcactagtggcctcagaaactggaacagc SEQ ID No: 23 KEL3/4-derecho ggccgcgggaattcgattagcaaggtgcaagaacactct SEQ ID No: 24 KEL6/7-izquierdo gccgcgaattcactagtggcagcaccaaccctatgttc SEQ ID No: 177 KEL6/7-derecho ggccgcgggaattcgatttcaggcacaggtgagcttc SEQ ID No: 178 RHCEex2for gccgcgaattcactagtgcgtctgcttccccctcc SEQ ID No: 25 RHex2rev ggccgcgggaattcgattctgaacagtgtgatgaccacc SEQ ID No: 26 RHDex3-izquierdo gccgcgaattcactagtgtcctggctctccctctct SEQ ID No: 179 RHCEex3-derecho ggccgcgggaattcgatttttttcaaaaccccggaag SEQ ID No: 180 RHCEex5-izquierdo gccgcgaattcactagtgggatgttctggccaagtg SEQ ID No: 27 RHex5rev ggccgcgggaattcgattggctgtcaccacactgactg SEQ ID No: 28 RHDψ-izquierdo ggccgcgggaattcgattgtagtgagctggcccatca SEQ ID No: 29RHDψ-derecho gccgcgaattcactagtgtgtctagtttcttaccggcaagtSEQ ID No: 30 RHD-izquierdoB gccgcgaattcactagtgttataataacacttgtccacaggg
SEQ ID No: 31 RHD-derechoC ggccgcgggaattcgattcggctccgacggtatc SEQ ID No: 32 BigC-izquierdo gccgcgaattcactagtgggccaccaccatttgaa SEQ ID No: 33 BigC-intrónderecho2 ggccgcgggaattcgattccatgaacatgccacttcac SEQ ID No: 34 RhDVI-izquierdo (dir)
ggccgcgggaattcgattctttgaattaagcacttcacagaSEQ ID No: 181 RhDVI-derecho (inv) gccgcgaattcactagtggccagaatcacactcctgct SEQ ID No: 182 MN-izquierdo gccgcgaattcactagtgtgagggaatttgtcttttgca SEQ ID No: 35 MN-derecho ggccgcgggaattcgattcagaggcaagaattcctcca SEQ ID No: 36 Ss-izquierdo gccgcgaattcactagtgtttttctttgcacatgtcttt SEQ ID No: 183 Ss-derecho ggccgcgggaattcgatttctttgtctttacaatttcgtgtg
SEQ ID No: 184 U-izquierdo gccgcgaattcactagtgcgctgatgttatctgtcttatttttc
SEQ ID No: 37 U-derecho ggccgcgggaattcgattgatcgttccaataataccagcc SEQ ID No: 38 DO-izquierdo ggccgcgggaattcgatttgatccctccctatgagctg SEQ ID No: 185 DO-derecho gccgcgaattcactagtgttatatgtgctcaggttcccagtSEQ
ID No: 186 JO-izquierdo gccgcgaattcactagtgcctggcttaaccaaggaaaa SEQ ID No: 39 JO-derecho ggccgcgggaattcgatttcatactgctgtggagtcctg SEQ ID No: 40 Colton-izquierdo gccgcgaattcactagtggccacgaccctctttgtct SEQ ID No: 187 Colton-derecho ggccgcgggaattcgatttacatgagggcacggaagat SEQ ID No: 188 Diego-izquierdo gccgcgaattcactagtgacttattcacgggcatccag SEQ ID No: 189 Diego-derecho ggccgcgggaattcgattaagctccacgttcctgaaga SEQ ID No: 190 Wr-izquierdo gccgcgaattcactagtgggcttcaaggtgtccaactc SEQ ID No: 636 Wr-derecho ggccgcgggaattcgattaggatgaagaccagcagagc SEQ ID No: 637 Yt-izquierdo ggccgcgggaattcgattccttcgtgcctgtggtagat SEQ ID No: 638 Yt-derecho gccgcgaattcactagtgttctgggacttctgggaatg SEQ ID No: 639Lu-izquierdo gccgcgaattcactagtgggacccagagagagagagactg SEQ ID No: 640 Lu-derecho ggccgcgggaattcgattgggagtccagctggtatgg SEQ ID No: 641.
8. Método, según cualquiera de las reivindicaciones 17, en el que la polimerasa de ADN utilizada en la PCR múltiple es polimerasa Taq o una polimerasa similar resistente al calor y cada ciclo por PCR múltiple comprende una etapa de desnaturalización por calor de 15 a 60 segundos a una temperatura de 90 a 98°C
(preferentemente aproximadamente de 30 segundos a aproximadamente 94°C), una etapa de alineamiento de 60 a 120 segundos a una temperatura de 54 a 60°C (preferentemente aproximadamente de 90 segundos a aproximadamente 57°C), y una etapa de extensión del cebador de 60 a 120 segundos a una temperatura de 68 a 76°C (preferentemente aproximadamente de 90 segundos a aproximadamente 72°C).
9. Método, según cualquiera de las reivindicaciones 18, en el que dicha PCR múltiple utiliza, sobre la base de un volumen de reacción de 50 µL, aproximadamente 100 ng de ADN genómico de dicho individuo, aproximadamente 5 nM de cada cebador quimérico, por par de cebadores quiméricos aproximadamente 0,2 µM de cada cebador universal marcado de forma detectable y aproximadamente 25 µL de tampón que contiene MasterMix 2x, dNTP y ADN polimerasa.
10. Método, según cualquiera de las reivindicaciones 19, en el que el genotipo para cada uno de dichos antígenos de células de la sangre se determina por hibridación de los productos de la amplificación por PCR múltiple, después de la desnaturalización, con las sondas de oligonucleótidos específicas de alelo del antígeno de células de la sangre contenidas en un arreglo de ADN o presentadas en cualquier otra forma, tal como soportadas en perlas, y analizado el patrón de hibridación.
11. Método, según la reivindicación 10, en el que las sondas específicas de alelo tienen una longitud de 15 a 40 nucleótidos, preferentemente de 17 a 30 nucleótidos.
12. Método, según la reivindicación 10 u 11, en el que el arreglo comprende para cada alelo de un antígeno de células de la sangre, como mínimo, dos, preferentemente, como mínimo, cinco, sondas sentido diferentes, y, como mínimo, dos, preferentemente, como mínimo cinco, sondas antisentido diferentes, cada una de las cuales cubre el sitio del polimorfismo de nucleótidos pero en varias posiciones.
13. Método, según la reivindicación 11, en el que el arreglo comprende, como mínimo, 72 sondas diferentes, preferentemente substancialmente todas las sondas, seleccionadas del grupo que comprende:
Alelo HPA1 a: HPA-1aa tacaggccctgcctctggg SEQ ID No: 41 HPA-1ab aggccctgcctctgggct SEQ ID No: 42 HPA-1ac ccctgcctctgggctcacc SEQ ID No: 43 HPA-1ad tgcctctgggctcacctcg SEQ ID No: 44 HPA-1ae ctctgggctcacctcgctg SEQ ID No: 45 HPA-1aa CR cagcgaggtgagcccagag SEQ ID No: 46 HPA-1ab CR cgaggtgagcccagaggca SEQ ID No: 47 HPA-1ac CR ggtgagcccagaggcaggg SEQ ID No: 48 HPA-1ad CR agcccagaggcagggcct SEQ ID No: 49 HPA-1ae CR cccagaggcagggcctgta SEQ ID No: 50
Alelo HPA1 b: HPA-1ba tacaggccctgcctccggg SEQ ID No: 51 HPA-1bb aggccctgcctccgggct SEQ ID No: 52 HPA-1bc ccctgcctccgggctcac SEQ ID No: 53 HPA-1bd ctgcctccgggctcacct SEQ ID No: 54 HPA-1be ctccgggctcacctcgct SEQ ID No: 55 HPA-1ba CR agcgaggtgagcccggag SEQ ID No: 56 HPA-1bb CR aggtgagcccggaggcag SEQ ID No: 57 HPA-1bc CR gtgagcccggaggcaggg SEQ ID No: 58 HPA-1bd CR agcccggaggcagggcct SEQ ID No: 59 HPA-1be CR cccggaggcagggcctgta SEQ ID No: 60
Alelo HPA2 a: HPA-2aa ctgacgcccacacccaag SEQ ID No: 61 HPA-2ab ctcctgacgcccacaccc SEQ ID No: 62 HPA-2ac ggctcctgacgcccacac SEQ ID No: 63 HPA-2ad agggctcctgacgcccac SEQ ID No: 64 HPA-2ae ccagggctcctgacgccc SEQ ID No: 65 HPA-2aa CR cttgggtgtgggcgtcag SEQ ID No: 66 HPA-2ab CR gggtgtgggcgtcaggag SEQ ID No: 67 HPA-2ac CR gtgtgggcgtcaggagcc SEQ ID No: 68 HPA-2ad CR gtgggcgtcaggagccct SEQ ID No: 69 HPA-2ae CR gggcgtcaggagccctgg SEQ ID No: 70
Alelo HPA2 b: HPA-2ba cctgatgcccacacccaag SEQ ID No: 71 HPA-2bb ctcctgatgcccacaccca SEQ ID No: 72 HPA-2bc ggctcctgatgcccacacc SEQ ID No: 73 HPA-2bd agggctcctgatgcccaca SEQ ID No: 74 HPA-2be ccagggctcctgatgccc SEQ ID No: 75 HPA-2ba CR cttgggtgtgggcatcagg SEQ ID No: 76 HPA-2bb CR tgggtgtgggcatcaggag SEQ ID No: 77 HPA-2bc CR ggtgtgggcatcaggagcc SEQ ID No: 78 HPA-2bd CR tgtgggcatcaggagccct SEQ ID No: 79 HPA-2be CR gggcatcaggagccctgg SEQ ID No: 80
Alelo HPA3 a: HPA-3 aa ccatccccagcccctccc SEQ ID No: 81 HPA-3ab gcccatccccagcccctc SEQ ID No: 82 HPA-3ac ctgcccatccccagcccc SEQ ID No: 83 HPA-3ad1 gctgcccatccccagccc SEQ ID No: 84 HPA-3 ad ggctgcccatccccagcc SEQ ID No: 85 HPA-3ad2 gggctgcccatccccagc SEQ ID No: 86 HPA-3ae ggggctgcccatcccca SEQ ID No: 87 HPA-3 aa CR gggaggggctggggatgg SEQ ID No: 88 HPA-3ab CR gaggggctggggatgggc SEQ ID No: 89 HPA-3ac CR ggggctggggatgggcag SEQ ID No: 90 HPA-3ad1 CR gggctggggatgggcagc SEQ ID No: 91 HPA-3ad CR ggctggggatgggcagcc SEQ ID No: 92 HPA-3ad2 CR gctggggatgggcagccc SEQ ID No: 93 HPA-3ae CR tggggatgggcagcccc SEQ ID No: 94
Alelo HPA3 b: HPA-3ba ccagccccagcccctcc SEQ ID No: 95 HPA-3bb gcccagccccagcccct SEQ ID No: 96 HPA-3bc ctgcccagccccagccc SEQ ID No: 97 HPA-3bd1 gctgcccagccccagcc SEQ ID No: 98 HPA-3bd ggctgcccagccccagc SEQ ID No: 99 HPA-3bd2 gggctgcccagccccag SEQ ID No: 100 HPA-3be ggggctgcccagcccca SEQ ID No: 101 HPA-3ba CR ggaggggctggggctgg SEQ ID No: 102 HPA-3bb CR aggggctggggctgggc SEQ ID No: 103 HPA-3bc CR gggctggggctgggcag SEQ ID No: 104 HPA-3bd1 CR ggctggggctgggcagc SEQ ID No: 105 HPA-3bd CR gctggggctgggcagcc SEQ ID No: 106 HPA-3bd2 CR ctggggctgggcagccc SEQ ID No: 107 HPA-3be CR tggggctgggcagcccc SEQ ID No: 108
Alelo HPA4 a: HPA-4aa gccacccagatgcgaaag SEQ ID No: 109 HPA-4ab cacccagatgcgaaagct SEQ ID No: 110
HPA-4ac cccagatgcgaaagctca SEQ ID No: 111 HPA-4ad cagatgcgaaagctcacc SEQ ID No: 112 HPA-4ae gatgcgaaagctcaccag SEQ ID No: 113 HPA-4aa CR ctggtgagctttcgcatc SEQ ID No: 114 HPA-4ab CR ggtgagctttcgcatctg SEQ ID No: 115 HPA-4ac CR tgagctttcgcatctggg SEQ ID No: 116 HPA-4ad CR agctttcgcatctgggtg SEQ ID No: 117 HPA-4ae CR ctttcgcatctgggtggc SEQ ID No: 118 Alelo HPA4 b: HPA-4ba gccacccagatgcaaaag SEQ ID No: 119 HPA-4bb ccacccagatgcaaaagct SEQ ID No: 120 HPA-4bc acccagatgcaaaagctcac SEQ ID No: 121 HPA-4bd cagatgcaaaagctcacca SEQ ID No: 122 HPA-4be gatgcaaaagctcaccagtaa SEQ ID No: 123 HPA-4ba CR ttactggtgagcttttgcatc SEQ ID No: 124 HPA-4bb CR tggtgagcttttgcatctg SEQ ID No: 125 HPA-4bc CR gtgagcttttgcatctgggt SEQ ID No: 126 HPA-4bd CR agcttttgcatctgggtgg SEQ ID No: 127 HPA-4be CR cttttgcatctgggtggc SEQ ID No: 128 Alelo HPA5 a: HPA-5aa gagtctacctgtttactatcaaagagg SEQ ID No: 129 HPA-5ab agtctacctgtttactatcaaagaggta SEQ ID No: 130 HPA-5ac gtctacctgtttactatcaaagaggtaa SEQ ID No: 131 HPA-5ad ctacctgtttactatcaaagaggtaaaa SEQ ID No: 132 HPA-5ae acctgtttactatcaaagaggtaaaaa SEQ ID No: 133 HPA-5aa CR tttttacctctttgatagtaaacaggt SEQ ID No: 134 HPA-5ab CR ttttacctctttgatagtaaacaggtag SEQ ID No: 135 HPA-5ac CR ttacctctttgatagtaaacaggtagac SEQ ID No: 136 HPA-5ad CR tacctctttgatagtaaacaggtagact SEQ ID No: 137 HPA-5ae CR cctctttgatagtaaacaggtagactc SEQ ID No: 138 Alelo HPA5 b: HPA-5ba gagtctacctgtttactatcaaaaagg SEQ ID No: 139 BPA-5bb agtctacctgtttactatcaaaaaggta SEQ ID No: 140 HPA-5bc gtctacctgtttactatcaaaaaggtaa SEQ ID No: 141 HPA-5ad ctacctgtttactatcaaaaaggtaaaa SEQ ID No: 142 HPA-5be acctgtttactatcaaaaaggtaaaaa SEQ ID No: 143 HPA-5ba CR tttttacctttttgatagtaaacaggt SEQ ID No: 144
HPA-5bb CR ttttacctttttgatagtaaacaggtag SEQ ID No: 145 HPA-5bc CR ttacctttttgatagtaaacaggtagac SEQ ID No: 146 HPA-5bd CR tacctttttgatagtaaacaggtagact SEQ ID No: 147 HPA-5be CR cctttttgatagtaaacaggtagactc SEQ ID No: 148
Alelo HPA15 a: Gov-aa ttattatcttgacttcagttacaggattt SEQ ID No: 149 Gov-ab tcttgacttcagttacaggatttacc SEQ ID No: 150 Gov-ac tgacttcagttacaggatttaccaa SEQ ID No: 151 Gov-ad cttcagttacaggatttaccaagaat SEQ ID No: 152 Gov-ae cagttacaggatttaccaagaatttg SEQ ID No: 153 Gov-aa CR caaattcttggtaaatcctgtaactg SEQ ID No: 154 Gov-ab CR attcttggtaaatcctgtaactgaag SEQ ID No: 155 Gov-ac CR tggtaaatcctgtaactgaagtcaa SEQ ID No: 156 Gov-ad CR ggtaaatcctgtaactgaagtcaaga SEQ ID No: 157 Gov-ae CR aaatcctgtaactgaagtcaagataataa SEQ ID No: 158
Alelo HPA15 b: Gov-ba tatcttgacttcagttccaggatt SEQ ID No: 159 Gov-bb cttgacttcagttccaggatttac SEQ ID No: 160 Gov-bc gacttcagttccaggatttacca SEQ ID No: 161 Gov-bd ttcagttccaggatttaccaag SEQ ID No: 162 Gov-be cagttccaggatttaccaagaatt SEQ ID No: 163 Gov-ba CR aattcttggtaaatcctggaactg SEQ ID No: 164 Gov-bb CR cttggtaaatcctggaactgaa SEQ ID No: 165 Gov-bc CR ggtaaatcctggaactgaagtca SEQ ID No: 166 Gov-bd CR gtaaatcctggaactgaagtcaag SEQ ID No: 167 Gov-be CR aatcctggaactgaagtcaagata SEQ ID No: 168
Alelo Colton a: Co.a.1 aacaaccagacggcggt SEQ ID No: 191 Co.a.2 accagacggcggtccag SEQ ID No: 192 Co.a.3 cagacggcggtccagga SEQ ID No: 193 Co.a.4 gacggcggtccaggacaa SEQ ID No: 194 Co.a.5 cggcggtccaggacaac SEQ ID No: 195 Co.a.1.cr gttgtcctggaccgccgt SEQ ID No: 196 Co.a.2.cr tcctggaccgccgtctg SEQ ID No: 197 Co.a.3.cr ctggaccgccgtctggt SEQ ID No: 198 Co.a.4.cr ggaccgccgtctggttg SEQ ID No: 199 Co.a.5.cr accgccgtctggttgtt SEQ ID No: 200
Alelo Colton b: Co.b.1 ggaacaaccagacggtggt SEQ ID No: 201 Co.b.2 acaaccagacggtggtccag SEQ ID No: 202 Co.b.3 accagacggtggtccagga SEQ ID No: 203 Co.b.4 gacggtggtccaggacaacg SEQ ID No: 204 Co.b.5 cggtggtccaggacaacg SEQ ID No: 205 Co.b.1.cr cgttgtcctggaccaccgt SEQ ID No: 206 Co.b.2.cr ttgtcctggaccaccgtctg SEQ ID No: 207 Co.b.3.cr ctggaccaccgtctggttgt SEQ ID No: 208 Co.b.4.cr ggaccaccgtctggttgttc SEQ ID No: 209 Co.b.5.cr accaccgtctggttgttcc SEQ ID No: 210
Alelo Diego a: Di.a.1 gtgaagtccacgccggc SEQ ID No: 211 Di.a.2 gaagtccacgccggcct SEQ ID No: 212 Di.a.3 agtccacgccggcctcc SEQ ID No: 213 Di.a.4 tccacgccggcctccct SEQ ID No: 214 Di.a.5 acgccggcctccctggcc SEQ ID No: 215 Di.a.1.cr gccagggaggccggcgt SEQ ID No: 216 Di.a.2.cr agggaggccggcgtgga SEQ ID No: 217 Di.a.3.cr ggaggccggcgtggact SEQ ID No: 218 Di.a.4.cr aggccggcgtggacttc SEQ ID No: 219 Di.a.5.cr ggccggcgtggacttca SEQ ID No: 220
Alelo Diego b: Di.b.1 ggtgaagtccacgctggc SEQ ID No: 221 Di.b.2 gtgaagtccacgctggcct SEQ ID No: 222 Di.b.3 tgaagtccacgctggcctcc SEQ ID No: 223 Di.b.4 gaagtccacgctggcctccct SEQ ID No: 224 Di.b.5 acgctggcctccctggccc SEQ ID No: 225 Di.b.1.cr ggccagggaggccagcgt SEQ ID No: 226 Di.b.2.cr cagggaggccagcgtgga SEQ ID No: 227 Di.b.3.cr agggaggccagcgtggact SEQ ID No: 228 Di.b.4.cr ggaggccagcgtggacttc SEQ ID No: 229 Di.b.5.cr ggccagcgtggacttcacc SEQ ID No: 230
Alelo Diego Wr a: Wr.a.1 tgggcttgcgttccaagt SEQ ID No: 231 Wr.a.2 ggcttgcgttccaagtttc SEQ ID No: 232 Wr.a.3 ttgcgttccaagtttccca SEQ ID No: 233 Wr.a.4 cgttccaagtttcccatct SEQ ID No: 234 Wr.a.5 tccaagtttcccatctgga SEQ ID No: 235 Wr.a.1 CR tccagatgggaaacttgga SEQ ID No: 236 Wr.a.2 CR agatgggaaacttggaacg SEQ ID No: 237 Wr.a.3 CR tgggaaacttggaacgcaa SEQ ID No: 238 Wr.a.4 CR gaaacttggaacgcaagcc SEQ ID No: 239 Wr.a.5 CR acttggaacgcaagccca SEQ ID No: 240
Alelo Diego Wr b: Wr.b.1 gggcttgcgttccgagtt SEQ ID No: 241 Wr.b.2 gcttgcgttccgagtttc SEQ ID No: 242 Wr.b.3 ttgcgttccgagtttccc SEQ ID No: 243 Wr.b.4 cgttccgagtttcccatc SEQ ID No: 244 Wr.b.5 tccgagtttcccatctgg SEQ ID No: 245 Wr.b.1 CR ccagatgggaaactcgga SEQ ID No: 246 Wr.b.2 CR gatgggaaactcggaacg SEQ ID No: 247 Wr.b.3 CR gggaaactcggaacgcaa SEQ ID No: 248 Wr.b.4 CR gaaactcggaacgcaagc SEQ ID No: 249 Wr.b.5 CR aactcggaacgcaagccc SEQ ID No: 250
Alelo Dombrock a: Do.a.1 taccacccaagaggaaact SEQ ID No: 251 Do.a.2 ccacccaagaggaaactgg SEQ ID No: 252 Do.a.3 acccaagaggaaactggttg SEQ ID No: 253 Do.a.4 caagaggaaactggttgca SEQ ID No: 254 Do.a.5 aggaaactggttgcagttga SEQ ID No: 255 Do a 6 cr ctcaactgcaaccagtttcc SEQ ID No: 256 Do a 7 cr caactgcaaccagtttcctc SEQ ID No: 257 Do a 8 cr tgcaaccagtttcctcttgg SEQ ID No: 258 Do a 9 cr accagtttcctcttgggtgg SEQ ID No: 259 Do a 10 cr cagtttcctcttgggtggta SEQ ID No: 260
Alelo Dombrock b: Do.b.1 taccacccaagaggagact SEQ ID No: 261 Do.b.2 ccacccaagaggagactgg SEQ ID No: 262 Do.b.3 acccaagaggagactggttg SEQ ID No: 263 Do.b.4 caagaggagactggttgca SEQ ID No: 264 Do.b.5 aggagactggttgcagttga SEQ ID No: 265 Do b 6 cr ctcaactgcaaccagtctcc SEQ ID No: 266 Do b 7 cr caactgcaaccagtctcctc SEQ ID No: 267
Do b 8 cr tgcaaccagtctcctcttgg SEQ ID No: 268 Do b 9 cr accagtctcctcttgggtgg SEQ ID No: 269 Do b 10 cr cagtctcctcttgggtggt SEQ ID No: 270 Alelo Dombrock-Joseph(a) positivo: Jo.a.pos.1 ccccagaacatgactaccac SEQ ID No: 271 Jo.a.pos.2 ccagaacatgactaccacaca SEQ ID No: 272 Jo.a.pos.3 agaacatgactaccacacacgc SEQ ID No: 273 Jo.a.pos.4 catgactaccacacacgctgt SEQ ID No: 274 Jo.a.pos.5 actaccacacacgctgtgg SEQ ID No: 275 Jo.a.pos.1.cr gccacagcgtgtgtggtagt SEQ ID No: 276 Jo.a.pos.2.cr acagcgtgtgtggtagtcatg SEQ ID No: 277 Jo.a.pos.3.cr agcgtgtgtggtagtcatgtt SEQ ID No: 278 Jo.a.pos.4.cr cgtgtgtggtagtcatgttctg SEQ ID No: 279 Jo.a.pos.5.cr gtggtagtcatgttctgggg SEQ ID No: 280 Alelo Dombrock-Joseph(a) negativo: Jo.a.neg.1 ctaccccagaacatgactatcac SEQ ID No: 281 Jo.a.neg.2 cccagaacatgactatcacaca SEQ ID No: 282 Jo.a.neg.3 cagaacatgactatcacacacgc SEQ ID No: 283 Jo.a.neg.4 catgactatcacacacgctgtg SEQ ID No: 284 Jo.a.neg.5 actatcacacacgctgtggc SEQ ID No: 285 Jo.a.neg.1.cr aatagccacagcgtgtgtgatagt SEQ ID No: 286 Jo.a.neg.2.cr cacagcgtgtgtgatagtcatg SEQ ID No: 287 Jo.a.neg.3.cr cagcgtgtgtgatagtcatgtt SEQ ID No: 288 Jo.a.neg.4.cr cgtgtgtgatagtcatgttctgg SEQ ID No: 289 Jo.a.neg.5.cr gtgatagtcatgttctggggtag SEQ ID No: 290 Alelo Duffy a: Fy.A.1 ccagatggagactatggtgcc SEQ ID No: 291 Fy.A.2 atggagactatggtgccaac SEQ ID No: 292 Fy.A.3 ggagactatggtgccaacctg SEQ ID No: 293 Fy.A.4 gactatggtgccaacctgga SEQ ID No: 294 Fy.A.5 tatggtgccaacctggaag SEQ ID No: 295 Fy.A.1.cr cttccaggttggcaccata SEQ ID No: 296 Fy.A.2.cr tccaggttggcaccatagtc SEQ ID No: 297 Fy.A.3.cr aggttggcaccatagtctcc SEQ ID No: 298 Fy.A.4.cr gttggcaccatagtctccat SEQ ID No: 299 Fy.A.5.cr gcaccatagtctccatctgg SEQ ID No: 300
Alelo Duffy b: Fy.B.1 cccagatggagactatgatgcc SEQ ID No: 301 Fy.B.2 gatggagactatgatgccaac SEQ ID No: 302 Fy.B.3 tggagactatgatgccaacctg SEQ ID No: 303 Fy.B.4 gactatgatgccaacctggaa SEQ ID No: 304 Fy.B.5 tatgatgccaacctggaagc SEQ ID No: 305 Fy.B.1.cr gcttccaggttggcatcata SEQ ID No: 306 Fy.B.2.cr ttccaggttggcatcatagtc SEQ ID No: 307 Fy.B.3.cr caggttggcatcatagtctcc SEQ ID No: 308 Fy.B.4.cr gttggcatcatagtctccatc SEQ ID No: 309 Fy.B.5.cr gcatcatagtctccatctggg SEQ ID No: 310 Alelo Duffy GATAbox normal: Fy.GATA.normal.1 agtccttggctcttatcttg SEQ ID No: 311 Fy.GATA.normal.2 agtccttggctcttatcttgga SEQ ID No: 312 Fy.GATA.normal.3 cttggctcttatcttggaagc SEQ ID No: 313 Fy.GATA.normal.4 gctcttatcttggaagcacagg SEQ ID No: 314 Fy.GATA.normal.5 cttatcttggaagcacaggcgc SEQ ID No: 315 Fy.GATA.normal.1.cr gcctgtgcttccaagataag SEQ ID No: 316 Fy.GATA.normal.2.cr tgtgcttccaagataagagc SEQ ID No: 317 Fy.GATA.normal.3.cr tgcttccaagataagagcca SEQ ID No: 318 Fy.GATA.normal.4.cr ttccaagataagagccaagga SEQ ID No: 319 Fy.GATA.normal.5.cr caagataagagccaaggact SEQ ID No: 320 Alelo de mutación Duffy GATAbox: Fy.GATA.mut.1 tccttggctcttaccttg SEQ ID No: 321 Fy.GATA.mut.2 tccttggctcttaccttgga SEQ ID No: 322 Fy.GATA.mut.3 tggctcttaccttggaagc SEQ ID No: 323 Fy.GATA.mut.4 gctcttaccttggaagcacag SEQ ID No: 324 Fy.GATA.mut.5 cttaccttggaagcacaggcg SEQ ID No: 325 Fy.GATA.mut.1.cr cctgtgcttccaaggtaag SEQ ID No: 326 Fy.GATA.mut.2.cr gtgcttccaaggtaagagc SEQ ID No: 327 Fy.GATA.mut.3.cr gcttccaaggtaagagcca SEQ ID No: 328 Fy.GATA.mut.4.cr ttccaaggtaagagccaagg SEQ ID No: 329
Fy.GATA.mut.5.cr
Alelo normal Duffy Fyx: Fy.X.(b).1 Fy.X.(b).2 Fy.X.(b).3 Fy.X.(b).4 Fy.X.(b).5 Fy.X.(b).1.cr Fy.X.(b).2.cr Fy.X.(b).3.cr Fy.X.(b).4.cr Fy.X.(b).5.cr
caaggtaagagccaagga
ttttcagacctctcttccgct agacctctcttccgctggc ctctcttccgctggcagc ctcttccgctggcagctc cttccgctggcagctctg cagagctgccagcggaa agctgccagcggaagag ctgccagcggaagagagg gccagcggaagagaggtc cagcggaagagaggtctg SEQ ID No: 330
SEQ ID No: 331 SEQ ID No: 332 SEQ ID No: 333 SEQ ID No: 334 SEQ ID No: 335 SEQ ID No: 336 SEQ ID No: 337 SEQ ID No: 338 SEQ ID No: 339 SEQ ID No: 340
Alelo de mutación Duffy Fyx:
Fy.X.1 Fy.X.2 Fy.X.3 Fy.X.4 Fy.X.5 Fy.X.1.cr Fy.X.2.cr Fy.X.3.cr Fy.X.4.cr Fy.X.5.cr
Alelo Kidd a: Jk.a.1 Jk.a.2 Jk.a.3 Jk.a.4 Jk.a.5 Jk.a.1.cr Jk.a.2.cr Jk.a.3.cr Jk.a.4.cr Jk.a.5.cr
Alelo Kidd b: Jk.b.1 Jk.b.2
gcttttcagacctctcttctgct SEQ ID No: 341
tcagacctctcttctgctggc acctctcttctgctggcagc ctcttctgctggcagctctg cttctgctggcagctctgc gcagagctgccagcagaa gagctgccagcagaagagag agctgccagcagaagagagg gccagcagaagagaggtctg cagcagaagagaggtctgaaa
cagccccatttgaggaca gccccatttgaggacatcta SEQ ID No: 342 SEQ ID No: 343 SEQ ID No: 344 SEQ ID No: 345 SEQ ID No: 346 SEQ ID No: 347 SEQ ID No: 348 SEQ ID No: 349 SEQ ID No: 350
SEQ ID No: 351 SEQ ID No: 352
ccatttgaggacatctactttg SEQ ID No: 353 atttgaggacatctactttgga SEQ ID No: 354 gaggacatctactttggactct SEQ ID No: 355 cagagtccaaagtagatgtcctc SEQ ID No: 356 agtccaaagtagatgtcctcaaa SEQ ID No: 357 aaagtagatgtcctcaaatggg SEQ ID No: 358 tagatgtcctcaaatggggc SEQ ID No: 359 atgtcctcaaatggggctg SEQ ID No: 360
tcagccccatttgagaaca SEQ ID No: 361 gccccatttgagaacatcta SEQ ID No: 362 Jk.b.3 cccatttgagaacatctactttg SEQ ID No: 363 Jk.b.4 atttgagaacatctactttggac SEQ ID No: 364 Jk.b.5 gagaacatctactttggactctg SEQ ID No: 365 Jk.b.1.cr ccagagtccaaagtagatgttctcSEQ ID No: 366 Jk.b.2.cr agagtccaaagtagatgttctcaaa SEQ ID No:
367 Jk.b.3.cr ccaaagtagatgttctcaaatgg SEQ ID No: 368 Jk.b.4.cr agtagatgttctcaaatggggc SEQ ID No: 369 Jk.b.5.cr atgttctcaaatggggctga SEQ ID No: 370 Alelo Kell K1: KEL.1.1 tccttaaactttaaccgaatgct SEQ ID No: 371 KEL.1.2 ttaaactttaaccgaatgctgaga SEQ ID No:
372 KEL.1.3 aactttaaccgaatgctgagactt SEQ ID No:
373 KEL.1.4 aaccgaatgctgagacttctg SEQ ID No: 374 KEL.1.5 cgaatgctgagacttctgatgag SEQ ID No: 375 KEL 1.6 CR actcatcagaagtctcagcattc SEQ ID No: 376 KEL 1.7 CR tcagaagtctcagcattcggt SEQ ID No: 377 KEL 1.8 CR aagtctcagcattcggttaaag SEQ ID No: 378 KEL 1.9 CR tctcagcattcggttaaagtttaa SEQ ID No:
379 KEL 1.10 CR agcattcggttaaagtttaagga SEQ ID No: 380 Alelo Kell K2: KEL.2.1 ccttaaactttaaccgaacgct SEQ ID No: 381 KEL.2.2 aactttaaccgaacgctgaga SEQ ID No: 382 KEL.2.3 ctttaaccgaacgctgagactt SEQ ID No: 383 KEL.2.4 aaccgaacgctgagacttct SEQ ID No: 384 KEL.2.5 cgaacgctgagacttctgatg SEQ ID No: 385 KEL 2.6 CR tcatcagaagtctcagcgttc SEQ ID No: 386 KEL 2.7 CR cagaagtctcagcgttcggt SEQ ID No: 387 KEL 2.8 CR agtctcagcgttcggttaaag SEQ ID No: 388 KEL 2.9 CR tctcagcgttcggttaaagtt SEQ ID No: 389 KEL 2.10 CR agcgttcggttaaagtttaagg SEQ ID No: 390 Alelo Kell K3: KEL.3.1 aatctccatcacttcatggct SEQ ID No: 391 KEL.3.2 tccatcacttcatggctgtt SEQ ID No: 392
KEL.3.3 atcacttcatggctgttcca SEQ ID No: 393 KEL.3.4 acttcatggctgttccagtt SEQ ID No: 394 KEL.3.5 tcatggctgttccagtttc SEQ ID No: 395 KEL.3.1.cr agaaactggaacagccatgaa SEQ ID No: 396 KEL.3.2.cr aactggaacagccatgaagtg SEQ ID No: 397 KEL.3.3.cr tggaacagccatgaagtgatg SEQ ID No: 398 KEL.3.4.cr acagccatgaagtgatggag SEQ ID No: 399 KEL.3.5.cr gccatgaagtgatggagatt SEQ ID No: 400
Alelo Kell K4: KEL.4.1 tctccatcacttcacggct SEQ ID No: 401 KEL.4.2 ccatcacttcacggctgtt SEQ ID No: 402 KEL.4.3 cacttcacggctgttcca SEQ ID No: 403 KEL.4.4 acttcacggctgttccag SEQ ID No: 404 KEL.4.5 tcacggctgttccagttt SEQ ID No: 405 KEL.4.1.cr aaactggaacagccgtgaa SEQ ID No: 406 KEL.4.2.cr ctggaacagccgtgaagtg SEQ ID No: 407 KEL.4.3.cr ggaacagccgtgaagtgatg SEQ ID No: 408 KEL.4.4.cr acagccgtgaagtgatgg SEQ ID No: 409 KEL.4.5.cr gccgtgaagtgatggaga SEQ ID No: 410
Alelo Kell K6: KEL.6.1 tactgcctgggggctgccccgcc SEQ ID No: 411 KEL.6.2 tgggggctgccccgcctgt SEQ ID No: 412 KEL.6.3 gctgccccgcctgtgac SEQ ID No: 413 KEL.6.4 ctgccccgcctgtgacaa SEQ ID No: 414 KEL.6.5 gccccgcctgtgacaac SEQ ID No: 415 KEL.6.1.cr gttgtcacaggcggggc SEQ ID No: 416 KEL.6.2.cr ttgtcacaggcggggcag SEQ ID No: 417 KEL.6.3.cr tcacaggcggggcagccc SEQ ID No: 418 KEL.6.4.cr acaggcggggcagccccca SEQ ID No: 419 KEL.6.5.cr aggcggggcagccccca SEQ ID No: 420
Alelo Kell K7: KEL.7.1 actgcctgggggctgcctcgcc SEQ ID No: 421 KEL.7.2 cctgggggctgcctcgcctgt SEQ ID No: 422 KEL.7.3 ggctgcctcgcctgtgac SEQ ID No: 423 KEL.7.4 ctgcctcgcctgtgacaacc SEQ ID No: 424 KEL.7.5 gcctcgcctgtgacaacc SEQ ID No: 425 KEL.7.1.cr ggttgtcacaggcgaggc SEQ ID No: 426 KEL.7.2.cr ggttgtcacaggcgaggcag SEQ ID No: 427 KEL.7.3.cr ttgtcacaggcgaggcagccc SEQ ID No: 428 KEL.7.4.cr acaggcgaggcagcccccagg SEQ ID No: 429 KEL.7.5.cr aggcgaggcagcccccagg SEQ ID No: 430
Alelo Lutheran a: Lu.a.1 ggagctcgcccccgcct SEQ ID No: 431 Lu.a.2 gctcgcccccgcctagc SEQ ID No: 432 Lu.a.3 cgcccccgcctagcctc SEQ ID No: 433 Lu.a.4 cccccgcctagcctcgg SEQ ID No: 434 Lu.a.5 cccgcctagcctcggct SEQ ID No: 435 Lu.a.1 CR agccgaggctaggcggg SEQ ID No: 436 Lu.a.2 CR ccgaggctaggcggggg SEQ ID No: 437 Lu.a.3 CR gaggctaggcgggggcg SEQ ID No: 438 Lu.a.4 CR gctaggcgggggcgagc SEQ ID No: 439 Lu.a.5 CR aggcgggggcgagctcc SEQ ID No: 440
Alelo Lutheran b: Lu.b.1 gggagctcgcccccacct SEQ ID No: 441 Lu.b.2 gagctcgcccccacctagc SEQ ID No: 442 Lu.b.3 ctcgcccccacctagcctc SEQ ID No: 443 Lu.b.4 cccccacctagcctcggct SEQ ID No: 444 Lu.b.5 cccacctagcctcggctga SEQ ID No: 445 Lu.b.1 CR tcagccgaggctaggtggg SEQ ID No: 446 Lu.b.2 CR agccgaggctaggtggggg SEQ ID No: 447 Lu.b.3 CR gaggctaggtgggggcgag SEQ ID No: 448 Lu.b.4 CR gctaggtgggggcgagctc SEQ ID No: 449 Lu.b.5 CR aggtgggggcgagctccc SEQ ID No: 450
Alelo MNS M:
M.1 gtgagcatatcagcatcaag SEQ ID No: 451
M.2 atcagcatcaagtaccactgg SEQ ID No: 452
M.3 catcaagtaccactggtgtg SEQ ID No: 453
M.4 taccactggtgtggcaatgc SEQ ID No: 454
M.5
ctggtgtggcaatgcaca SEQ ID No: 455 M.1.cr tgtgcattgccacaccagt SEQ ID No: 456 M.2.cr gcattgccacaccagtggta SEQ ID No: 457 M.3.cr ccacaccagtggtacttgatg SEQ ID No: 458 M.4.cr accagtggtacttgatgct SEQ ID No: 459 M.5.cr cttgatgctgatatgctcac SEQ ID No: 460 Alelo MNS N:
N.1 tgtgagcatatcagcattaag SEQ ID No: 461
N.2 atcagcattaagtaccactgagg SEQ ID No: 462
N.3 cattaagtaccactgaggtgg SEQ ID No: 463
N.4 accactgaggtggcaatgc SEQ ID No: 464
N.5 ctgaggtggcaatgcacact SEQ ID No: 465 N.1.cr gtgtgcattgccacctcagt SEQ ID No: 466 N.2.cr gcattgccacctcagtggta SEQ ID No: 467 N.3.cr ccacctcagtggtacttaatgc SEQ ID No: 468 N.4.cr cctcagtggtacttaatgct SEQ ID No: 469 N.5.cr cttaatgctgatatgctcaca SEQ ID No: 470 Alelo MNS S: big.S.1 tttgctttataggagaaatggga SEQ ID No: 471 big.S.2 ctttataggagaaatgggaca SEQ ID No: 472 big.S.3 ttataggagaaatgggacaacttg SEQ ID No:
big.S.4 gagaaatgggacaacttgtcc SEQ ID No: 474 big.S.5 aaatgggacaacttgtccatc SEQ ID No: 475 big.S.1.cr gatggacaagttgtcccattt SEQ ID No: 476 big.S.2.cr gacaagttgtcccatttctcc SEQ ID No: 477 big.S.3.cr aagttgtcccatttctcctata SEQ ID No: 478 big.S.4.cr tgtcccatttctcctataaagca SEQ ID No: 479 big.S.5.cr cccatttctcctataaagcaaaa SEQ ID No: 480
Alelo MNS s: little.s.1 tgctttataggagaaacggga SEQ ID No: 481 little.s.2 tttataggagaaacgggaca SEQ ID No: 482 little.s.3 ggagaaacgggacaacttg SEQ ID No: 483 little.s.4 gagaaacgggacaacttgtc SEQ ID No: 484 little.s.5 aaacgggacaacttgtccat SEQ ID No: 485 little.s.1.cr tggacaagttgtcccgttt SEQ ID No: 486 little.s.2.cr acaagttgtcccgtttctcc SEQ ID No: 487 little.s.3.cr agttgtcccgtttctcctata SEQ ID No: 488 little.s.4.cr tgtcccgtttctcctataaagc SEQ ID No: 489 little.s.5.cr ccgtttctcctataaagca SEQ ID No: 490
Alelo MNS U-positivo: U.pos.1 ttgctgctctctttagctcc SEQ ID No: 491 U.pos.2 ctctctttagctcctgtagtgat SEQ ID No: 492
U.pos.3 U.pos.4 494 U.pos.5 U.pos.1.cr U.pos.2.cr 497 U.pos.3.cr 498 U.pos.4.cr U.pos.5.cr Alelo MNS U-negativo: U.neg.1 U.neg.2 502 U.neg.3 503 U.neg.4 504 U.neg.5 U.neg.1.cr U.neg.2.cr 507 U.neg.3.cr 508 U.neg.4.cr U.neg.5.cr 510
Alelo de Rhesus C(307T):
Rh.big.C.1 Rh.big.C.2 Rh.big.C.3 Rh.big.C.4 Rh.big.C.5 Rh.big.C.1.cr Rh.big.C.2.cr Rh.big.C.3.cr
agctcctgtagtgataatactca SEQ ID No: 493 gtagtgataatactcattatttttg SEQ ID No: taatactcattatttttggggtg SEQ ID No: 495 caccccaaaaataatgagtatta SEQ ID No: 496 caaaaataatgagtattatcactaca SEQ ID No: agtattatcactacaggagctaaa SEQ ID No: atcactacaggagctaaagag SEQ ID No: 499 gagctaaagagagcagcaaa SEQ ID No: 500 ttttgctgctctctttatctcc SEQ ID No: 501 gctctctttatctcctgtagagat SEQ ID No: tatctcctgtagagataacactca SEQ ID No: gtagagataacactcattattttt SEQ ID No: taacactcattatttttggggt SEQ ID No: 505 accccaaaaataatgagtgtta SEQ ID No: 506 aaaaataatgagtgttatctctaca SEQ ID No: agtgttatctctacaggagataaa SEQ ID No: atctctacaggagataaagagag SEQ ID No: 509 gagataaagagagcagcaaaatta SEQ ID No:
tgagccagttcccttctgg gagccagttcccttctgg ctgagccagttcccttctg ccttctgggaaggtggtc ccttctgggaaggtggtca tgaccaccttcccagaagg gaccaccttcccagaagg ccagaagggaactggctc SEQ ID No: 511 SEQ ID No: 512 SEQ ID No: 513 SEQ ID No: 514 SEQ ID No: 515 SEQ ID No: 516 SEQ ID No: 517 SEQ ID No: 518
Rh.big.C.4.cr ccagaagggaactggctca Rh.big.C.5.cr cagaagggaactggctcag Alelo de Rhesus c(307C):
Rh.little.c.1 Rh.little.c.2 Rh.little.c.3 Rh.little.c.4 Rh.little.c.5 Rh.little.c.1.cr Rh.little.c.2.cr Rh.little.c.3.cr Rh.little.c.4.cr Rh.little.c.5.cr
gagccagttccctcctgg agccagttccctcctgg tgagccagttccctcctg cctcctgggaaggtggt cctcctgggaaggtggtc gaccaccttcccaggagg accaccttcccaggagg ccaggagggaactggct ccaggagggaactggctc caggagggaactggctca
SEQ ID No: 519 SEQ ID No: 520
SEQ ID No: 521 SEQ ID No: 522 SEQ ID No: 523 SEQ ID No: 524 SEQ ID No: 525 SEQ ID No: 526 SEQ ID No: 527 SEQ ID No: 528 SEQ ID No: 529 SEQ ID No: 530
Alelo de inserto específico de intrón2 BigC de Rhesus:
RhC.intron.2.1 RhC.intron.2.2 RhC.intron.2.3 RhC.intron.2.4 RhC.intron.2.5
agggtgccctttgtcacttc SEQ ID No: 531 gccctttgtcacttcccagt SEQ ID No: 532 cctttgtcacttcccagtgg SEQ ID No: 533 ttgtcacttcccagtggtacaa SEQ ID No: 534 tcacttcccagtggtacaatca SEQ ID No: 535
RhC.intron.2.1.cr gaagtgacaaagggcaccct SEQ ID No: 536 RhC.intron.2.2.cr actgggaagtgacaaagggc SEQ ID No: 537 RhC.intron.2.3.cr ccactgggaagtgacaaagg SEQ ID No: 538 RhC.intron.2.4.cr tgtaccactgggaagtgacaaa SEQ ID No: 539 RhC.intron.2.5.cr tgattgtaccactgggaagtga SEQ ID No: 540
Alelo de Rhesus E: Rh.big.E.1 Rh.big.E.2 Rh.big.E.3 Rh.big.E.4 Rh.big.E.5 Rh.big.E.1.cr Rh.big.E.2.cr Rh.big.E.3.cr Rh.big.E.4.cr Rh.big.E.5.cr
Alelo de Rhesus e: Rh.little.e.1
gccaagtgtcaactctcctct SEQ ID No: 541 caagtgtcaactctcctctgct SEQ ID No: 542 gtgtcaactctcctctgctgag SEQ ID No: 543 caactctcctctgctgagaagtc SEQ ID No: 544 tctcctctgctgagaagtcc SEQ ID No: 545 ggacttctcagcagaggagag SEQ ID No: 546 cttctcagcagaggagagttga SEQ ID No: 547 ctcagcagaggagagttgacac SEQ ID No: 548 gcagaggagagttgacacttg SEQ ID No: 549 gaggagagttgacacttggc SEQ ID No: 550
gccaagtgtcaactctgctct SEQ ID No: 551 Rh.little.e.2 Rh.little.e.3 Rh.little.e.4 Rh.little.e.5 Rh.little.e.1.cr Rh.little.e.2.cr Rh.little.e.3.cr Rh.little.e.4.cr Rh.little.e.5.cr
Alelo de Rhesus RHD: RhD 1 RhD 2 RhD 3 RhD 4 RhD 5 RhD cr 1 RhD cr 2 RhD cr 3 RhD cr 4 RhD cr 5
Alelo r's de Rhesus: rs.T.l rs.T.2 rs.T.3 rs.T.4 rs.T.5 rs.T.l.cr rs.T.2.cr rs.T.3.cr rs.T.4.cr rs.T.5.cr
Alelo DVI de Rhesus: Rh.DVI.l Rh.DVI.2 Rh.DVI.3 Rh.DVI.4 Rh.DVI.5
- 83
caagtgtcaactctgctctgct SEQ ID No: 552
tgtcaactctgctctgctgag caactctgctctgctgagaag tctgctctgctgagaagtcc ggacttctcagcagagcagag ttctcagcagagcagagttga tcagcagagcagagttgacac gcagagcagagttgacacttg gagcagagttgacacttggc
ttccccacagctccatcat acagctccatcatgggctac tccatcatgggctacaacttc tcatgggctacaacttcagct gggctacaacttcagcttgct agcaagctgaagttgtagccc agctgaagttgtagcccatga gaagttgtagcccatgatgg gcccatgatggagctgt tgatggagctgtggggaa
ggaaggtcaacttggtgca ggaaggtcaacttggtgcagt caacttggtgcagttggtg acttggtgcagttggtggt ttggtgcagttggtggtgat catcaccaccaactgcacca caccaccaactgcaccaagt accaactgcaccaagttgac actgcaccaagttgaccttcc tgcaccaagttgaccttcc
atttcaaccctcttggcctt aaccctcttggcctttgttt tcttggcctttgtttccttg ggtatcagcttgagagctcg atcagcttgagagctcggag SEQ ID No: 553 SEQ ID No: 554 SEQ ID No: 555 SEQ ID No: 556 SEQ ID No: 557 SEQ ID No: 558 SEQ ID No: 559 SEQ ID No: 560
SEQ ID No: 561 SEQ ID No: 562 SEQ ID No: 563 SEQ ID No: 564 SEQ ID No: 565 SEQ ID No: 566 SEQ ID No: 567 SEQ ID No: 568 SEQ ID No: 569 SEQ ID No: 570
SEQ ID No: 571 SEQ ID No: 572 SEQ ID No: 573 SEQ ID No: 574 SEQ ID No: 575 SEQ ID No: 576 SEQ ID No: 577 SEQ ID No: 578 SEQ ID No: 579 SEQ ID No: 580
SEQ ID No: 581 SEQ ID No: 582 SEQ ID No: 583 SEQ ID No: 584 SEQ ID No: 585
Rh.DVI.l.cr aaggccaagagggttgaaat SEQ ID No: 586 Rh.DVI.2.cr aaacaaaggccaagagggtt SEQ ID No: 587 Rh.DVI.3.cr caaggaaacaaaggccaaga SEQ ID No: 588 Rh.DVI.4.cr cgagctctcaagctgatacc SEQ ID No: 589 Rh.DVI.5.cr ctccgagctctcaagctgat SEQ ID No: 590 Alelo con mutación del pseudogén RHD de Rhesus: RhD Y 1 tttctttgcagacttaggtgc SEQ ID No: 591 RhD Y 2 ctttgcagacttaggtgcaca SEQ ID No: 592 RhD Y 3 tttgcagacttaggtgcacagt SEQ ID No: 593 RhD Y 4 acttaggtgcacagtgcgg SEQ ID No: 594 RhD Y 5 cttaggtgcacagtgcggt SEQ ID No: 595 RhD Y cr 1 caccgcactgtgcacctaa SEQ ID No: 596 RhD Y cr 2 ccgcactgtgcacctaagtc SEQ ID No: 597 RhD Y cr 3 gcactgtgcacctaagtctgc SEQ ID No: 598 RhD Y cr 4 tgcacctaagtctgcaaaga SEQ ID No: 599 RhD Y cr 5 cacctaagtctgcaaagaaatagcg SEQ ID No: 600 Alelo normal del pseudogén RHD de Rhesus: RhD non Y 1 ctatttctttgcagacttatgtgc SEQ ID No: 601 RhD non Y 2 tctttgcagacttatgtgcaca SEQ ID No: 602 RhD non Y 3 ttgcagacttatgtgcacagtg SEQ ID No: 603 RhD non Y 4 acttatgtgcacagtgcggt SEQ ID No: 604 RhD non Y 5 cttatgtgcacagtgcggtg SEQ ID No: 605 RhD non Y cr 1 acaccgcactgtgcacataa SEQ ID No: 606 RhD non Y cr 2 accgcactgtgcacataagtc SEQ ID No: 607 RhD non Y cr 3 gcactgtgcacataagtctgc SEQ ID No: 608 RhD non Y cr 4 tgtgcacataagtctgcaaag SEQ ID No: 609
RhD non Y cr 5 cacataagtctgcaaagaaatagcg SEQ ID No:
610 Alelo Yt-a: Yt.a.1 gcgggagacttccacgg SEQ ID No: 611 Yt.a.2 gggagacttccacggcct SEQ ID No: 612 Yt.a.3 gagacttccacggcctgc SEQ ID No: 613 Yt.a.4 gacttccacggcctgca SEQ ID No: 614 Yt.a.5 ttccacggcctgcaggta SEQ ID No: 615 Yt.a.1 CR tacctgcaggccgtggaa SEQ ID No: 616 Yt.a.2 CR tgcaggccgtggaagtc SEQ ID No: 617 Yt.a.3 CR gcaggccgtggaagtctc SEQ ID No: 618 Yt.a.4 CR aggccgtggaagtctccc SEQ ID No: 619 Yt.a.5 CR ccgtggaagtctcccgc SEQ ID No: 620 Alelo Yt-b: Yt.b.1 gcgggagacttcaacggc SEQ ID No: 621 Yt.b.2 gggagacttcaacggcctg SEQ ID No: 622 Yt.b.3 gagacttcaacggcctgca SEQ ID No: 623 Yt.b.4 gacttcaacggcctgcag SEQ ID No: 624 Yt.b.5 ttcaacggcctgcaggtaa SEQ ID No: 625 Yt.b.1 CR ttacctgcaggccgttgaa SEQ ID No: 626 Yt.b.2 CR ctgcaggccgttgaagtc SEQ ID No: 627Yt.b.3 CR tgcaggccgttgaagtctc SEQ ID No:
628
Yt.b.4 CR caggccgttgaagtctccc SEQ ID No: 629
Yt.b.5 CR gccgttgaagtctcccgc SEQ ID No: 630
14. Método, según cualquiera de las reivindicaciones 1013, en el que las sondas de oligonucleótidos comprenden en el extremo 5' un enlazador, preferentemente una fracción -(CH2)6-, y un grupo reactivo, preferentemente un grupo amino, para la unión a un soporte de arreglo, preferentemente un portaobjetos recubierto con poli-Llisina.
15. Método, según cualquiera de las reivindicaciones 1014, en el que el arreglo comprende uno o más oligonucleótidos con una secuencia que no aparece en el ADN de dicha especie de mamíferos para permitir la sustracción del fondo, seleccionados preferentemente del grupo que comprende:
a3 cagaccataagcacaggcgt SEQ ID No: 631, a9 gctcgtccacagtgcgttat SEQ ID No: 632, a17 cggcgttcaagcaaaccgaa SEQ ID No: 633, a23 gacctatagctccactcaga SEQ ID No: 634, a27 tagggtactgatgagcactc SEQ ID No: 635, a33 tcagccctatcgcaggatgt SEQ ID No: 169, a35 gagacacttgacagtagcca SEQ ID No: 170, a38 ggcagggcacctcagtttat SEQ ID No: 171, a42 tcaccagccagactgtgtag SEQ ID No: 172, a43 cttcacgcaagttgtccaga SEQ ID No: 173.
16. Método, según cualquiera de las reivindicaciones 1015, en el que el arreglo comprende uno o más controles positivos de hibridación, preferentemente
CS05 gtcctgacttctagctcatg SEQ ID No: 174, y su complemento marcado de forma detectable se añade a los productos de la amplificación por PCR múltiple.
17. Método, según cualquiera de las reivindicaciones 10-16, en el que las sondas, antes de añadir los productos de la amplificación por PCR múltiple, se someten a tratamientos de prehibridación y desnaturalización del ADN.
18. Método, según cualquiera de las reivindicaciones 1017, en el que los productos desnaturalizados de la amplificación por PCR múltiple se aplican en el arreglo de ADN sin purificación previa.
19. Método, según cualquiera de las reivindicaciones 1018, en el que para cada antígeno de células de la sangre la relación de las intensidades de las señales para cada uno de los alelos se utiliza para asignar el genotipo.
20. Kit para la determinación del genotipo de antígenos de células de la sangre mediante un método, según cualquiera de las reivindicaciones 1-19, que comprende un par de cebadores quiméricos específicos de antígeno de células de la sangre para que a cada antígeno de células de la sangre se determine el genotipo y, como mínimo, un cebador universal marcado de forma detectable, preferentemente un par de cebadores universales marcados de forma detectable, tal como se define en cualquiera de las reivindicaciones 1-7.
21. Kit, según la reivindicación 20, que comprende además un arreglo de ADN, tal como se define en cualquiera de las reivindicaciones 10-15.
22. Conjunto de pares de cebadores quiméricos específicos de antígenos de células de la sangre útiles en una PCR mútiple, que comprende, como mínimo, dos, preferentemente substancialmente todos, los pares de cebadores seleccionadas del grupo tal como se define en la reivindicación 7.
23. Conjunto de sondas de oligonucleótidos específicas de alelos de antígenos de células de la sangre útiles para la determinación del genotipo de antígenos de células de la sangre, que comprende, como mínimo, 72 sondas diferentes, preferentemente substancialmente todas las sondas, seleccionadas del grupo tal como se define en la reivindicación 13.
Patentes similares o relacionadas:
SECUENCIAS GENOMICAS DE CEBADORES Y SONDA PARA LA CUANTIFICACION DE ADENOVIRUS PORCINOS (PADV), del 30 de Junio de 2011, de UNIVERSIDAD DE BARCELONA: Secuencias genómicas de cebadores y sonda para la cuantificación de adenovirus porcinos (PAdV).La invención se refiere a un test sensible para la detección de […]
 MÉTODO Y SISTEMA PARA ANALIZAR REACCIONES USANDO UN SISTEMA DE INFORMACIÓN, del 4 de Mayo de 2011, de ABBOTT LABORATORIES: Un método de determinación de si una muestra de test contiene un ácido nucleico objetivo, comprendiendo el método: (a) poner en contacto […]
MÉTODO Y SISTEMA PARA ANALIZAR REACCIONES USANDO UN SISTEMA DE INFORMACIÓN, del 4 de Mayo de 2011, de ABBOTT LABORATORIES: Un método de determinación de si una muestra de test contiene un ácido nucleico objetivo, comprendiendo el método: (a) poner en contacto […]
 APARATO PARA LLEVAR A CABO LA PCR Y MONITORIZACIÓN DE LA REACCIÓN EN TIEMPO REAL DURANTE CICLOS DE TEMPERATURA, del 21 de Marzo de 2011, de UNIVERSITY OF UTAH RESEARCH FOUNDATION: Aparato para llevar a cabo la PCR y control de la reacción en tiempo real durante los ciclos de temperatura que comprende: una serie de recipientes […]
APARATO PARA LLEVAR A CABO LA PCR Y MONITORIZACIÓN DE LA REACCIÓN EN TIEMPO REAL DURANTE CICLOS DE TEMPERATURA, del 21 de Marzo de 2011, de UNIVERSITY OF UTAH RESEARCH FOUNDATION: Aparato para llevar a cabo la PCR y control de la reacción en tiempo real durante los ciclos de temperatura que comprende: una serie de recipientes […]
 MÉTODOS PARA EL RÁPIDO ANÁLISIS FORENSE DE ADN MITOCONDRIAL Y CARATERIZACIÓN HETEROPLASMIA DE ADN MITOCONDRIAL, del 16 de Febrero de 2011, de IBIS BIOSCIENCES, INC: Un método forense que comprende los pasos de: (i) determinar una primera masa molecular de un primer producto de amplificación de un Cebador amplicón identificador […]
MÉTODOS PARA EL RÁPIDO ANÁLISIS FORENSE DE ADN MITOCONDRIAL Y CARATERIZACIÓN HETEROPLASMIA DE ADN MITOCONDRIAL, del 16 de Febrero de 2011, de IBIS BIOSCIENCES, INC: Un método forense que comprende los pasos de: (i) determinar una primera masa molecular de un primer producto de amplificación de un Cebador amplicón identificador […]
HUELLA GENOMICA PARA EL PRONOSTICO DE LA EVOLUCION DE ADENOCARCINOMA COLORECTAL, del 2 de Febrero de 2011, de UNIVERSIDAD AUTONOMA DE MADRID: La presente invención se relaciona con una huella genómica formada por un conjunto de 13 genes, cuya cuantificación de la expresión respecto a genes […]
CONJUNTO DE CEBADORES, SONDAS, PROCEDIMIENTO Y KIT PARA EL GENOTIPADODEL POLIMORFISMO GENETICO - 1131T/C DEL GEN APO A5, del 27 de Enero de 2011, de UNIVERSIDAD DE MALAGA: Conjunto de cebadores, sondas, procedimiento y kit para el genotipado del polimorfismo genético -1131T/C del gen APO A5.La presente invención se refiere […]
HUELLA GENOMICA DE CANCER DE MAMA, del 24 de Enero de 2011, de CENTRO DE INVESTIGACIONES ENERGETICAS, MEDIOAMBIENTALES Y TECNOLOGICAS: La presente invención se relaciona con métodos in vitro para determinar el pronóstico de un sujeto diagnosticado de cáncer de mama a desarrollar […]
INFIDELIDAD DE LA TRANSCRIPCIÓN, SU DETECCIÓN Y SUS USOS, del 3 de Febrero de 2012, de TRANSMEDI SA: Un método para detectar la predisposición o la etapa de un cáncer en un sujeto mamífero, comprendiendo dicho método evaluar in vitro o ex vivo la presencia o la […]