METODO PARA EL ENSAMBLAJE DE UN POLINUCLEOTIDO DE CADENA DOBLE.
Un método de ensamblaje de un polinucleótido de cadena doble que comprende:
a) seleccionar un oligonucleótido de iniciación de cadena parcialmente doble, en el que dicho oligonucleótido de iniciación comprende al menos un saliente;
b) poner en contacto dicho oligonucleótido de iniciación de cadena parcialmente doble con un siguiente oligonucleótido de cadena doble más terminal, en el que dicho siguiente oligonucleótido de cadena doble más terminal es contiguo al oligonucleótido de iniciación y comprende al menos un saliente, y en el que al menos un saliente de dicho siguiente oligonucleótido de cadena doble más terminal es complementario a al menos un saliente de dicho oligonucleótido de iniciación;
c) repetir (b) para añadir secuencialmente el siguiente oligonucleótido de cadena doble más terminal al oligonucleótido de iniciación extendido, con lo que se sintetiza dicho polinucleótido de cadena doble,
en el que dicho oligonucleótido de iniciación en la etapa a) tiene un saliente 3'' y uno 5'', y/o
en el que dicho siguiente oligonucleótido más terminal tiene un saliente 3'' y uno 5''
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2002/024488.
Solicitante: CENTOCOR, INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 200 GREAT VALLEY PARKWAY,MALVERN, PA 19355.
Inventor/es: EVANS, GLEN, A.
Fecha de Publicación: .
Fecha Concesión Europea: 9 de Diciembre de 2009.
Clasificación Internacional de Patentes:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/66 C12N 15/00 […] › Métodos generales para insertar un gen en un vector para formar un vector recombinante, utilizando la escisión y la unión; Utilización de "linkers" no funcionales o de adaptadores, p. ej. "linkers" que contienen la secuencia para una endonucleasa de restricción.
Clasificación PCT:
- C12P19/34 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
Fragmento de la descripción:
Método para el ensamblaje de un polinucleótido de cadena doble.
Antecedentes de la invención
La presente invención se refiere, de modo general, al área de la bioinformática y más específicamente a métodos para el ensamblaje de polinucleótidos dirigido por ordenador.
Las enzimas, anticuerpos, receptores y ligandos son polipéptidos que han evolucionado mediante presión selectiva para realizar funciones biológicas muy específicas dentro de un organismo vivo. El uso de un polipéptido para aplicaciones tecnológicas específicas puede requerir que el polipéptido funcione en entornos o sobre sustratos para los que no se seleccionó evolutivamente. Los polipéptidos aislados de microorganismos que crecen en entornos extremos proporcionan abundantes pruebas de que estas moléculas son, en general, maleables con respecto a estructura y función. Sin embargo, el proceso para aislar un polipéptido de su entorno nativo es costoso y requiere tiempo. Por lo tanto, se necesitan nuevos métodos para desarrollar de forma sintética material genético que codifique un polipéptido que posea una actividad deseada.
Existen dos maneras de obtener material genético para manipulaciones de ingeniería genética: (1) aislamiento y purificación de un polinucleótido en forma de ADN o ARN a partir de fuentes naturales o (2) la síntesis de un polinucleótido usando diversos enfoques químicos-enzimáticos. El primer enfoque se limita a secuencias de origen natural que no se prestan fácilmente a la modificación específica. El último enfoque es mucho más complicado y requiere un trabajo más intenso. Sin embargo, el enfoque químico-enzimático tiene muchas características atractivas incluyendo la posibilidad de preparar, sin ninguna limitación significativa, cualquier secuencia de polinucleótidos deseable.
Actualmente existen dos métodos generales para el ensamblaje sintético de oligonucleótidos en largos fragmentos de polinucleótidos. En primer lugar, se permite que los oligonucleótidos que abarcan toda la secuencia a sintetizar hibriden, y a continuación se reparan las mellas con ligasa. A continuación, se clona el fragmento directamente, o se clona después de la amplificación mediante la reacción en cadena de la polimerasa (PCR). El polinucleótido se usa posteriormente para ensamblaje in vitro en secuencias más largas. El segundo método general para la síntesis de genes utiliza polimerasa para rellenar huecos de cadena sencilla en los pares hibridados de oligonucleótidos. Después de la reacción de la polimerasa, las regiones de cadena sencilla de los oligonucleótidos se vuelven de cadena doble, y después de la digestión con endonucleasa de restricción, pueden clonarse directamente o usarse para ensamblaje adicional de secuencias más largas ligando fragmentos de cadena doble diferentes. Típicamente, posteriormente a la reacción de la polimerasa, cada segmento debe clonarse lo que retrasa significativamente la síntesis de fragmentos largos de ADN y reduce enormemente la eficacia de este enfoque. Itakura et al., Science vol. 198, p 1056-1063, (1978) describe un método en el cual se forman y se ligan dos oligonucleótidos de cadena parcialmente doble con salientes.
La creación de polinucleótidos totalmente nuevos, o la modificación sustancial de polinucleótidos existentes, requiere mucho tiempo, es cara, requiere múltiples y complejas etapas, y en algunos casos en imposible. Por lo tanto, existe una gran necesidad de un medio eficaz para ensamblar polinucleótidos sintéticos de cualquier secuencia deseada. Dicho método podría aplicarse de forma universal. Por ejemplo, el método podría usarse para preparar eficazmente una selección de polinucleótidos que tienen sustituciones específicas en una secuencia conocida que se expresa y se criba en busca de una función mejorada. La presente invención satisface estas necesidades proporcionando métodos eficaces y potentes para la síntesis de un polinucleótido diana que codifica un polipéptido diana.
Sumario de la invención
La presente invención proporciona métodos para el ensamblaje sintético de polinucleótidos y algoritmos relacionados. En particular, la presente invención proporciona métodos rápidos y eficaces para generar cualquier secuencia de ácido nucleico, incluyendo genes completos, segmentos cromosómicos, cromosomas y genomas. Puesto que este enfoque se basa en un enfoque completamente sintético, no existen limitaciones, tales como la disponibilidad de ácidos nucleicos existentes, para impedir la construcción de incluso segmentos muy grandes de ácido nucleico.
Breve descripción de los dibujos
Símbolos de referencia similares en los diversos dibujos indican elementos similares.
La Figura 1 representa placas de 96 pocillos para la síntesis de oligonucleótidos F (es decir, directos
o de cadena más
), síntesis de oligonucleótidos R (es decir, inversos
o de cadena menos
), y una placa T (es decir, temperatura
) para la hibridación de oligonucleótidos F y T.
La Figura 2 representa el plan de mezclado de oligonucleótidos en el que oligonucleótidos F y oligonucleótidos R hibridan para formar un polinucleótido contiguo.
La Figura 3 representa el esquema de ensamblaje de una secuencia de polinucleótido diana que define un gen, genoma, conjunto de genes o secuencia polipeptídica. La secuencia se diseña por ordenador y se usa para generar un conjunto de fragmentos de oligonucleótidos analizados que abarcan la cadena + y - de una secuencia de polinucleótido diana que codifica un polipéptido diana.
La Figura 4 representa un esquema de los módulos de síntesis de polinucleótidos. Un cabezal de nanodispensado con una pluralidad de válvulas depositará productos químicos de síntesis en recipientes de ensamblaje. La distribución de productos químicos desde el depósito de reactivos puede controlarse usando una bomba de jeringa. Por debajo de las cámaras de reacción hay un conjunto de recipientes de ensamblaje unidos a microcanales que mueven los fluidos mediante microfluídica.
La Figura 5 representa que la síntesis de oligonucleótidos, el ensamblaje de oligonucleótidos mediante mezclado e hibridación, y el ligamiento pueden realizarse usando el mezclado microfluídico.
La Figura 6 representa el mezclado secuencial de oligonucleótidos sintetizados en matrices.
La Figura 7 representa la fase de mezclado de los componentes de oligonucleótido a través de los colectores de ensamblaje que da como resultado el completo ensamblaje de todos los oligonucleótidos a partir de la matriz.
La Figura 8 representa un ejemplo de un módulo de ensamblaje que comprende un conjunto completo de colectores de mezclado producidos usando microfabricación en una única unidad. Diversas configuraciones del colector de mezclado permitirán el ensamblaje de mayores números de matrices de pocillos de oligonucleótidos componentes analizados.
La Figura 9 representa la configuración para el ensamblaje de oligonucleótidos sintetizados en una matriz predefinida. El paso a través del dispositivo de ensamblaje en presencia de ADN ligasa y otro tampón apropiado y componentes químicos facilitará el ensamblaje de polinucleótidos de cadena doble.
La Figura 10 representa un ejemplo del diseño del dispositivo de mezclado. Microsurcos o canales microfluídicos se graban en la superficie del dispositivo de mezclado. El dispositivo proporciona un recipiente de micro-reacción en la unión de dos canales para 1) mezclar los dos chorros, 2) mantenimiento controlado o ciclado de la temperatura en el punto de la unión y 3) expulsión de la mezcla ligada desde el canal de salida al siguiente conjunto de cámaras de mezclado y ligamiento.
La Figura 11 representa el diseño de una plataforma de síntesis de polinucleótidos que comprende placas de micropocillos a las que se dirigen una pluralidad de canales para el microdispensado.
La Figura 12 representa un ejemplo de una plataforma de síntesis de polinucleótidos de alta capacidad usando microplacas de micropocillos de alta densidad capaces de sintetizar más de 1536 oligonucleótidos componentes por placa.
La Figura 13 representa un formado de ensamblaje de polinucleótidos que usa síntesis de oligonucleótidos unidos a una superficie en lugar de síntesis soluble. En esta configuración, los oligonucleótidos se sintetizan con un enlazador que permite la unión a un soporte sólido.
La Figura 14 representa un diagrama de ensamblaje de polinucleótido sistemático...
Reivindicaciones:
1. Un método de ensamblaje de un polinucleótido de cadena doble que comprende:
en el que dicho oligonucleótido de iniciación en la etapa a) tiene un saliente 3' y uno 5', y/o
en el que dicho siguiente oligonucleótido más terminal tiene un saliente 3' y uno 5'.
2. Un método de ensamblaje de un polinucleótido diana que comprende:
3. El método de la reivindicación 2, en el que dicho oligonucleótido de iniciación se extiende de manera bidireccional.
4. El método de la reivindicación 2, en el que dicho oligonucleótido de iniciación se extiende de manera unidireccional.
5. El método de la reivindicación 2, en el que dicho oligonucleótido de iniciación es el oligonucleótido 5' más terminal de dicho polipéptido diana.
6. El método de la reivindicación 2, en el que dicho oligonucleótido de iniciación es el oligonucleótido 3' más terminal de dicho polipéptido diana.
7. El método de la reivindicación 2, en el que el polinucleótido diana comprende una secuencia que codifica un polipéptido diana.
8. El método de la reivindicación 7, en el que dicho polipéptido diana es una proteína.
9. El método de la reivindicación 8, en el que dicha proteína es una enzima.
10. El método de la reivindicación 2, en el que dicho oligonucleótido de iniciación comprende una secuencia identificada por un programa informático.
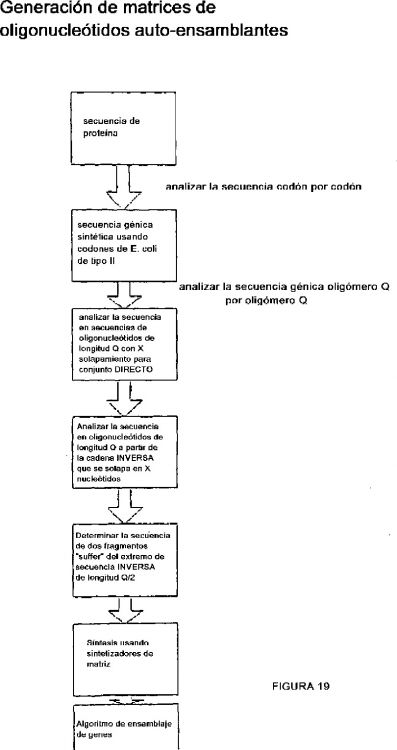
11. El método de la reivindicación 10, en el que dicho programa informático comprende un algoritmo que comprende las siguientes etapas:
sufferde longitud Q/2 en el extremo de la secuencia INVERSA; y
12. El método de la reivindicación 2, en el que dicho saliente complementario de dicho siguiente oligonucleótido de cadena doble más terminal comprende aproximadamente el cincuenta por ciento de la longitud de la cadena que tiene dicho saliente complementario.
13. El método de la reivindicación 12, en el que dicha cadena que tiene dicho saliente complementario tiene aproximadamente de 15 a 1000 nucleótidos de longitud.
14. El método de la reivindicación 13, en el que dicha cadena que tiene dicho saliente complementario tiene aproximadamente de 20 a 500 nucleótidos de longitud.
15. El método de la reivindicación 14, en el que dicha cadena que tiene dicho saliente complementario tiene aproximadamente de 25 a 100 nucleótidos de longitud.
16. El método de la reivindicación 2, en el que el oligonucleótido de iniciación está unido a un soporte sólido.
17. Un método de ensamblaje de un polinucleótido de cadena doble que comprende:
Patentes similares o relacionadas:
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Método para romper un ácido nucleico y añadir un adaptador por medio de transposasa y reactivo, del 1 de Julio de 2020, de MGI Tech Co., Ltd: Un metodo para romper un acido nucleico y anadir un adaptador por medio de una transposasa, que comprende las siguientes etapas: interrumpir […]
Vacuna subunitaria contra Mycoplasma spp., del 1 de Julio de 2020, de Agricultural Technology Research Institute: Una composición para prevenir una infección por Mycoplasma spp., que comprende: un principio activo, que comprende una proteína de PdhA; y un adyuvante […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Etiquetado y evaluación de una secuencia diana, del 13 de Mayo de 2020, de RhoDx, Inc: Un método para modificar un ácido nucleico, que comprende: (a) poner en contacto un ácido nucleico de cadena sencilla con una actividad de transferasa […]
Preparación de bibliotecas de ácido nucleico marcado usando protocolo de adición en un solo tubo, del 13 de Mayo de 2020, de ILLUMINA, INC: Un método para preparar una biblioteca de fragmentos de ácido nucleico marcados que comprende: (a) poner en contacto una célula individual […]
Proteínas de captura de la superficie celular recombinantes, del 13 de Mayo de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para detectar y aislar células que producen altos niveles de una proteína heterodimérica que tiene una primera subunidad y una segunda subunidad, […]