METODO DE MUTAGENESIS.
Un método para introducir una o más mutaciones en una molécula de ácido nucleico,
que comprende las siguientes etapas:
(a) sintetizar un cebador mutagénico a partir de una molécula plantilla de ácido nucleico, en donde dicho cebador mutagénico contiene una o más mutaciones respecto a una molécula de partida;
(b) aislar una forma de cadena sencilla de dicho cebador mutagénico;
(c) madurar dicho cebador mutagénico de cadena sencilla a una forma de cadena sencilla de dicha molécula de partida; y
(d) sintetizar una cadena complementaria a partir de dicho cebador mutagénico para producir una molécula de ácido nucleico de forma circular que contenga dichas mutaciones
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2006/000059.
Solicitante: MEDIMMUNE LIMITED.
Nacionalidad solicitante: Reino Unido.
Dirección: MILSTEIN BUILDING GRANTA PARK,CAMBRIDGE CB21 6GH.
Inventor/es: MINTER,RALPH,RAYMOND MEDIMMUNE LIMITED, LANE,STEVEN,GODFREY MEDIMMUNE LIMITED, HOLGATE,ROBERT,GEORGE,EDWARD MEDIMMUNE LIMITED, GROVES,MARIA,ANASTASIA,THERESA MEDIMMUNE LIMITED.
Fecha de Publicación: .
Fecha Concesión Europea: 14 de Abril de 2010.
Clasificación Internacional de Patentes:
- C12N15/10B
Clasificación PCT:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
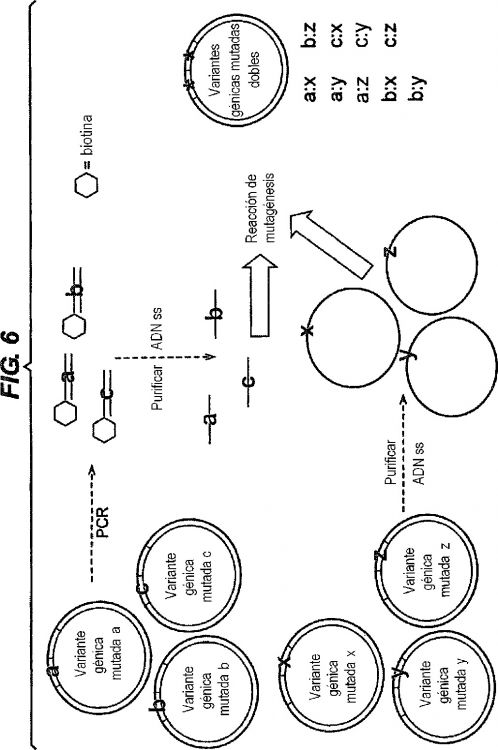
Fragmento de la descripción:
Método de mutagénesis.
La presente invención se refiere a un método para producir moléculas de ácido nucleico mutagenizadas. En particular, la presente invención se refiere a un método en el que el propio cebador usado en la reacción de mutagénesis (el cebador mutagénico) se sintetiza a partir de una molécula plantilla de ácido nucleico. El resultado de la reacción de mutagénesis es una molécula de ácido nucleico circular mutagenizada que puede ser transformada, por ejemplo, en bacterias sin necesidad de modificaciones adicionales. En las realizaciones preferidas, el método se aplica a poblaciones de moléculas, por ejemplo en la creación o en el escrutinio de poblaciones de moléculas. El método también puede usarse de un modo combinatorio, en el que se usan múltiples cebadores mutagénicos para mutar múltiples moléculas de partida.
Una de las etapas implicadas en las técnicas biológicas moleculares es la introducción de ácido nucleico en un vector. Por ejemplo, se puede digerir e introducir un fragmento de ADN o una población de fragmentos de ADN mediante incorporación a un vector. Por ejemplo, se puede digerir un fragmento de ADN (o población de fragmentos) y eliminarlo de un primer vector (por ejemplo, un plásmido) en un segundo vector en un proceso conocido como subclonación. De este modo, se digiere un primer vector con las mismas enzimas de restricción que el segundo vector de tal modo que se producen extremos sobresalientes complementarios para la ligación. El fragmento de ADN y el segundo vector son ligados entonces en virtud a sus extremos sobresalientes complementarios y el resultado es un vector completado, que contiene el fragmento de ADN, que puede ser replicado en una célula hospedante (por ejemplo, en bacterias).
Por lo tanto, se pueden producir grandes cantidades de ADN para su uso en diversas aplicaciones de biología molecular. O el vector en el que se ha introducido el ADN puede tener un propósito particular (por ejemplo, puede ser un vector de sobreexpresión). Una vez introducido en un vector de expresión de ese tipo, la proteína codificada por el fragmento de restricción puede ser expresada a partir del vector en una célula hospedante. Una vez expresada, la proteína resultante podría ser purificada para su uso final. O, particularmente cuando el método se use para incorporar una población de fragmentos de ADN en un vector de expresión, la proteína expresada puede usarse en un escrutinio (por ejemplo, mediante afinidad de unión a una diana molecular).
También se conocen varios métodos para introducir mutaciones en ADN. El trabajo de Barik (Barik, Methods in Molecular Biology Volumen 192: PCR Cloning Protocols, 2ª edición, páginas 189-196, Editores B-Y Chen y H. W Janes, Humana Press Inc, Totowa, NJ; Burke y Barik, Methods In Molecular Biology Volumen 226: PCR Protocols, 2ª edición, páginas 525-531, Editores JMS Bartless y D Stirling, Humana Press Inc, Totowa, NJ) muestra cómo usar una estrategia basada en PCR para introducir mutaciones en ADN lineal. Según dicho método, se han usado los denominados megaprímeros para introducir mutaciones en una plantilla lineal en una reacción de PCR. Sin embargo, dichos métodos dan como resultado un producto de ADN de doble cadena lineal que debe ser subclonado a su vez en un vector apropiado a fin de producir una forma replicante para, por ejemplo, el aumento de escala o para la expresión de proteínas.
Miyazaki propuso un método que implica PCR de un plásmido entero (Miyazaki, Methods In Molecular Biology: Directed Evolution Library Creation: Methods and Protocols. Editores Amold y Georglou, Humana Press Inc. Totowa, NJ; Biotechniques 33: 1033-1038). En este método se usa un plásmido de cadena doble como plantilla en una reacción PCR, al que se añade para cebado un producto de PCR de cadena doble que contiene la mutación que se va a introducir. Sin embargo, la propiedad de cadena doble del cebador y la plantilla pueden provocar una auto-maduración de las cadenas de cebador y las cadenas de plantilla en lugar de una maduración cruzada, y por lo tanto se usa una relación específica de cebador a plantilla para minimizar la auto-maduración. Wang y Malcolm (1999) también describen un método de mutagénesis basado en PCR de plásmidos (Wang y Malcolm, Biotechniques, volumen 26, nº 4, 1999, páginas 680-684).
Además de los problemas de auto-maduración que se producen con una plantilla de cadena doble, existen otros problemas cuando se usa una plantilla de cadena doble en una reacción PCR. En una reacción exponencial, la capacidad para introducir errores es elevada, ya que el error introducido en una ronda se traslada a cada ronda posterior en base exponencial. Además, cuando dicha plantilla de PCR es una plantilla grande, tal como un vector, por ejemplo un plásmido, esto aumenta aún más la probabilidad de introducir errores.
También se conoce el uso de oligonucleótidos sintéticos para mutar ADN (Kunkel y col., (1985) PNAS USA, volumen 82, páginas 488-492; Sidhu y col., Phage Display for the Selection of Novel Binding Peptides). La patente EP 1108783 también se refiere a un método de mutagénesis que usa oligonucleótidos sintéticos. Sin embargo, existen limitaciones en la longitud de los oligonucleótidos que pueden sintetizarse químicamente. Adicionalmente, para programar el sintetizador, se debe conocer la secuencia y debe introducirse por adelantado en el sintetizador para su producción.
Por tanto, existe una necesidad de métodos alternativos de mutagénesis. En particular, de métodos que produzcan un resultado que pueda ser transformado directamente en bacterias, fácil de usarse en etapas posteriores, tal como en escrutinios de expresión (evitando la necesidad de etapas adicionales de digestión y ligación para introducir ácido nucleico en un vector apropiado). También existe la necesidad de métodos de mutagénesis rápidos y eficaces, y de métodos que sean fácilmente escalables, por ejemplo, para uso en la producción de poblaciones para escrutinio o producción de bibliotecas.
La presente invención, en un aspecto, se refiere a un método para introducir una o más mutaciones en una molécula de ácido nucleico, que comprende: madurar una forma de cadena sencilla de un cebador mutagénico con una molécula de partida, habiendo sido sintetizado dicho cebador mutagénico a partir de una molécula de ácido nucleico plantilla y que contiene una o más mutaciones respecto a dicha molécula de partida; y sintetizar una cadena complementaria a partir de dicho cebador mutagénico de tal modo que se produzca una molécula de ácido nucleico de forma circular que contenga dichas mutaciones.
En un aspecto, la presente invención proporciona un método para introducir una o más mutaciones en una molécula de ácido nucleico, que comprende las siguientes etapas:
Por tanto, el cebador mutagénico es sintetizado a partir de una molécula de ácido nucleico plantilla usando una enzima adecuada, usando métodos conocidos en la técnica. Es preferible que dicho cebador mutagénico sea sintetizado usando un método basado en reacción en cadena de polimerasa (PCR).
La molécula plantilla y la molécula de partida pueden ser la misma, o pueden contener la misma secuencia, que forma la base de la síntesis del cebador mutagénico. Por tanto, en algunas realizaciones, la secuencia de la plantilla para la síntesis de cebador mutagénico es la misma que la encontrada en la molécula de partida. En estas realizaciones, las mutaciones pueden introducirse en virtud a la introducción de errores durante la síntesis del cebador mutagénico de la molécula de partida, por ejemplo, mediante una polimerasa propensa al error. Por tanto, en algunas realizaciones, el cebador mutagénico se sintetiza mediante un método de síntesis en el que se introducen errores cuando se sintetiza una segunda cadena a partir de una primera cadena, preferiblemente mediante PCR propensa...
Reivindicaciones:
1. Un método para introducir una o más mutaciones en una molécula de ácido nucleico, que comprende las siguientes etapas:
2. El método de acuerdo con la reivindicación 1, en el que antes de dicha etapa de maduración (c), el cebador de cadena sencilla es mezclado con la molécula de partida de cadena sencilla.
3. El método de la reivindicación 1, en el que el cebador de cadena sencilla de la etapa (b) es aislado mediante el uso de un medio separativo.
4. El método de acuerdo con la reivindicación 2, que además comprende la etapa de aislar dicha molécula de partida de cadena sencilla antes de la etapa (c).
5. El método de acuerdo con la reivindicación 4, que además comprende una etapa de exclusión por tamaño llevada a cabo sobre dicha molécula de partida de cadena sencilla antes de la etapa (c).
6. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicha molécula de partida es ADN circular de cadena sencilla.
7. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que la molécula de partida es un vector de expresión o un vector de panel de fago.
8. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicha molécula de forma circular es ADN circular cerrado covalentemente.
9. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, que además comprende la etapa de transformar la molécula de ácido nucleico mutagenizado en células hospedantes.
10. El método de acuerdo con la reivindicación 9, en el que dicha transformación se lleva a cabo usando electroporación.
11. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, que comprende una etapa llevada a cabo en las condiciones que permiten seleccionar preferentemente la cadena complementaria que contiene las mutaciones.
12. El método de la reivindicación 11, en el que dicha selección preferente de la cadena complementaria es el resultado de la digestión preferente de la molécula de partida.
13. El método de la reivindicación 11, en el que la molécula de partida contiene modificaciones de tal modo que la cadena complementaria se seleccione preferentemente.
14. El método de la reivindicación 13, que además comprende la etapa de introducir dichas modificaciones.
15. El método de la reivindicación 13 ó de la reivindicación 14, en el que la modificación es la introducción de dU en lugar de dT.
16. El método de la reivindicación 13 ó de la reivindicación 14, en el que la modificación es la metilación o la introducción de un gen condicionalmente letal.
17. El método de una cualquiera de las reivindicaciones 11 a 16, en el que dicha selección preferente se lleva a cabo transformando dicha molécula de forma circular de la etapa (d) en una célula hospedante que sea selectiva para la cadena complementaria, no modificada.
18. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicho cebador mutagénico se sintetiza mediante un método basado en PCR.
19. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicho cebador mutagénico se sintetiza mediante un método de síntesis en el que se introducen errores cuando se sintetiza una segunda cadena a partir de una primera cadena.
20. El método de acuerdo con la reivindicación 19, en el que dicho método es PCR propensa al error.
21. El método de acuerdo con la reivindicación 19 ó con la reivindicación 20, en el que la secuencia de la plantilla para la síntesis del cebador mutagénico es la misma obtenida en la molécula de partida.
22. El método de acuerdo con la reivindicación 3, en el que la etapa de síntesis de dicho cebador mutagénico introduce un resto de unión bien en la cadena positiva o bien en la cadena negativa del cebador mutagénico, y dicha preparación se lleva a cabo a través de la unión de dicho resto de unión con su pareja de unión en dicho medio separativo.
23. El método de acuerdo con la reivindicación 22, en el que dicho resto de unión se introduce en la cadena positiva del cebador mutagénico y la cadena negativa del cebador mutagénico se eluye de dicho medio separativo.
24. El método de acuerdo con la reivindicación 22 ó con la reivindicación 23, en el que dicho resto de unión es biotina y dicha pareja de unión es estreptavidina.
25. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicha(s) mutación(es) incluye sustituciones.
26. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicha(s) mutación(es) incluye eliminaciones.
27. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicha(s) mutación(es) incluye adiciones.
28. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dichas mutaciones son mutaciones en una pluralidad de nucleótidos.
29. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicho cebador mutagénico tiene al menos un 80% de complementariedad con la molécula de partida.
30. El método de acuerdo con la reivindicación 29, en el que dicho cebador mutagénico tiene al menos un 90% de complementariedad con la molécula de partida.
31. El método de acuerdo con la reivindicación 30, en el que dicho cebador mutagénico tiene al menos un 95% de complementariedad con la molécula de partida.
32. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que dicho cebador mutagénico tiene un tamaño entre 100 nucleótidos y 4.000 nucleótidos.
33. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que la relación molar de cebador mutagénico a molécula de partida en la etapa (c) es aproximadamente 3:1.
34. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que existen múltiples moléculas de partida y/o múltiples moléculas de cebador mutagénico.
35. El método de acuerdo con una cualquiera de las reivindicaciones precedentes, en el que la molécula de partida, la plantilla o el cebador mutagénico contienen un dominio variable de anticuerpo.
36. El método de acuerdo con la reivindicación 35, en el que dicho dominio variable de anticuerpo es un dominio de anticuerpo VL.
37. El método de acuerdo con la reivindicación 35, en el que dicho dominio variable de anticuerpo es un dominio de anticuerpo VH.
38. El método de acuerdo con la reivindicación 35, en el que la molécula de partida contiene un dominio de anticuerpo VH y un dominio de anticuerpo VL y la molécula de cebador mutagénico contiene un dominio de anticuerpo VL para mutar el dominio VL de la molécula de partida.
39. El método de la reivindicación 35, en el que la molécula de partida contiene un dominio de anticuerpo VL y un dominio de anticuerpo VH y la molécula de cebador mutagénico contiene un dominio de anticuerpo VH para mutar el dominio de anticuerpo VH sobre la molécula de partida.
40. El método de la reivindicación 38 ó de la reivindicación 39, en el que se usan un primer cebador mutagénico y un segundo cebador mutagénico para mutar la molécula de partida, conteniendo el primer cebador mutagénico un dominio de anticuerpo VH para mutar el dominio VH en la molécula de partida, y conteniendo el segundo cebador mutagénico un dominio de anticuerpo VH para mutar el dominio VL en la molécula de partida.
41. El método de la reivindicación 40, en el que dicho primer cebador mutagénico y dicho segundo cebador mutagénico se usan en etapas de reacción de mutagénesis separadas.
42. El método de la reivindicación 41, en el que dicho primer cebador mutagénico y dicho segundo cebador mutagénico se usan en la misma etapa de reacción de mutagénesis.
43. El método de la reivindicación 38 ó de la reivindicación 39, en el que se usa un único cebado mutagénico que contiene ambos dominios, VH y VL, para mutar los dominios VH y VL en la molécula de partida.
44. El método de acuerdo con cualquiera de las reivindicaciones 34 a 43, en el que la molécula de partida comprende ácido nucleico que codifica un scFv.
Patentes similares o relacionadas:
VECTORES MUTACIONALES DE OLIGODESOXINUCLEÓTIDOS DE CADENA SENCILLA, del 9 de Mayo de 2011, de Cibus International L.P: Un método de obtención de una célula animal que contiene un cambio genético predeterminado en un gen cromosómico fijado como diana que comprende: a) proporcionar […]
EVOLUCIÓN IN VITRO CONTINUA, del 2 de Febrero de 2011, de CEPHALON AUSTRALIA (VIC) PTY LTD: Un método para la mutación de una proteína de interés, síntesis de un mutante de la proteína de interés y selección de un mutante de la proteína de interés que […]
 MODIFICACION GENETICA DE CELULAS SOMATICAS Y USOS DE LAS MISMAS, del 11 de Mayo de 2010, de PPL THERAPEUTICS (SCOTLAND) LIMITED: Un procedimiento de transferencia nuclear que comprende:
(a) preparar una célula somática no humana para la transferencia nuclear por un procedimiento que comprende […]
MODIFICACION GENETICA DE CELULAS SOMATICAS Y USOS DE LAS MISMAS, del 11 de Mayo de 2010, de PPL THERAPEUTICS (SCOTLAND) LIMITED: Un procedimiento de transferencia nuclear que comprende:
(a) preparar una célula somática no humana para la transferencia nuclear por un procedimiento que comprende […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]