POLIPEPTIDOS PARA ENSAMBLAJE OLIGOMERICO DE ANTIGENOS.
Un polipéptido que comprende: (a) un dominio antigénicos; (b) un dominio de oligomerización;
y (c) un dominio transmembrana, en el que los dominios (a), (b) y (c) no se encuentran todos juntos en el mismo polipéptido en la naturaleza y en el que el dominio (b) es un dominio de superenrollamiento de la adhesina NadA de Neisseria meningitidis
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/IB2005/002528.
Solicitante: NOVARTIS VACCINES AND DIAGNOSTICS S.R.L..
Nacionalidad solicitante: Italia.
Dirección: VIA FIORENTINA 1 53100 SIENA (SI) ITALIA.
Inventor/es: MASIGNANI,VEGA,CHIRON SRL, RAPPUOLI,RINO,CHIRON SRL, SCARSELLI,MARIA,CHIRON SRL, CAPECCHI,Barbara,Chiron Srl.
Fecha de Publicación: .
Fecha Solicitud PCT: 22 de Julio de 2005.
Clasificación Internacional de Patentes:
- C12N15/62 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Secuencias de ADN que codifican proteínas de fusión.
Clasificación PCT:
- C07K14/705 C […] › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › Receptores; Antígenos celulares de superficie; Determinantes celulares de superficie.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
PDF original: ES-2355980_T3.pdf


Fragmento de la descripción:
CAMPO DE LA INVENCIÓN
La presente invención se encuentra en el campo de la presentación de antígenos. Más particularmente, se refiere a la modificación de proteínas para permitir su expresión en forma oligomérica, por ejemplo, en la superficie de una célula. 5
ANTECEDENTES DE LA INVENCIÓN
Muchos polipéptidos que son naturalmente inmunogénicos pierden esta propiedad cuando se expresan de forma recombinante. En algunos casos el polipéptido nativo tiene elementos estructurales que no se forman durante la expresión en un huésped heterólogo, por ejemplo, las modificaciones post-traduccionales pueden ser incorrectas, las interacciones intermoleculares que influyen en la conformación puede perderse, etc. Una causa adicional de 10 inmunogenicidad perdida es cuando un polipéptido (por ejemplo, un polipéptido expuesto en superficie) es oligomérico de forma natural y cuando esta estructura cuaternaria se requiere para inmunogenicidad (por ejemplo, cuando el polipéptido tiene epítopos que se presentan solamente cuando está presente una estructura oligomérica cuaternaria específica). La pérdida de estructura oligomérica puede significar que la proteína monomérica es menos inmunógena que su homólogo oligomérico nativo. 15
En otros casos el polipéptido nativo puede ser un polipéptido transmembrana que no es susceptible de expresión en un huésped recombinante. Estos problemas con frecuencia vistos cuando van a expresarse polipéptidos de eucariotas (incluyendo los de virus de eucariotas) en procariotas. Un modo de mejorar la expresión de polipéptidos transmembrana virales es retirar sus dominios transmembrana y expresar solamente los dominios extracelulares antigénicos {1}. Sin embargo, esta tecnología de “receptor soluble” sufre de nuevo pérdida de estructura cuaternaria. Si 20 existe un receptor nativo en una forma oligomérica en la superficie de un virus y la oligomerización surge de secuencias en la región transmembrana, el receptor soluble perderá su capacidad de oligomerizar y esta pérdida puede tener consecuencias funcionales, por ejemplo, pérdida de señalización o de avidez. La pérdida de actividad de unión, incluso aunque se conserve afinidad de unión, es un problema particular para antígenos, por ejemplo, los usados en vacunas.
Se han desvelado técnicas para proteínas que oligomerizan en las referencias 2 y 3. 25
Es objeto de la presente invención proporcionar modos de mejorar la expresión de polipéptidos y particularmente de polipéptidos antigénicos, por ejemplo, para conservar su estructura oligomérica.
SUMARIO DE LA INVENCIÓN
La invención se basa en un sistema para la expresión de polipéptidos antigénicos en forma oligomérica. El polipéptido antigénico se fusiona con un polipéptido de oligomerización de modo que el polipéptido de oligomerización 30 puede interaccionar con otros polipéptidos de oligomerización y proporcionar múltiples copias del polipéptido antigénico en proximidad estrecha en forma de un oligómero. La expresión de los polipéptidos en forma oligomérica de este modo puede mejorar su inmunogenicidad en comparación con una forma monomérica.
De este modo la invención proporciona un procedimiento para expresar un polipéptido de interés en una forma oligomérica recombinante, en el que el polipéptido de interés se fusiona con un polipéptido de oligomerización de modo 35 que puede asociarse una pluralidad de dominios de oligomerización para presentar el polipéptido de interés en forma oligomérica. En particular, el procedimiento puede aplicarse (a) para presentar el polipéptido oligomerizado en la superficie de una membrana, pero incluyendo una secuencia transmembrana en la estructura y (b) para presentar el polipéptido oligomerizado mediante el uso de elementos estructurales de una adhesina, tal como una adhesina bacteriana, por ejemplo, la adhesina NadA {4} de Neisseria meningitidis. 40
La invención puede aplicarse a cualquier polipéptido antigénico, incluyendo antígenos virales y no virales. Es particularmente adecuada para expresar polipéptidos de superficie en una forma oligomérica, tal como las partes extracelulares de proteínas de superficie que se encuentran de forma natural en una forma oligomérica. El polipéptido de interés puede ser el polipéptido de longitud completa o, como alternativa, puede ser un fragmento de un polipéptido de longitud completa, por ejemplo puede comprender uno o más dominios del polipéptido de longitud completa. 45
La invención proporciona un polipéptido que comprende: (a) un dominio antigénico; (b) un dominio de oligomerización; y (c) un dominio transmembrana, en el que los dominios (a), (b) y (c) no se encuentran juntos en el mismo polipéptido en la naturaleza (y, en particular, en el que los dominios (a) y (b) no se encuentran juntos en el mismo polipéptido en la naturaleza) y en el que el dominio (6) es un dominio de superenrollamiento de la adhesina NadA de Neisseria Meningitidis. Los dominios están en el orden (a)-(b)-(c), yendo del extremo C terminal al N terminal o del 50 extremo N terminal al C terminal. Es más habitual tener el dominio transmembrana en o cerca del extremo C terminal de la proteína.
La inclusión de un dominio transmembrana en el polipéptido permite que una pluralidad de dominios de oligomerización se asocien para presentar el polipéptido en forma oligomérica en la superficie de una membrana. Además de asociarse mediante sus dominios de oligomerización, los polipéptidos también pueden asociarse mediante interacción de sus dominios transmembrana dentro de una bicapa lipídica, manteniendo de este modo la estructura oligomérica. La inclusión de secuencias transmembrana también puede ayudar en el correcto plegamiento de algunos 5 antígenos. Los multímeros de la referencia 2 se diseñan para evitar la presencia de secuencias transmembrana.
La invención proporciona un polipéptido que comprende: (a) un dominio antigénico; y (b) un dominio de oligomerización de una adhesina, en el que los dominios (a) y (b) no se encuentran juntos en el mismo polipéptido en la naturaleza y en el que el dominio (b) es un dominio de superenrollamiento de la NadA de Neisseno meningitides. Los dominios están en el orden (a)-(b), yendo del extremo C terminal al N terminal o del extremo N terminal al C terminal. El 10 polipéptido generalmente incluirá secuencias además de (a) y (b). Para la presentación en superficie de polipéptidos antigénicos, por ejemplo, la invención generalmente implicará el uso de un dominio transmembrana además de un dominio de oligomerización, como se ha descrito anteriormente. La adhesina es preferentemente una adhesina bacteriana, más preferentemente una adhesina “Oca” y más preferentemente la adhesina NadA de Neisseria Meningitidis. 15
DESCRIPCIÓN DE LOS DIBUJOS
La Figura 1 ilustra la estructura de un monómero E2 de coronavirus SRAS.
La Figura 2 ilustra los dominios dentro de la proteína NadA de meningococos {4}.
La Figura 3 ilustra los dominios dentro de la proteína gp120 Env de VIH. La Figura 4 ilustra dominios dentro de NadA. La Figura 5 muestra proteínas híbridas que comprenden regiones tanto de Env como de NadA. La 20 Figura 6 muestra la composición de las construcciones de gp120-NadA (824aa) y gp140-NadA (741 aa).
La Figura 7 transferencias de Western de E. coli que expresan gp120-NadA y gp140-NadA.
La Figura 8 muestra SDS-PAGE de E.coli que expresa gp120-NadA y gp140-NadA.
Las Figuras 9-11 muestran análisis de FACS de E.coli que expresan gp120-NadA o gp140-NadA.
La Figura 12 muestra unión a CD4 dependiente de dosis por E.coli que expresa gp140-NadA. 25
Las Figuras 13-14 muestran el número de E.coli que expresan gp140-NadA que se unen a CD4.
La Figura 15 muestra análisis de FACS de unión a CD4 por E.coli que expresan gp140-NadA, con o sin preincubación con gp140 puro.
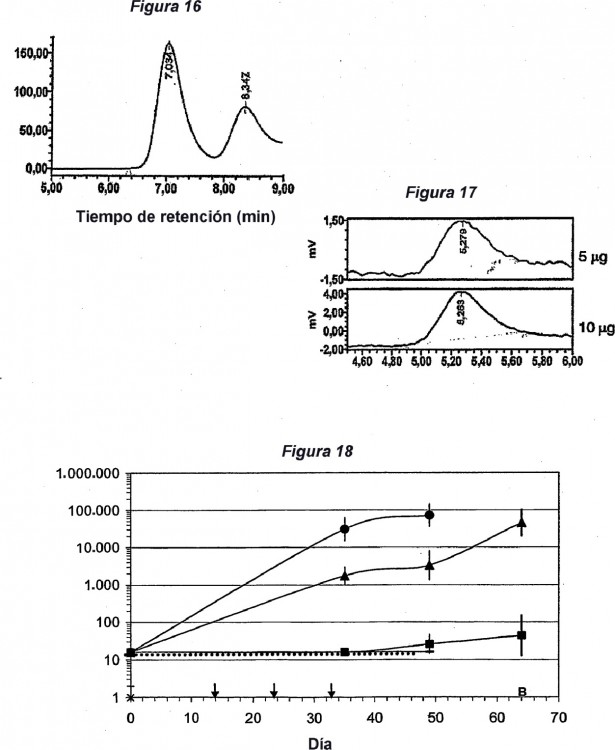
Las Figuras 16 y 17 muestran análisis de HPLC de complejos CD4/Env.
La Figura 18 muestra titulaciones de anticuerpos anti-gp140V2 según se determinaron mediante ELISA. La 30 línea horizontal punteada muestra la titulación pre-sangrado. Las flechas en el eje X muestran cuando se administraron las dosis de sensibilización y la “B” muestra la dosis de refuerzo. Las cuatro líneas de datos son, desde la parte superior a la inferior: el control negativo (-); OMV-gp140-NadA (■); OMV-gp120-NadA y gp140 (▲); y el control... [Seguir leyendo]
Reivindicaciones:
1. Un polipéptido que comprende: (a) un dominio antigénicos; (b) un dominio de oligomerización; y (c) un dominio transmembrana, en el que los dominios (a), (b) y (c) no se encuentran todos juntos en el mismo polipéptido en la naturaleza y en el que el dominio (b) es un dominio de superenrollamiento de la adhesina NadA de Neisseria meningitidis. 5
2. El polipéptido de cualquier reivindicación precedente, en el que el dominio antigénico es un antígeno de superficie de una bacteria o virus.
3. El polipéptido de la reivindicación 2, en el que el dominio antigénico comprende el dominio extraviral de una proteína de fusión viral.
4. El polipéptido de la reivindicación 3, en el que la proteína de fusión se selecciona del grupo que consiste en: la 10 proteína Env de un retrovirus; la proteína F de un paramixovirus; la proteína Gp del virus Ébola; la proteína hemaglutinina del virus de la gripe; las proteínas de pico de un coronavirus; la glucoproteína del virus de la rabia (RVG); la proteína de fusión de un arbovirus; las proteínas de fusión de un togaviridae; la proteína de fusión de un flaviviridae; la proteína de fusión de un alfavirus; la proteína E del virus del dengue; la proteína E del virus de la hepatitis C; la proteína E del virus de la fiebre amarilla; la proteína E del virus de la encefalitis japonesa; la proteína E del virus del Nilo 15 Occidental; la proteína E del virus de la encefalitis transportada por garrapatas (TBE); la proteína de fusión del virus del sarampión; la proteína de pico E1 del virus del Bosque de Semliki; la proteína de fusión de un bunyaviridae; y la proteína de fusión de un arenaviridae.
5. El polipéptido de la reivindicación 4, en el que la proteína de fusión es la proteína de la envoltura del VIH.
6. El polipéptido de la reivindicación 4, en el que el dominio antigénico es del producto de escisión N terminal del 20 polipéptido de fusión.
7. El polipéptido de la reivindicación 2, en el que el dominio antigénico es de una proteína de superficie de una bacteria seleccionada del grupo que consiste en: Neisseria meningitidis; Neisseria gonorrhoeae; Streptococcus pneumoniae; Streptococcus pyogenes; Streptococcus agalactiae; Staphylococcus aureus; Haemophilus influenza; Moraxella catarrhalis; Helicobacter pylori; Chlamydia trachomatis; y Chlamydia pneumoniae. 25
8. El polipéptido de cualquier reivindicación precedente, en el que el dominio transmembrana es de una proteína transmembrana bacteriana.
9. El polipéptido de cualquier reivindicación precedente, en el que los dominios (b) y (c) son de NadA de Neisseria meningitidis.
10. El polipéptido de cualquier reivindicación precedente, en el que al menos uno de (a) y (c) tiene un origen 30 eucariota y al menos otro de (a) y (c) tiene un origen procariota.
11. El polipéptido de la reivindicación 9, en el que el dominio (c) es de un procariota y el dominio (a) es de un virus de eucariota.
12. Un polipéptido que comprende: (a) un dominio antigénico de la proteína de fusión viral de un virus de eucariota con envoltura; (b) un dominio de superenrollamiento de NadA de Neisseria meningitidis y (c) un dominio transmembrana 35 de la misma adhesina bacteriana que (b).
13. Un polipéptido de la fórmula NH2-A-B-C-D-E-F-G-H-COOH en el que: A es una secuencia líder opcional; B es una secuencia de engarce opcional; C es una secuencia antigénica; D es una secuencia de engarce opcional; E es una secuencia de superenrollamiento de NadA de Neisseria meningitidis, es una secuencia de engarce opcional; G es una secuencia transmembrana; y H es una cola citoplasmática opcional. 40
14. Ácido nucleico que codifica el polipéptido de cualquier reivindicación precedente.
15. Una proteína oligomérica, que comprende polipéptidos oligomerizados de una cualquiera de las reivindicaciones 2 a 13.
16. La proteína oligomérica de la reivindicación 15, que es un trímero.
17. Una célula huésped, expresando la célula huésped el polipéptido de una cualquiera de las reivindicaciones 1 a 45 13 en su superficie.
18. La célula huésped de la reivindicación 17, en la que la célula está seleccionada del grupo que consiste en: Escherichia coli, Bacillus subtilis, Vibrio cholerae, Salmonella typhi, Salmonella typhimurium, Neisseria lactamica,
Neisseria cinerea, Mycobacterium, Shigella spp., Yersinia enterocolitica, y Listeria monocytogenes.
19. Un preparación de membrana derivada de la célula huésped de la reivindicación 17 o reivindicación 18 que comprende el polipéptido de las reivindicaciones 1-13.
20. Una composición farmacéutica que comprende el polipéptido de una cualquiera de las reivindicaciones 1 a 13, el ácido nucleico de la reivindicación 14, la proteína de la reivindicación 15 o reivindicación 16, la célula huésped de la 5 reivindicación 17 o reivindicación 18 o la preparación de membrana de la reivindicación 19.
21. La composición de la reivindicación 20 para su uso en la inducción de una respuesta inmune en un mamífero.
Patentes similares o relacionadas:
Cadena ligera de enteroquinasa modificada, del 22 de Julio de 2020, de NOVO NORDISK A/S: Un análogo de la cadena ligera de la enteroquinasa bovina que comprende una secuencia de aminoácidos establecida en la SEQ ID NO: 1, en donde dicho análogo comprende […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Antígenos de coagulasa estafilocócica y métodos para su uso, del 13 de Mayo de 2020, de UNIVERSITY OF CHICAGO: Una composición inmunógena que comprende al menos dos dominios 1-2 de coagulasa estafilocócica diferentes, en donde cada uno de los al menos dos dominios […]
Reconocimiento de unión a diana celular mediante un agente bioactivo usando transferencia de energía de resonancia de bioluminiscencia intracelular, del 6 de Mayo de 2020, de PROMEGA CORPORATION: Un sistema de ensayo que comprende: (a) una biblioteca de agentes bioactivos, cada uno de los cuales está fijado a un fluoróforo; (b) una diana celular fusionada a […]
Etiqueta de epítopo y método de detección, captura y/o purificación de polipéptidos etiquetados, del 15 de Abril de 2020, de ChromoTek GmbH: Péptido epítopo aislado que tiene de 12 a 25 aminoácidos, en donde la secuencia de aminoácidos comprende una secuencia según se define en SEQ ID NO: 32 (X1X2RX4X5AX7SX9WX11X12), […]
Utilización diagnóstica de un polipéptido de fusión que comprende una proteína vírica y un enzima MGMT, del 15 de Abril de 2020, de INSTITUT PASTEUR: Utilización in vitro de un polipéptido de fusión que comprende una proteína vírica y i) el enzima 6-metilguanina-ADN-metiltransferasa (MGMT, EC 2.1.1.63) o un homólogo […]