Método para mejorar la estabilidad del ADN lineal en sistemas de transcripción/traducción in vitro, exentos de células.
Método para la mejora de la estabilidad del ADN lineal corto frente a las exonucleasas en sistemas de trans- cripción/traducción in vitro exentos de células,
empleando exonucleasas que contienen lisados o en sistemas de expresión de proteína celular que contienen exonucleasas en donde la estabilidad del ADN lineal corto se mejora añadiendo ADN lineal que no se transcribe en dicho sistema de trans- cripción/traducción in vitro.
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E02008545.
Solicitante: F. HOFFMANN-LA ROCHE AG.
Inventor/es: HOFFMANN, THOMAS, HEINDL, DIETER, METZLER, THOMAS, MUTTER, WOLFGANG, DR., WATZELE, MANFRED, DR., NEMETZ, CORDULA, DR.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/68 C12N 15/00 […] › Estabilización del vector.
PDF original: ES-2222417_T3.pdf
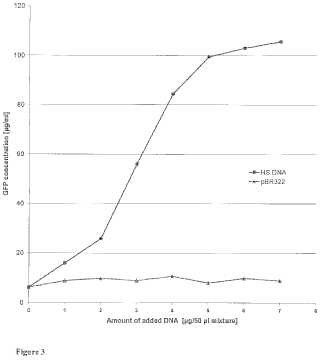
Fragmento de la descripción:
Método para mejorar la estabilidad del ADN lineal en sistemas de transcripción/traducción in vitro, exentos de células.
La presente invención se refiere a un método para mejorar la estabilidad del ADN lineal corto, frente a las exonucleasas en sistemas de transcripción/traducción in vitro, exentos de células, empleando lisados que contienen exonucleasas o en sistemas celulares en donde la estabilidad del ADN lineal corto se mejora añadiendo ADN lineal inespecífico.
La transcripción/traducción in vitro, dependiente del ADN, exento de células, trabaja completamente bien en la práctica con respecto a la expresión del ADN circular de doble hélice y con respecto a la expresión del ADN lineal largo. Las tentativas de una expresión de fragmentos de ADN lineal más corto ha tenido únicamente un éxito limitado. Cuanto más pequeño es el ADN empleado más difícil ha sido obtener apreciables cantidades de producto génico. Se estableció que estas dificultades eran debidas a la presencia de exonucleasas. Por lo tanto se demostró que la exonucleasa V es responsable de la degradación del ADN lineal cuando los lisados S30 de E. coli fueron transcritos y traducidos in vitro. La exonucleasa V se compone de tres subunidades (los productos génicos recB, recC, recD) . Esta exonucleasa escinde el ADN lineal a partir de su extremo 3'.
Se intentó remediar este problema mediante la mutación de las subunidades de esta exonucleasa con el fin de eliminar la actividad lítica. Yang et al., (1980) PNAS vol. 77, nº 12 pp 7029-7033 describen una síntesis mejorada de proteína partiendo de moldes de ADN lineal empleando la cepa de E. coli CF300 después de la deleción de la exonucleasa V (eliminación de los genes recB, recC, de la cepa recB21) .
Leavel Basset et al., (1983) , J. Bacteriol. vol. 156, nº 3, pp. 1359-1362, mutaron adicionalmente los genes de la RNasa y polinucleótido fosforilasa (rna-19 pnp-7) en la cepa recB21 (cepa CLB7) y lograron una expresión de la proteína significativamente mayor con moldes de ADN lineal después de un período de una hora de incubación.
Lesley et al., (1991) , J. Biol.. Chem. vol. 266, nº 4, pp 2632-2638, emplea una cepa recD BL21 deficiente en exonucleasa V, a la cual ha dado el nombre de cepa SL119, y describe por primera vez el método de síntesis de la proteína in vitro partiendo de moldes generados por PCR. Se dispone comercialmente de lisados de la cepa SL 119 (Promega) para la transcripción/traducción in vitro empleando moldes lineales.
Sin embargo, una desventaja de las medidas descritas más arriba es que todos estos mutantes crecen muy despacio y también que los lisados obtenidos de estas cepas tienen una velocidad de síntesis significativamente más pobre. Aparentemente esta exonucleasa juega un importante papel en el metabolismo de las bacterias. Por lo tanto, parece ser importante emplear lisados o cultivos celulares en los cuales la exonucleasa esté presente.
Otra medida concebible para proteger no obstante los ácidos nucleicos contra la degradación exonucleolítica, es el modificar los ácidos nucleicos bien sea protegiendo ambos extremos, o bien sea empleando bloques de construcción nucleótida modificados, como se describe en la literatura para ácidos nucleicos en el campo anti-sentido y en las siguientes citas:
Las moléculas de ADN/ARN monocatenarias pueden protegerse protegiendo los extremos con grupos alquilo y modificando las bases. Pandolfi et al. (1999) Nucleosides & Nucleotides, 18 (9) , 2051-2069. Verheijen et al., (2000) Bioorganic & Medicinal Chemistr y Letters 10, 801-804, muestran una mayor estabilidad de las moléculas de ADN monocatenarias protegiendo los extremos con enlaces 4-hidroxi-N-acetilprolinol, L-serinol ó mediante 3'-3'-fosfodiéster. Los enlaces fosforotioato puros o mixtos y oligonucleótidos químicamente modificados p. ej., metil-fosfonatos y fosforamidatos son más estables y se degradan más lentamente por las exonucleasas Kandimalla et al., NAR (1997) vol 25, nº 2, pp. 370-378. Tohda et al., (1994) Journal of Biotechnology (Revista de Biotecnología
) 34 (1994) 61-69 muestran que el ARN que contiene fosforo- tioatos es más estable frente a las nucleasas y por lo tanto tiene una mayor eficiencia de traducción. Sin embargo, por regla general solamente podrían producirse pequeñas cantidades de proteína. Tang et al., (1993) NAR, vol. 21 nº 11 pp 2279-2735 muestran que las estructuras de bucle en horquilla protegen el extremo 3' de los ADN monocatenarios contra la degradación exonucleolítica. Hirao et al., (1993) FEBS, vol. 321, nº 2, 3, 169-172 muestran que la horquilla que forma el fragmento d de ADN (GCGAAGC) , es extremadamente resistente a las nucleasas de los extractos de E. coli. Yoshizawa et al., (1994) NAR, vol. 22 nº 12 pp. 2217-2221 describen que una estabilización del extremo 3' de ARNm por hibridación con la misma horquilla da como resultado un aumento de 200 veces la eficiencia de la traducción in vitro con extractos de E. coli. Good y Nielsen (1998) PNAS USA 95, 2073-6 muestran que las moléculas sintéticas que contienen bases que están acopladas a un esqueleto de péptidos (ácido péptido nucleico, PNA) son resistentes a la escisión hidrolítica en extractos de E. coli y pueden ser empleados como molécula sin sentido. Burdick y Emlen (1985) , J. Inmunology 135, 2593-7 describen que en inmunocomplejos ADN anti-ADN, las moléculas de IgG pueden proteger el ADN que está unido a la misma, de la degradación nucleolítica. La patente EP 0 967 274 A2 describe métodos para la preparación de moléculas de ADN lineal de doble cadena en forma de pesa. En este método se escinde un plásmido con enzimas de restricción y las moléculas resultantes de doble cadena cerradas no covalentemente, se modifican a continuación para formar construcciones en forma de pesa mediante la digestión de los extremos con una endonucleasa de restricción que forma colgantes monocatenarios y a continuación ligando los oligómeros en forma de horquilla emparejados, sobre los colgantes monocatenarios resultantes. Esta construcción tiene una mayor estabilidad frente a las exonucleasas de la T7 ADN polimerasa.
Una desventaja de estas medidas es que la síntesis de estos bloques de construcción de nucleótidos modificados, es a menudo muy complicada. Por una parte, la preparación de los nucleótidos modificados es complicada y cara. Por otro lado, se necesitan unos pasos del procedimiento que consumen un tiempo adicional, para incorporar la modificación. Además, pueden surgir dificultades empleando los ácidos nucleicos modificados como moldes. En el caso de los fosfotioatos una desventaja es por ejemplo que la síntesis es muy complicada y se forma una mezcla de diastereómeros, la cual, después de la incorporación, no es óptimamente adecuada como molde para la transcripción o la traducción.
Otros sistemas de expresión exentos de células sin estrategias de protección están descritas en la técnica anterior: En la patente US 5571690, Hecht describe un método para la síntesis exenta de células de una proteína, partiendo de un molde que ha sido generado en una reacción PCR. En este método, se amplifica la secuencia completa de genes incluyendo la región promotora del fago de un plásmido. Después, en una transcripción in vitro, se emplea un lisado de reticulocitos de conejo para la traducción. Con este método ha sido posible producir 57 μg/ml de una proteína empleando ARNm que ha sido modificado después de la transcripción con un 5'CAP. Martemyanov et al., (1997) FEBS Lett. 414, 268-270 emplean un extracto S30 de E. coli para la síntesis exenta de células de una proteína partiendo de un molde el cual ha sido generado en una reacción PCR de 2 pasos. En este método el gen diana se amplifica en primer lugar en una reacción PCR con ayuda de dos cebadores oligonucleótidos gen-específicos y a continuación se someten a una segunda reacción PCR en la cual se emplea un llamado megacebador para fusionar el promotor T7 y el sitio de unión ribosómica con el gen amplificado. Sólo fué posible producir cantidades radioactivamente detectables de proteína. Yang y col., (2000) , J. Bacteriol. 182, 295-302 emplean un extracto de E. coli para demostrar la síntesis exenta de células de una proteína que parte de un molde que ha sido generado en una reacción PCR. En este método fue solamente posible producir cantidades de proteína radioactivamente detectables. Nakano et al., (1999) Biotechnol & Bioeng. 64, 194-199 emplean un extracto S30 de E. coli en un reactor de fibra hueca para... [Seguir leyendo]
Reivindicaciones:
1. Método para la mejora de la estabilidad del ADN lineal corto frente a las exonucleasas en sistemas de transcripción/traducción in vitro exentos de células, empleando exonucleasas que contienen lisados o en sistemas de expresión de proteína celular que contienen exonucleasas en donde la estabilidad del ADN lineal corto se mejora añadiendo ADN lineal que no se transcribe en dicho sistema de transcripción/traducción in vitro.
2. Método según se ha reivindicado en la reivindicación 1, en donde el ADN no transcrito en dicho sistema es ADN cortado de esperma de arenque, esperma de salmón o timo de ternera.
3. Método según se ha reivindicado en una de las reivindicaciones 1 ó 2, en donde el ADN no transcrito en dicho sistema es ADN cortado de esperma de arenque.
4. Método según se ha reivindicado en una de las reivindicaciones 1 a 3 en donde los sistemas de transcripción/traducción in vitro exentos de células son sistemas de transcripción/traducción in vitro acoplados exentos de células.
5. Método según se ha reivindicado en una de las reivindicaciones 1 a 4, en donde los lisados que contienen las exonucleasas son lisados procarióticos.
6. Método según se ha reivindicado en una de las reivindicaciones 1 a 5, en donde los lisados que contienen las exonucleasas son lisados de E. coli.
7. Método según se ha reivindicado en una de las reivindicaciones 1 a 4, en donde los lisados que contienen las exonucleasas son lisados eucarióticos.
8. Método según se ha reivindicado en la reivindicación 7, en donde los lisados que contienen las exonucleasas son lisados de células eucarióticas que se han desarrollado en un cultivo celular.
9. Método según se ha reivindicado en una de las reivindicaciones 1 a 8, en donde el ADN no transcrito en dicho sistema se añade a una concentración de 1 a 200 μg/ml por mezcla de traducción.
10. Método según se ha reivindicado en una de las reivindicaciones 1 a 9, en donde el ADN no transcrito en dicho sistema se añade a una concentración de 10 a 100 μg/ml por mezcla de traducción.
11. Método según se ha reivindicado en una de las reivindicaciones 1 a 10, en donde el ADN no transcrito en dicho sistema tiene una longitud de por lo menos 10 bp.
12. Empleo de un método como se ha reivindicado en una de las reivindicaciones 1-11, para expresar una proteína.
13. Empleo del método según se ha reivindicado en una de las reivindicaciones 1-11, para expresar una proteína, en donde el gen deseado se amplifica directamente a partir de un banco de genes o fracción de ARN mediante una PCR o RT-PCR.
14. Empleo del método según se ha reivindicado en una de las reivindicaciones 1-11, para expresar una proteína, en un reactor CFCF y CECF.
15. Kit que consiste en uno o varios recipientes que contienen los siguientes componentes:
16. Solución reactivo que contiene:
Patentes similares o relacionadas:
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Método para romper un ácido nucleico y añadir un adaptador por medio de transposasa y reactivo, del 1 de Julio de 2020, de MGI Tech Co., Ltd: Un metodo para romper un acido nucleico y anadir un adaptador por medio de una transposasa, que comprende las siguientes etapas: interrumpir […]
Vacuna subunitaria contra Mycoplasma spp., del 1 de Julio de 2020, de Agricultural Technology Research Institute: Una composición para prevenir una infección por Mycoplasma spp., que comprende: un principio activo, que comprende una proteína de PdhA; y un adyuvante […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Preparación de bibliotecas de ácido nucleico marcado usando protocolo de adición en un solo tubo, del 13 de Mayo de 2020, de ILLUMINA, INC: Un método para preparar una biblioteca de fragmentos de ácido nucleico marcados que comprende: (a) poner en contacto una célula individual […]
Proteínas de captura de la superficie celular recombinantes, del 13 de Mayo de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para detectar y aislar células que producen altos niveles de una proteína heterodimérica que tiene una primera subunidad y una segunda subunidad, […]
Etiquetado y evaluación de una secuencia diana, del 13 de Mayo de 2020, de RhoDx, Inc: Un método para modificar un ácido nucleico, que comprende: (a) poner en contacto un ácido nucleico de cadena sencilla con una actividad de transferasa […]