Secuencias consenso codificantes de cánceres de mama y colorrectales humanos.
Un procedimiento de diagnóstico de cáncer de mama en un ser humano,
que comprende las etapas de:
determinar en una muestra de ensayo con respecto a una muestra normal del ser humano, una mutación somática humana en un gen ABCA3 o su ADNc o proteína codificados; e identificar la muestra como cáncer de mama cuando se determina la mutación somática.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2007/017866.
Solicitante: JOHNS HOPKINS UNIVERSITY.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 100 NORTH CHARLES STREET, 5TH FLOOR BALTIMORE, MD 21201 ESTADOS UNIDOS DE AMERICA.
Inventor/es: VOGELSTEIN, BERT, KINZLER, KENNETH, W., SJOBLOM,TOBIAS, JONES,SIAN, PARSONS,D. WILLIAMS, WOOD,LAURA D, LIN,JIMMY, BARBER,THOMAS, MANDELKER,DIANA, VELCULESU,VICTOR E.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2421354_T3.pdf
Fragmento de la descripción:
Secuencias consenso codificantes de cánceres de mama y colorrectales humanos La presente invención se realizó usando fondos de subvención del gobierno de los Estados Unidos. Bajo el término de las subvenciones, el gobierno de los Estados Unidos conserva determinados derechos en la invención. Las subvenciones utilizadas incluyen las subvenciones NIH CA 121113, CA 43460, CA 57345, CA 62924, GM 07309, RR 017698, P30-CA43703 y CA109274 y DAMD17-03-1-0241 del Departament of Defense grant
Campo técnico de la invención La presente invención se refiere al área de caracterización del cáncer. En particular se refiere a cánceres de mama y colorrectales.
Antecedentes de la invención Es un hecho ampliamente aceptado que el cáncer humano es una enfermedad genética ocasionada por la acumulación secuencial de mutaciones en oncogenes y genes supresores tumorales (1) . Estas mutaciones específicas de tumores (es decir, somáticas) proporcionan pistas con respecto a los procesos celulares causantes de tumorigénesis y se ha demostrado que son útiles con fines de diagnóstico y terapéuticos. Sin embargo, hasta ahora, solo se ha analizado una pequeña fracción de los genes y se desconoce el número y el tipo de alteraciones sensibles al desarrollo de tipos tumorales (2) . En el pasado, las selecciones de genes seleccionados para análisis mutacional en cáncer se han guiado por la información de estudios de ligamiento en familias propensas al cáncer, identificación de anomalías cromosómicas en tumores o atributos funcionales conocidos de genes individuales o familias de genes (2-4) . Se ha encontrado que una gran cantidad de genes se sobreexpresan en cáncer [50]. Además, la sobreexpresión de transportadores ABC como ABCA3 se ha relacionado con la resistencia a fármacos en células tumorales humanas [51-53]. La determinación de la secuencia del genoma humano junto con mejoras en la secuenciación y estrategias bioinformáticas han hecho ahora que sea posible, en principio, examinar el genoma de células cancerosas de una manera exhaustiva y objetiva. Dicha estrategia no solo proporciona los medios para descubrir otros genes que contribuyen a la tumorigénesis sino que también conduce a conocimientos mecanicistas que solo son obvios a través de una perspectiva de sistemas biológicos. El análisis genético exhaustivo de cánceres humanos podría conducir al descubrimiento de un conjunto de genes, relacionados entre sí a través de un fenotipo compartido, orientado a la importancia de procesos celulares específicos o rutas específicas.
Existe una necesidad continua en la técnica de identificar genes y modelos de mutaciones génicas útiles para identificar y estratificar cánceres de pacientes individuales.
Sumario de la invención De acuerdo con una realización de la invención se proporciona un procedimiento para el diagnóstico del cáncer de mama en un ser humano. En una muestra de ensayo se determina una mutación somática en el gen ABCA3 o su ADNc codificado o proteína codificada con respecto a una muestra normal del ser humano. La muestra se identifica como cáncer de mama cuando se determina la mutación somática.
También se describe un procedimiento para el diagnóstico del cáncer colorrectal en un ser humano. En una muestra de ensayo se determina una mutación somática en un gen o su ADNc codificado o proteína codificada con respecto a una muestra normal del ser humano. El gen se selecciona del grupo que consiste en los indicados en la Figura 14 (Tabla S6) . La muestra se identifica como cáncer colorrectal si se determina la mutación somática.
Se proporciona un procedimiento para estratificar cánceres de mama para ensayar agentes terapéuticos anticancerosos candidatos o conocidos. Se determina una firma mutacional de genes CAN para un cáncer de mama determinando al menos una mutación somática en una muestra de ensayo con respecto a una muestra normal de un ser humano. La al menos una mutación somática está en el gen ABCA3. Se forma un primer grupo de cánceres de mama que tienen la firma mutacional de genes CAN. La eficacia de un agente terapéutico anticanceroso candidato o conocido del primer grupo se compara con la eficacia de un segundo grupo de cánceres de mama que tiene una firma mutacional de genes CAN diferente. Se identifica una firma mutacional de genes CAN que se correlaciona con una eficacia aumentada o disminuida del agente terapéutico anticanceroso candidato o conocido con respecto a otros grupos.
Se describe un procedimiento para estratificar cánceres colorrectales para ensayar agentes terapéuticos anticancerosos candidatos o conocidos. Se determina una firma mutacional de genes CAN para un cáncer colorrectal determinando al menos una mutación somática en una muestra de ensayo con respecto a una muestra normal del ser humano. La al menos una mutación somática está uno o más genes seleccionados del grupo que consiste en los genes indicados en la Figura 14 (Tabla S6) . Se forma un primer grupo de cánceres colorrectales que tienen la firma mutacional de genes CAN. La eficacia de agente terapéutico anticanceroso candidato o conocido del primer grupo se compara con la eficacia de un segundo grupo de cánceres colorrectales que tienen una firma mutacional de genes CAN diferente. Se identifica una firma mutacional de genes CAN que se correlaciona con una eficacia aumentada o disminuida del agente terapéutico anticanceroso candidato o conocido con respecto a otros grupos.
En el presente documento se describe un procedimiento para caracterizar un cáncer de mama en un ser humano. En una muestra de ensayo se determina una mutación somática en un gen o su ADNc codificado o proteína codificada con respecto a una muestra normal del ser humano. El gen se selecciona del grupo que consiste en los genes indicados en la Figura 13 (Tabla S5) .
Se describe otro procedimiento para la caracterización de un cáncer colorrectal en un ser humano. En una muestra de ensayo se determina una mutación somática en un gen o su ADNc codificado o proteína codificada con respecto a una muestra normal del ser humano. El gen se selecciona del grupo que consiste en los genes indicados en la Figura 14 (Tabla S6) .
Breve descripción de las figuras
Figura 1A y 1B. Esquema de Exploraciones de Descubrimiento y Validación de Mutaciones.
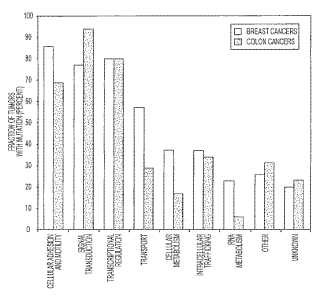
Figura 2. Frecuencia de mutación de los grupos de genes CAN. Los genes CAN se agruparon por función usando grupos de Ontología de Genes, dominios INTERPRO y bibliografía disponible. Las barras indican la fracción de tumores (35 de mama o 35 colorrectales) al menos con un gen mutado en el grupo funcional.
Figura 3. (Fig. S1) Frecuencias de mutación de codón. Barras blancas, codones CCDS (n=7.479.318 en 13.023 genes) ; barras rojas, codones afectados por mutaciones de sustituciones de bases en cánceres de mama (n=789) ; barras azules, codones afectados por mutaciones de sustituciones de bases en cánceres colorrectales (n=669) .
Figura 4. (Fig. S2) genes CCDS excluidos del análisis. Ciento treinta y cuatro transcritos de 119 genes que se emparejaban estrechamente con más de un locus genómico (círculo grande) y/o que estaban localizados en el cromosoma Y (círculo pequeño) se excluyeron del análisis.
Figura 5. (Tabla 1.) Resumen de mutaciones somáticas Figura 6. (Tabla 2) Espectro de sustituciones de una sola base Figura 7. (Tabla 3.) Clasificación funcional de genes CAN*
Figura 8. (Tabla S1) Cebadores usados para amplificación y secuenciación por PCR (página 1 solo de 1333; se dispone públicamente de todas las secuencias de cebadores en un archivo transferible (1133427_som_Tablas.zip) en el sitio web del journal Science (www.sci-encemag.org) con Material de apoyo Online localizado en la página web /cgi/content/full/sci;1133427/DC1)
Fig. 9. (Tabla S2A.) Características de las muestras de cáncer colorrectal.
Fig. 10. (Tabla S2B.) Características de las muestras de cáncer de mama.
Fig. 11. (Tabla S3.) Distribución de mutaciones en cánceres individuales.
Fig. 12. (Tabla S4.) Mutaciones somáticas identificadas en cánceres de mama o colorrectales.
Figura 13. (Tabla S5.) Genes CAN de mama.
Figura 14. (Tabla S6.) Genes CAN colorrectales.
Descripción detallada de la invención Los autores de la invención han desarrollado procedimientos para la caracterización de cánceres de mama basándose en firmas genéticas. Estas firmas comprenden el gen ABCA3 que está mutado en cáncer de mama. Las firmas pueden usarse como un medio diagnóstico, pronóstico, identificación de metástasis, estratificación para estudios farmacológicos y para asignar un tratamiento apropiado.
De acuerdo con la presente invención una mutación somática... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento de diagnóstico de cáncer de mama en un ser humano, que comprende las etapas de:
determinar en una muestra de ensayo con respecto a una muestra normal del ser humano, una mutación somática humana en un gen ABCA3 o su ADNc o proteína codificados; e identificar la muestra como cáncer de mama cuando se determina la mutación somática.
2. Un procedimiento para estratificar cánceres de mama para ensayar agentes terapéuticos anticancerosos candidatos o conocidos, que comprende las etapas de:
determinar una firma mutacional de genes CAN para un cáncer de mama determinando al menos una mutación somática en una muestra de ensayo con respecto a una muestra normal de un ser humano, en el que la al menos una mutación somática está en un gen ABCA3; formar un primer grupo de cánceres de mama que tengan la firma mutacional de genes CAN; comparar la eficacia de un agente terapéutico anticanceroso candidato o conocido sobre el primer grupo con la eficacia sobre un segundo grupo de cánceres de mama que tiene una firma mutacional de genes CAN diferente; identificar una firma mutacional de genes CAN que se correlacione con una eficacia aumentada o disminuida del agente terapéutico anticanceroso candidato o conocido con respecto a otros grupos.
3. El procedimiento de la reivindicación 2 en el que la firma mutacional de genes CAN comprende al menos 2 genes, seleccionados de la Figura 13 (Tabla S5) .
4. El procedimiento de la reivindicación 2 en el que la muestra de ensayo es una muestra de tejido mamario, y en el que la muestra normal es una muestra de tejido mamario.
5. Un procedimiento de caracterización de un cáncer de mama en un ser humano, que comprende las etapas de:
determinar en una muestra de ensayo con respecto a una muestra normal del ser humano, una mutación somática en un gen ABCA3 o su ADNc o proteína codificados.
6. El procedimiento de la reivindicación 1 o la reivindicación 5 en el que la muestra de ensayo es una muestra de tejido mamario o una metástasis de cáncer de mama sospechosa, y en el que la muestra normal es una muestra de tejido mamario.
7. El procedimiento de la reivindicación 1 o la reivindicación 5, en el que la mutación se selecciona de las mostradas en la Fig. 12 (Tabla S4) .
8. El procedimiento de la reivindicación 2 en el que la al menos una mutación se selecciona de las mostradas en la Fig. 12 (Tabla S4) .
9. El procedimiento de la reivindicación 1, 2 o 5 en el que la mutación provoca una sustitución L290M.
10. El procedimiento de la reivindicación 1, 2 o 5 en el que la mutación provoca una sustitución H1069Q.
11. El procedimiento de la reivindicación 1, 2 o 5 en el que la mutación provoca una sustitución E801D.
Tránsito intracelular
Cuando los registros CCDS múltiples derivan del mismo gen, los cebadores de exones compartidos se enumeran con el registro CCDS con el número ordinal más bajo. Los exones codificantes mayores de 350 pb se amplificaron y se secuenciaron con pares de cebadores múltiples. Posición del extremo 5' del cebador PCR directo con respecto a la primera base del exón. indica el cebador de secuenciación universa.
5. GTAAAACGACGGCCAGT3'. Los números sin firma indican la posición del extremo 3' del cebador PCR inverso con respecto a la primera base del exón. Los signos positivos indican la posición el extremo 3' del cebador PCR inverso con respecto a la última base de este exón. El primer cebador no cumplió el criterio de calidad de que 90 % de bases en la región diana tenga una puntuación de calidad Phred de al menos 20 en tres cuartas partes de las muestra tumorales analizadas en la Exploración de Descubrimiento.
Fig. 9. Tabla S2A Características de las muestras de cáncer colorrectal
ID Edad Sexo Procedencia Localización del tumor Estado Tipo de muestra Exploración
del paciente del tejido original clinico
(años)
Co74 35 F Metástasis hepática Colon sigmoide IV Línea celular Descubrimiento
Co92 46 F Metástasis hepática Colon del lado derecho IV Línea celular Descubrimiento
Co108 77 F Metástasis hepática Colon del lado derecho IV Línea celular Descubrimiento
Mx22 66 F Metástasis hepática Desconocida IV Xenoinjerto Descubrimiento
Mx27 74 F Metástasis hepática Colon del lado derecho IV Xenoinjerto Descubrimiento
Mx30 62 F Metástasis hepática Colon sigmoide IV Xenoinjerto Descubrimiento
Mx32 56 F Metástasis hepática Colon del lado derecho IV Xenoinjerto Descubrimiento
Mx3B 67 M Metástasis hepática Recto IV Xenoinjerto Descubrimiento
Mx41 57 M Metástasis hepática Colon del lado derecho IV Xenoinjerto Descubrimiento
Mx42 72 M Metástasis hepática Colon del lado izquierdo IV Xenoinjerto Descubrimiento
Mx43 72 M Metástasis hepática Colon sigmoide IV Xenoinjerto Descubrimiento
Co79 81 M Metástasis de nodulo linfático Colon del lado derecho IV Línea celular Validación
Co82 80 F Tumor colorrectal primario Colon del lado derecho IV Línea celular Validación
Co84 40 M Metástasis de nodulo linfático Colon del lado derecho III Línea celular Validación
Co94 46 M Metástasis de nodulo linfático Recto IV Línea celular Validación
Mx3 64 M Tumor colorrectal primario Colon del lado derecho IV Xenoinjerto Validación
Mx8 65 M Tumor colorrectal primario Rectal IV Xenoinjerto Validación
Mx26 45 F Metástasis hepática Colon del lado derecho IV Xenoinjerto Validación
Mx29 51 M Metástasis hepática Colon del lado izquierdo IV Xenoinjerto Validación
Mx31 50 M Metástasis hepática Recto IV Xenoinjerto Validación
Mx34 82 F Metástasis de nodulo linfático Colon del lado derecho IV Xenoinjerto Validación
Mx35 57 F Metástasis pulmonar Recto IV Xenoinjerto Validación
Mx40 75 F Metástasis de nodulo linfático Colon del lado derecho III Xenoinjerto Validación
Mx45 67 F Metástasis hepática Colon transverso IV Xenoinjerto Validación
HxO05 62 F Metástasis hepática Flexura esplénica IV Xenoinjerto Validación
Hx169 68 M Metástasis hepática Recto IV Xenoinjerto Validación
Hx172 69 F Metástasis hepática Desconocida IV Xenoinjerto- Validación
HX174 73 F Metástasis hepática Colon sigmoide IV Xenoinjerto Validación
Hx185 39 F Metástasis hepática Recto Colon sigmoide IV Xenoinjerto Validación
Hx188 60 M Metástasis hepática Lado izquierdo IV Xenoinjerto Validación
Hx189 72 M Metástasis hepática Lado izquierdo IV Xenoinjerto Validación
Hx190 73 M Metástasis hepática Lado derecho IV Xenoinjerto Validación
Hx206 54 M Metástasis hepática Recto IV Xenoinjerto Validación
Hx218 56 F Metástasis hepática Lado izquierdo IV Xenoinjerto Validación
Hx219 69 M Metástasis hepática Ciego IV Xenoinjerto Validación
Hx220 83 F Metástasis hepática Lado derecho IV Xenoinjerto Validación
Hx223 45 F Metástasis hepática Recto sigmoide IV Xenoinjerto Validación
Fig. 10.Tabla S2B. Caracerísticas de las muestras de cancer de mama
ID Edad del paciente Tipo de Procedencia Estado Estado Estado Estado Tipo de muestra Exploración
(años) adenocarcinoma del tejido RE* RP* Mer-2/neu*
HCC1008 67 Ductal Nódulo linfáticoe IIA - - +++ Línea celular Descubrimiento
HCC1BS4 61 Ductal Tumor mamario primario IIA - - N/D Línea celular Descubrimiento
HCC38 SO Ductal Tumor mamario primario IIB - - - Línea celular Descubrimiento
HCC1143 52 Ductal Tumor mamario primario IIA - - - Línea celular Descubrimiento
HCC1187 41 Ductal Tumor mamario primario IIA - - - Línea celular Descubrimiento
HCC139S 43 Ductal Tumor mamario primario 1 - - - Línea celular Descubrimiento
HCC1S99 44 Ductal Tumor mamario primario IIIA - - - Línea celular Descubrimiento
HCC1837 24 Ductal Tumor mamario primario IIB - - - Línea celular Descubrimiento
HCC21S7 48 Ductal Tumor mamario primario IIIA - + ++ Línea celular Descubrimiento
HCC2218 38 Ductal Tumor mamario primario IIIA - - +++ Línea celular Descubrimiento
Hs57BT 74 Carcinosarcoma Tumor mamario primario N/D - - + Línea celular Descubrimiento
HCC2713 35 Ductal Tumor mamario primario I - N/D ++ Línea celular Validación
BB1T 51 Ductal Tumor mamario primario IIIC - - - Tejido tumoral microdiseccionado Validación
BB2T 51 Ductal Tumor mamario primario IIA + - ++ Tejido tumoral microdiseccionado Validación
BB3T 56 Ductal Tumor mamario primario IIA + + +++ Tejido tumoral microdiseccionado Validación
BB4T 52 Lobulillar Tumor mamario primario IIIA + + ++ Tejido tumoral microdiseccionado Validación
BB5T 42 Ductal Tumor mamario primario IIIA + + - Tejido tumoral microdiseccionado Validación
BB7T 59 Ductal Tumor mamario primario IIA + + +++ Tejido tumoral microdiseccionado Validación
BB9T 47 Ductal Tumor mamario primario IIA + + +++ Tejido tumoral microdiseccionado Validación
BBIOT 55 Ductal Tumor mamario primario IIB + + + Tejido tumoral microdiseccionado Validación
BB12T 58 Ductal Tumor mamario primario IIIA - - + Tejido tumoral microdiseccionado Validación
BB13T 63 Ductal Tumor mamario primario IIIC - - - Tejido tumoral microdiseccionado Validación
BB14T 74 Ductal Tumor mamario primario IIA + + - Tejido tumoral microdiseccionado Validación
BB15T 79 Ductal Tumor mamario primario IIB + + + Tejido tumoral microdiseccionado Validación
BB16T 40 Ductal Tumor mamario primario IIB + + + Tejido tumoral microdiseccionado Validación
BB18T 53 Ductal Tumor mamario primario IIA + + - Tejido tumoral microdiseccionado Validación
BB20T 83 Ductal Tumor mamario primario IIA + + - Tejido tumoral microdiseccionado Validación
BB21T 77 Ductal Tumor mamario primario IIIA + + - Tejido tumoral microdiseccionado Validación
BB22T SO Ductal Tumor mamario primario IIIC + + + Tejido tumoral microdiseccionado Validación
BB23T 46 Ductolobulillar Tumor mamario primario IIIC + - + Tejido tumoral microdiseccionado Validación
BB24T 66 Ductal Tumor mamario primario IIA - - + Tejido tumoral microdiseccionado Validación
BB27T 46 Ductal Tumor mamario primario IIIA + - - Tejido tumoral microdiseccionado Validación
BB28T 47 Ductal Tumor mamario primario IIIA - + - Tejido tumoral microdiseccionado Validación
BB2BT 54 Ductal Tumor mamario primario IIA + - - Tejido tumoral microdiseccionado Validación
BB30T 60 Ductal Tumor mamario primario IIB - - + Tejido tumoral microdiseccionado Validación
BB31T 56 Ductal Tumor mamario primario IIIA - - - Tejido tumoral microdiseccionado Validación
BB32T 52 Ductal Tumor mamario primario IIA + - + Tejido tumoral microdiseccionado Validación
BB33T 36 Ductal Tumor mamario primario IV + + ++ Tejido tumoral microdiseccionado Validación
BB34T 38 Ductolobulillar Tumor mamario primario IIA + + +++ Tejido tumoral microdiseccionado Validación
8B3ST 67 Ductal Tumor mamario primario IV + + N/D Tejido tumoral microdiseccionado Validación
BB36T 60 Ductal Tumor mamario primario IIB - - ++ Tejido tumoral microdiseccionado Validación
BB37T 47 Ductal Tumor mamario primario IIA + - - Tejido tumoral microdiseccionado Validación
B838T 39 Ductal Tumor mamario primario IIB + + ++ Tejido tumoral microdiseccionado Validación
BB39T 39 Lobulillar Tumor mamario primario IIB + + +++ Tejido tumoral microdiseccionado Validación
BB40T 43 Ductal Tumor mamario primario IIA + - - Tejido tumoral microdiseccionado Validación
BB42T 56 Ductal Tumor mamario primario IIB - - - Tejido tumoral microdiseccionado Validación
BB43T 54 Ductolobulillar Tumor mamario primario IIIA + + +++ Tejido tumoral microdiseccionado Validación
BB44T 46 Ductolobulillar Tumor mamario primario I + + + Tejido tumoral microdiseccionado Validación
*El estado del receptor de estrógenos (RE) y del receptor de progesterona (RP) se determina por inmunoestioquímica. -, negativo; +, positivo; *Estado Her-2/neu: -, negativo; +, poco positivo; ++ moderadamente positivo o +++ muy positivo; N/D, datos no disponibles
ll ll
ll ll
(homocigoto)
(homocigoto)
Las posiciones genómicas son coordenadas en el documento de publicación del genoma humano 35.1UCSB de Santa Cruz mayo 2004, hg17. Las coordenadas se secuencian genómicas de las mutaciones están en la cadena codificante. Todos los cambios son heterocigotos salvo los indicados como homocigotos. g secuencia genómica; p secuencia de ADNc, s secuencia de proteína, del deleción, dup duplicación, ins. Inserción. chr cromosoma; las mutaciones en las secuencias no codificantes se anotan mediante un número intrónico precedido por IVS, comenzando con números positivos desde el sitio donante de corte y empalme G de GT y los números negativos comenzando desde el sitio aceptor de corte y empalme G del AG. FS mutación con desplazamiento de la fase de lectura; sp. Mutación en el sitio de corte y empalme; UTR, mutación en la región 5' o 3' no traducida; indel., insección en marco, deleción o cambio de duplicación que afectan a más de un solo codon. El cambio de aminoácido resultante de mutaciones en la metionina iniciadora de la traducción se indican como "desconocido".
La fracción de los 35 tumores de mama evaluados en las exploraciones de descubrimiento y valoración contiene mutaciones de los genes dividido entre el número de bases secuenciadas con éxito en ese gen (es decir, bases con puntuación de calidad Phred ≥ 20) .
La fracción de los 35 tumores colorrectales evaluada en las exploraciones de descubrimiento y valoración contiene mutaciones de los genes dividido entre el número debases secuenciadas con éxito en ese gen (es decir, bases con puntuación de calidad Phred ≥ 20) .
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]