CEBADORES DE AMPLIFICACIÓN DE ÁCIDOS NUCLEICOS PARA ESTUDIOS DE CLONALIDAD BASADA EN PCR.
Un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de IGH VH-JH que comprende un cebador directo y un cebador inverso,
en el que dicho cebador directo se selecciona de los cebadores de la familia VH mostrados en la Fig. 3B, y en el que dicho cebador inverso es el cebador consenso JH mostrado en la Fig. 3B
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/NL2003/000690.
Solicitante: ERASMUS UNIVERSITEIT ROTTERDAM.
Nacionalidad solicitante: Países Bajos.
Dirección: DR. MOLEWATERPLEIN 50 3015 GE ROTTERDAM PAISES BAJOS.
Inventor/es: VAN DONGEN,JACOBUS,JOHANNES,MARIA, HUMMEL,MICHAEL, GARZIA SANZ,RAMON, PARREIRA,ANTONIO, SMITH,JOHN,LEWIS, EVANS,PAUL,ANTHONY,STUART, MACINTYRE,ELIZABETH,ANNE, BASTARD,CHRISTIAN, DAVI,FREDERIC,BERNARD,LOUIS, SAN MIQUEL,JESUS,FERNANDO, MORGAN,GARETH,JOHN, KNEBA,MICHAEL, LANGERAK,ANTHONIE,WILLEM, LAVENDER,FRANCES,LOUISE, SCHUURING,EDUARDUS,MARIA,DOMINICUS, BRUGGEMANN,MONIKA.
Fecha de Publicación: .
Fecha Solicitud PCT: 13 de Octubre de 2003.
Fecha Concesión Europea: 6 de Octubre de 2010.
Clasificación Internacional de Patentes:
- C12Q1/68M6B
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
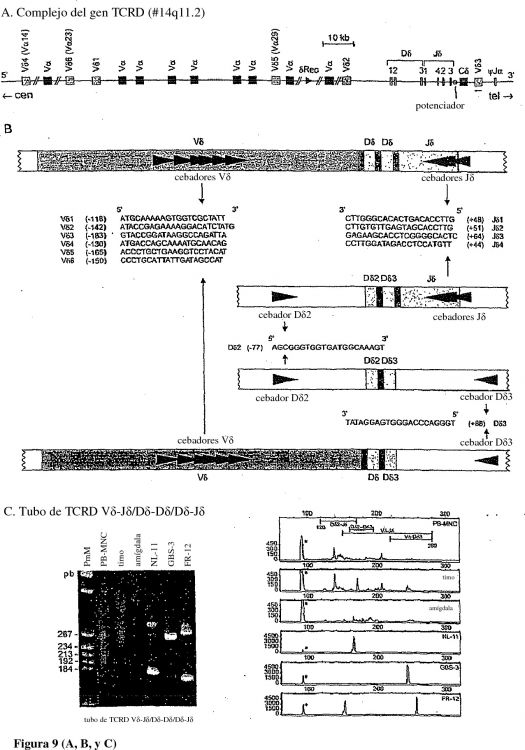
Fragmento de la descripción:
La presente invención se refiere a estudios de clonalidad basados en PCR para, entre otros, el diagnóstico temprano de los trastornos linfoproliferativos. En la mayoría de los pacientes con presuntos trastornos linfoproliferativos, la histomorfología o citomorfología complementadas con inmunohistología o inmunofenotipado mediante citometría de flujo pueden distinguir entre las linfoproliferaciones malignas y 5 reactivas. Sin embargo, en un 5 a un 10% de los casos, es más complicado realizar el diagnóstico. El diagnóstico de las neoplasias malignas linfoides se puede respaldar mediante el estudio de la clonalidad basado en el hecho de que en principio todas las células de una neoplasia maligna tienen un origen clonal común.
La mayoría de las neoplasias malignas linfoides pertenece al linaje de células B (90 al 95%), y 10 solamente una minoría pertenece al linaje de células T (5-7%) o al linaje de células NK (<2%). Las leucemias linfoblásticas agudas (LLA) tienen un origen de células T en el 15 al 20% de los casos, pero en el grupo de las leucemias linfoides maduras y en los linfomas no Hodgkin (LNH) las neoplasias malignas de células T son relativamente infrecuentes, excepto para subgrupos específicos tales como los linfomas cutáneos (Tabla 1). Por lo tanto, la gran mayoría de las neoplasias malignas linfoides (>98%) contiene 15 genes de inmunoglobulinas (Ig) y/o receptores de células T (TCR) reordenados de manera idéntica (clonalmente), y en el 25 al 30% de los casos también se hallan anormalidades cromosómicas bien definidas, todo lo cual puede servir como marcador de la clonalidad.1, 2
Los loci de los genes de Ig y TCR contienen muchos segmentos génicos variables (V), diversos (D), y de unión (J) diferentes, que están sometidos a procesos de reordenamiento durante la 20 diferenciación linfoide temprana.3, 4 Los reordenamientos V-D-J están mediados por un complejo enzimático recombinasa en el que las proteínas RAG1 y RAG2 desempeñan un papel fundamental reconociendo y cortando el ADN en las secuencias señal de recombinación (RSS), que están localizadas en dirección 3' respecto de los segmentos génicos V, a ambos lados de los segmentos génicos D, y en dirección 5' de los segmentos génicos J (Figura 1). Las RSS inadecuadas reducen o incluso impiden 25 completamente el reordenamiento.
El proceso de reordenamiento comienza en general con un reordenamiento D a J seguido por un reordenamiento V a D-J en el caso de los genes de la cadena pesada de Ig (IGH), TCR beta (TCRB), y TCR delta (TCRD) (Figura 1), o implica reordenamientos directos V a J en el caso de los genes de Ig kappa (IGK), Ig lambda (IGL), TCR alfa (TCRA), y TCR gamma (TCRG). Las secuencias entre los 30 segmentos génicos reordenados generalmente se delecionan en forma de un producto de escisión circular, también denominado círculo de escisión de TCR (TREC) o círculo de escisión de receptores de células B (BREC) (Figura 1).
Los reordenamientos de los genes de Ig y TCR durante la diferenciación linfoide temprana siguen generalmente un orden jerárquico. Durante la diferenciación de las células B: primero se reordenan los 35 genes IGH, después IGK, lo que potencialmente da como resultado la expresión de IgH/κ o seguido por la deleción de IGK y el reordenamiento de IGL, potencialmente seguido por la expresión de IgH/λ.5 Esto im-plica que prácticamente todas las células B Igλ+ tienen deleciones del gen IGK monoalélicas o bialélicas. Durante la diferenciación de las células T: primero se reordenan los genes TCRD, después TCRG, lo que potencialmente da como resultado la expresión de TCRγδ o seguido por el reordenamiento posterior de 40 TCRB y la deleción de TCRD con el reordenamiento posterior de TCRA, potencialmente seguido por la expresión de TCRαβ. Los patrones de reordenamiento de los genes de Ig y TCR en las neoplasias malignas linfoides se ajustan en general al orden jerárquico anteriormente descrito, aunque también se hallan patrones de reordenamiento poco habituales en particular en LLA.6
Las muchas combinaciones diferentes de segmentos génicos V, D, y J representan el 45 denominado repertorio combinatorio (Tabla 2), que se estima que es ~2×106 para las moléculas Ig, ~3×106 para las moléculas TCRαβ y ~5×103 para las moléculas TCRγδ. En los sitios de unión de los segmentos génicos V, D, y J, se da la deleción y la inserción aleatoria de nucleótidos durante el proceso de reordenamiento, lo que da como resultado regiones de unión muy diversas, lo que contribuye de ma-nera significativa al repertorio total de moléculas Ig y TCR, que se estima que es >1012.5 50
Los linfocitos B maduros amplían adicionalmente su repertorio de Ig tras el reconocimiento del antígeno en los centros foliculares por medio de la hipermutación somática, un proceso que conduce a la maduración por afinidad de las moléculas Ig. El proceso de hipermutación somática se centra en el exón V-(D-)J de los genes IGH y de la cadena ligera de Ig, e implica mutaciones de nucleótidos individuales y a veces también inserciones o deleciones de nucleótidos. Los genes de Ig mutados somáticamente se 55 hallan también en las neoplasias malignas células B maduras de origen folicular o post-folicular.7
Los genes de Ig y TCR reordenados funcionalmente dan como resultado la expresión en las membranas superficiales de moléculas de Ig, TCRαβ, o TCRγδ. Basándose en el concepto de que solamente un único tipo de molécula Ig o TCR se expresa en un linfocito o en un clon de linfocitos, los genes reordenados clonalmente de las neoplasias malignas linfoides maduras podrían ser detectables a 60
nivel proteico. La detección de la expresión de cadenas ligeras de Ig únicas (Igκ o Igλ) se ha usado du-rante mucho tiempo para distinguir entre los linfocitos B reactivos (policlonales) (proporción Igκ/Igλ normal: 0,7 - 2,8) frente a los linfocitos B anormales (clonales) con proporciones Igκ/Igλ de >4,0 o <0,5.8-10 En la gran mayoría (>90%) de las neoplasias malignas de células B maduras, la expresión de cadenas ligeras de Ig únicas puede respaldar el origen clonal de la neoplasia maligna. 5
Además, el desarrollo de muchos anticuerpos diferentes contra dominios variables de las diversas cadenas de TCR permite la detección de dominios Vβ, Vγ y Vδ monotípicos, cuando se comparan con los valores de referencia adecuados.11-16 En la interpretación de los resultados de Vβ monotípicos mediante el uso de 20 a 25 anticuerpos contra diferentes familias de Vβ (Tabla 2), se debería tener en cuenta que las expansiones de células T TCRαβ+ clonales clínicamente benignas (con frecuencia 10 CD8+) se hallan frecuentemente en sangre periférica (PB) de individuos mayores.13 Estas expansiones de células T clonales en PB son, sin embargo, relativamente pequeñas en tamaño: <40% de los linfocitos T de PB y <0,5x106/ml de PB.13 Todavía no está claro hasta qué punto tales clones de células T clínicamente benignas se pueden hallar también en los tejidos linfoides.
Los resultados de la expresión de los dominios Vγ y Vδ monotípicos se deberían interpretar con 15 cautela, debido a que en los individuos sanos una gran fracción de los linfocitos T TCRγδ+ policlonales normales se ha seleccionado para el uso de Vγ9-Jγ1.2 y Vδ2-Jδ1.18, 19 Por lo tanto, las frecuencias elevadas de linfocitos T Vγ9+/Vδ2+ en PB se debería considerar como un hallazgo normal, a menos que el recuento absoluto sea de 1 a 2×106/ml de PB. Se debería indicar que la mayoría de las neoplasias malignas de células T TCRγδ+ expresan Vδ1 u otro segmento génico que no es Vδ2 en combinación con 20 un único dominio Vγ (en general no Vγ9).15, 20
La detección de la expresión restringida de Igκ o Igλ o la expresión de Vβ, Vγ o Vδ monotípicos es relativamente fácil en los estudios de citometría de flujo de muestras de PB y de médula ósea (BM) de pacientes con leucemias de células T o células B maduras. Sin embargo, esto parece ser más difícil en muestras de tejidos con presuntos trastornos linfoproliferativos que están mezclados con linfocitos 25 normales (reactivos).
En contraste con las técnicas basadas en anticuerpos, las técnicas moleculares son generalmente aplicables para la detección de los genes de Ig/TCR reordenados clonalmente, así como anormalidades cromosómicas bien definidas. Previamente esto implicaba el análisis...
Reivindicaciones:
1. Un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de IGH VH-JH que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores de la familia VH mostrados en la Fig. 3B, y en el que dicho cebador inverso es el cebador consenso JH mostrado en la Fig. 3B. 5
2. Un ensayo de amplificación de ácido nucleico, preferiblemente un ensayo de PCR, y más preferiblemente un ensayo de PCR multiplex, que usa al menos un grupo de cebadores según la reivindicación 1.
3. El ensayo de amplificación de ácido nucleico de la reivindicación 2, caracterizado porque es un ensayo de PCR. 10
4. El ensayo de amplificación de ácido nucleico de la reivindicación 2 o 3, caracterizado porque es un ensayo de PCR multiplex, que comprende además uno o más de los grupos de cebadores elegidos del grupo que consiste en:
a) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de IGH DH-JH que comprende un cebador directo y un cebador inverso, en el que 15 dicho cebador directo se selecciona de los cebadores de la familia DH mostrados en la Fig. 4A, y en el que dicho cebador inverso es el cebador consenso JH mostrado en la Fig. 4A.
b) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de IGK VK-JK que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores de la familia VK mostrados en la Fig. 5B, y 20 en el que dicho cebador inverso es un cebador JK mostrado en la Fig. 5B.
c) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de IGK VK/intrón-Kde que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores VK o del cebador INTR mostrados en la Fig. 5B, y en el que dicho cebador inverso es el cebador Kde mostrado en la Fig. 25 5B.
d) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de IGL Vλ-Jλ que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores Vλ mostrados en la Fig. 6B, y en el que dicho cebador inverso es el cebador Jλ mostrado en la Fig. 6B. 30
e) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de TCRB Vβ-Jβ que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores de la familia Vβ mostrados en la Fig. 7B, y en el que dicho cebador inverso se selecciona de los cebadores JβA y JβB mostrados en la Fig. 7B. 35
f) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de TCRB Dβ-Jβ que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores Dβ mostrados en la Fig. 7B, y en el que dicho cebador inverso se selecciona de los cebadores JβA y JβB mostrados en la Fig. 7B.
g) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un 40 reordenamiento de TCRG Vγ-Jγ que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores de la familia Vγ mostrados en la Fig. 8B, y en el que dicho cebador inverso se selecciona de los cebadores Jγ mostrados en la Fig. 8B.
h) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un 45 reordenamiento de TCRD Vδ-Jδ que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores Vδ mostrados en la Fig. 9B, y en el que dicho cebador inverso se selecciona de los cebadores Jδ mostrados en la Fig. 9B.
i) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de TCRD Dδ-Dδ que comprende un cebador directo y un cebador inverso, en el 50 que dicho cebador directo es el cebador Dδ2 mostrado en la Fig. 9B, y en el que dicho cebador inverso es el cebador D83 mostrado en la Fig. 9B.
j) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de TCRD Dδ-Jδ que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo es el cebador Dδ2 mostrado en la Fig. 9B, y en el que dicho cebador 55 inverso se selecciona de los cebadores Jδ mostrados en la Fig. 9B.
k) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar un reordenamiento de TCRD Vδ-Dδ que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo se selecciona de los cebadores Vδ mostrados en la Fig. 9B, y en el que dicho cebador inverso es el cebador D83 mostrado en la Fig. 9B.
l) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar una 5 translocación cromosómica (11;14)(BCL1-IGH) que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo es el cebador BCL1/MTC tal como se muestra en la Fig. 10A, y en el que dicho cebador inverso es el cebador consenso JH mostrado en la Fig. 10A.
m) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar una translocación cromosómica t(14;18)(BCL2-IGIH), que comprende un cebador directo y un 10 cebador inverso, en el que dicho cebador directo se selecciona de los cebadores MBR, los cebadores 3'MBR y los cebadores mcr mostrados en la Fig. 11A, y en el que dicho cebador inverso es el cebador consenso JH mostrado en la Fig. 11A.
n) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar el gen TBXAS1 humano que comprende un cebador directo y un cebador inverso, en el que dicho 15 cebador directo es el cebador TBXAS1/X9U de la Fig. 12A, y en el que dicho cebador inverso es el cebador TBXAS 1/X9L de la Fig. 12A.
o) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar el gen de la proteína activadora de la recombinación (RAG1) humana que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo es el cebador RAG1/X2U de la Fig. 12A, y en el 20 que dicho cebador inverso es el cebador RAG1/X2L de la Fig. 12A.
p) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar la proteína con dedo de zinc de leucemia promielocítica (PLZF) humana que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo es el cebador PLZF/X1U de la Fig. 12A, y en el que dicho cebador inverso es el cebador PLZF/X1L de la Fig. 12A. 25
q) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar el gen AF4 humano (Exón 3) que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo es el cebador AF4/X3U de la Fig. 12A, y en el que dicho cebador inverso es el cebador AF4/X3L de la Fig. 12A.
r) un grupo de cebadores de amplificación de ácido nucleico capaces de amplificar el gen AF4 30 humano (Exón11) que comprende un cebador directo y un cebador inverso, en el que dicho cebador directo es el cebador AF4/X11U de la Fig. 12A, y en el que dicho cebador inverso es el cebador AF4/X11L de la Fig. 12A.
5. Un método para detectar un reordenamiento de IGH VH-JH, que comprende el uso de uno o más grupos de cebadores según la reivindicación 1 en un ensayo de amplificación de ácido 35 nucleico según cualquiera de las reivindicaciones 2-4.
6. Un método para detectar dos o más reordenamientos, dos o más translocaciones o al menos un reordenamiento y al menos una translocación, en el que dicho reordenamiento al menos comprende un reordenamiento de IGH VH-JH, seleccionado del grupo que consiste en un reordenamiento de IGH DH-JH, un reordenamiento de IGK Vκ-Jκ, un reordenamiento de IGK Vκ/intrón-Kde, un 40 reordenamiento de IGL Vλ-Jλ, un reordenamiento de TCRB Vβ-Jβ, un reordenamiento de TCRB Dβ-Jβ, un reordenamiento de TCRG Vγ-Jγ, un reordenamiento de TCRD Vδ-Jδ, un reordenamiento de TCRD Dδ-Dδ, un reordenamiento de TCRD Dδ-Jδ, un reordenamiento de TCRD Vδ-Dδ, una translocación t(11;14)(BCL1-IGH) y una translocación t(14;18)(BCL2-IGH), mediante el uso de un ensayo de amplificación de ácido nucleico según la reivindicación 4. 45
7. Un método para estudiar reordenamientos clonales, que comprende al menos un reordenamiento de IGH VH-JH, y/o anormalidades cromosómicas mediante el uso de al menos un grupo de cebadores según la reivindicación 1, y opcionalmente al menos un grupo de cebadores tal como se definieron en la reivindicación 4a-4r.
8. Un método según la reivindicación 7 para la detección de la enfermedad residual 50 mínima (ERM) o para la identificación de objetivos de PCR a ser usados para la detección de la ERM por medio de PCR cuantitativa en tiempo real.
9. Un método según la reivindicación 7 o 8, en el que se detecta un ácido nucleico amplificado mediante el uso de un análisis de fragmentos de PCR de alta resolución automatizado.
10. Un equipo para la detección de al menos un reordenamiento de IGH VH-JH y 55 opcionalmente un reordenamiento seleccionado del grupo que consiste en un reordenamiento de IGH DH-JH, un reordenamiento de IGK Vκ-Jκ, un reordenamiento de IGK Vκ/intrón-Kde, un reordenamiento de IGL Vλ-Jλ, un reordenamiento de TCRB Vβ-Jβ, un reordenamiento de TCRB Dβ-Jβ, un reordenamiento de
TCRG Vγ-Jγ, un reordenamiento de TCRD Vδ-Jδ, un reordenamiento de TCRD Dδ-Dδ, un reordenamiento de TCRD Dδ-Jδ, un reordenamiento de TCRD Vδ-Dδ, y una translocación seleccionada de t(11;14)(BCL1-IGH) y t(14;18)(BCL2-IGH), que comprende el uso de al menos un grupo de cebadores según la reivindicación 1, y opcionalmente al menos un grupo de cebadores tal como se definieron en la reivindicación 4a-4m. 5
11. Un equipo según la reivindicación 10, que comprende además al menos un grupo de cebadores tal como se definieron en la reivindicación 4º-4r.
Patentes similares o relacionadas:
MARCADORES PARA LA SELECCIÓN DE TERAPIAS PERSONALIZADAS PARA EL TRATAMIENTO DEL CÁNCER, del 7 de Febrero de 2012, de FUNDACION INSTITUT DE RECERCA DE L'HOSPITAL UNIVERSITARI VALL D'HEBRON: La invención se relaciona con la identificación de los niveles de expresión de aprataxina (APTX) como marcador de respuesta a terapias basadas en inhibidores de topoisomerasa […]
MÉTODOS PARA SELECCIONAR REGÍMENES DE TRATAMIENTO Y PREDECIR RESULTADOS EN PACIENTES CON CÁNCER DE MAMA, del 6 de Febrero de 2012, de AMERICAN DIAGNOSTICA INC: Un método para seleccionar de dos o más regímenes de tratamiento, un régimen de tratamiento que tiene el mayor beneficio esperado para un […]
 METODO Y KIT PARA EL PRONOSTICO DEL LINFOMA DE CELULAS DEL MANTO, del 23 de Enero de 2012, de HOSPITAL CLINIC DE BARCELONA UNIVERSIDAD DE BARCELONA FUNDACIO CLINIC PER A LA RECERCA BIOMEDICA INSTITUT D'INVESTIGATIONS BIOMEDIQUES AUGUST PI I SUNYER (IDIBAPS): Método y kit para el pronóstico del linfoma de células del manto.
El método y el kit son útiles como herramientas para clasificar un paciente diagnosticado con linfoma […]
METODO Y KIT PARA EL PRONOSTICO DEL LINFOMA DE CELULAS DEL MANTO, del 23 de Enero de 2012, de HOSPITAL CLINIC DE BARCELONA UNIVERSIDAD DE BARCELONA FUNDACIO CLINIC PER A LA RECERCA BIOMEDICA INSTITUT D'INVESTIGATIONS BIOMEDIQUES AUGUST PI I SUNYER (IDIBAPS): Método y kit para el pronóstico del linfoma de células del manto.
El método y el kit son útiles como herramientas para clasificar un paciente diagnosticado con linfoma […]
PROCEDIMIENTOS Y COMPOSICIONES PARA EL DIAGNÓSTICO Y TRATAMIENTO DEL CÁNCER DE PULMÓN UTILIZANDO EL GEN DE PDGFRA, KIT O KDR COMO MARCADOR GENÉTICO, del 17 de Enero de 2012, de GENENTECH, INC.: Procedimiento para diagnosticar la presencia de un cáncer de pulmón en un mamífero, comprendiendo el procedimiento la detección de si el gen de PDGFRA está […]
MÉTODOS Y SONDAS PARA LA DETECCIÓN DEL CÁNCER, del 12 de Enero de 2012, de VYSIS, INC. MAYO FOUNDATION FOR MEDICAL EDUCATION AND RESEARCH: Un conjunto de sondas cromosómicas que comprende la siguiente combinación de cuatro sondas: una sonda específica del locus 5p15, una sonda específica del locus 8q24, […]
COMPOSICIÓN Y PROCEDIMIENTO PARA LA DETECCIÓN, DIAGNÓSTICO Y TERAPÍA DE NEOPLASIAS HEMATOLÓGICAS, del 22 de Diciembre de 2011, de CORIXA CORPORATION: Uso de una cantidad eficaz de un anticuerpo monoclonal aislado que se une específicamente a un polipéptido que comprende la secuencia expuesta en SEC ID Nº: 4 […]
MÉTODO NORMALIZADO Y OPTIMIZADO DE REACCIÓN EN CADENA DE LA POLIMERASA DE TRANSCRIPTASA INVERSA CUANTITATIVA EN TIEMPO REAL PARA LA DETECCIÓN DE MRD EN LEUCEMIA, del 14 de Diciembre de 2011, de UNIVERSITE DE LA MEDITERRANEE: Conjunto de ácidos nucleicos ABL para RQ-PCR que comprende una sonda nucleotídica que tiene la secuencia SEC ID N.º 2 y cebadores directo e inverso que tienen las secuencias […]
BIOMARCADORES Y PROCEDIMIENTOS PARA DETERMINAR LA SENSIBILIDAD A MODULADORES DEL RECEPTOR DEL FACTOR DE CRECIMIENTO EPIDÉRMICO, del 12 de Diciembre de 2011, de BRISTOL-MYERS SQUIBB COMPANY: Un procedimiento in vitro para predecir la probabilidad de que un mamífero responda terapéuticamente a un procedimiento de tratamiento del […]