SECUENCIAS PARA LA DETECCION Y LA IDENTIFICACION DE STAPHYLOCCOCUS AUREUS RESISTENTE A LA METICILINA.
Un método para detectar la presencia de una cepa de Staphylococcus aureus resistente a meticilina (MRSA) en una muestra,
dicha cepa de MRSA siendo resistente debido a la presencia de un inserto SCCmec conteniendo un gen mecA, dicho SCCmec estando insertado en ácidos nucleicos bacterianos generando por lo tanto un punto de unión en el extremo derecho polimórfico (MREJ), dicho método comprende el paso de hibridación de los ácidos nucleicos de la muestra con varias sondas y/o cebadores, caracterizados, por:
(i) dichos cebadores y/o sondas son específicos para cepas MRSA y son capaces de hibridarse con ácidos nucleicos MREJ polimórficos, dichos MREJ polimórficos comprendiendo los MREJ de tipos i,a,x; y
(ii) dichos cebadores y/o sondas en conjunto pueden hibridar con al menos cuatro tipos de MREJ seleccionados de MREJ tipos de i,a,x
Tipo: Resumen de patente/invención. Número de Solicitud: W0200824CA.
Solicitante: GENEOHM SCIENCES CANADA INC.
Nacionalidad solicitante: Canadá.
Dirección: 2050, BOULEVARD RENE LEVESQUE OUEST 4EME ETAGE,SAINTE-FOY, QC G1V 2K8.
Inventor/es: HULETSKY,ANN, ROSSBACH,VALERY.
Fecha de Publicación: .
Fecha Concesión Europea: 4 de Noviembre de 2009.
Clasificación Internacional de Patentes:
- C12Q1/68M10B
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Secuencias para la detección y la identificación de Staphyloccocus aureus resistente a la meticilina.
Antecedentes de la invención
Significado clínico de Staphylococcus aureus
La especie Staphylococcus aureus positiva a la coagulasa está muy documentada como patógeno oportunista humano. Las infecciones intrahospitalarias ocasionadas por S. aureus son una causa principal de morbomortalidad. Algunas de las infecciones más frecuentes ocasionadas por S. aureus afectan a la piel, e incluyen forúnculos o diviesos, celulitis, impétigo e infecciones de heridas posoperatorias. Algunas de las infecciones más graves producidas por S. aureus son la bacteremia, neumonía, osteomielitis, endocarditis aguda, miocarditis, pericarditis, cerebritis, meningitis, necrólisis epidérmica tóxica y varios abscesos. El envenenamiento por alimentos mediado por enterotoxinas estafilocócicas es otro síndrome importante asociado a S. aureus. El síndrome del choque tóxico, una enfermedad adquirida en la comunidad, se ha atribuido también a la infección o colonización con S. aureus toxígeno (Murray et al. Eds, 1999, Manual of Clinical Microbiology, 7ª Ed., ASM Press, Washington, D.C.).
S. aureus resistente a la meticilina (MRSA) apareció en la década de 1980 como un problema clínico y epidemiológico principal en los hospitales. Los MRSA son resistentes a todas las ß-lactamas incluyendo las penicilinas, cefalosporinas, carbapenemos y monobactamos, que son los antibióticos usados más frecuentemente para curar las infecciones por S. aureus. Las infecciones por MRSA pueden tratarse solamente con los antibióticos más tóxicos y más costosos, que son utilizados normalmente como la última línea de defensa. Como el MRSA puede propagarse fácilmente de paciente a paciente a través del personal, los hospitales en todo el mundo están haciendo frente al problema para controlar el MRSA. Por consiguiente, existe una necesidad de desarrollar pruebas rápidas y sencillas de identificación o de diagnóstico para la detección y/o identificación del MRSA destinadas a reducir su diseminación y mejorar el diagnóstico y el tratamiento de los pacientes infectados.
La resistencia a la penicilina en el S. aureus es única porque se debe a la adquisición del ADN procedente de otros estafilococos negativos a la coagulasa (CNS), que codifican una proteína que se une a la penicilina (PBP) resistente a ß-lactama suplementaria, que adquiere las funciones biosintéticas de las PBP normales cuando la célula se expone a antibióticos con ß-lactama. S. aureus normalmente contiene cuatro PBP, de los cuales 1, 2 y 3 PBP son esenciales. La PBP de baja afinidad en MRSA, denominada PBP 2a (o PBP 2'), está codificada por el gen mecA cromosómico y funciona como una transpeptidasa resistente a ß-lactama. El gen mecA está ausente en S. aureus sensible a la meticilina pero está ampliamente distribuido entre otras especie de estafilococos y está muy conservado (Ubukata et al., 1990, Antimicrob. Agents Chemother. 34:170-172).
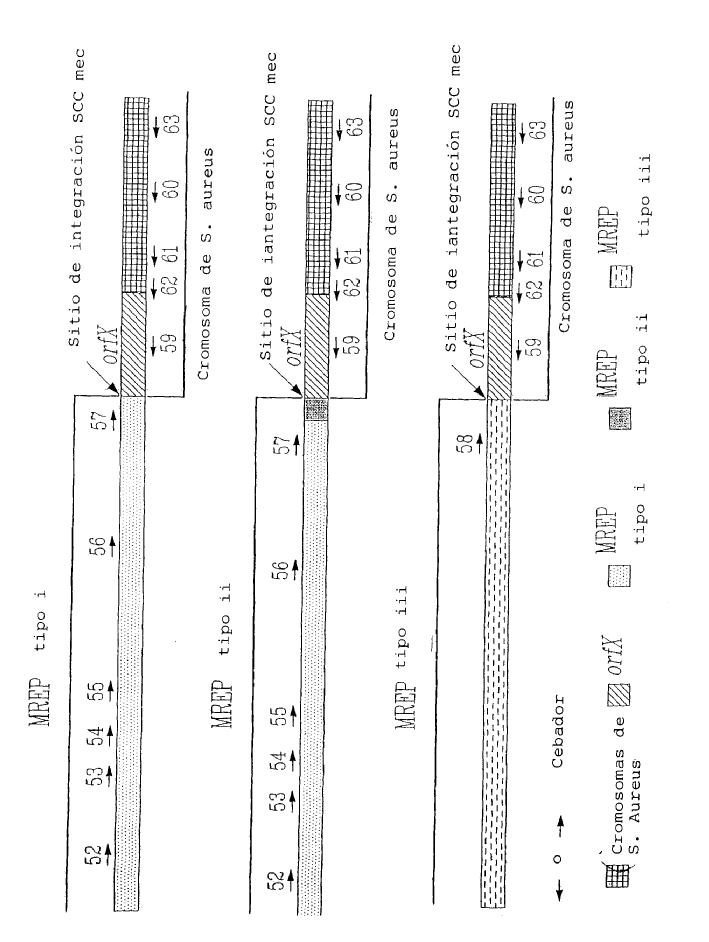
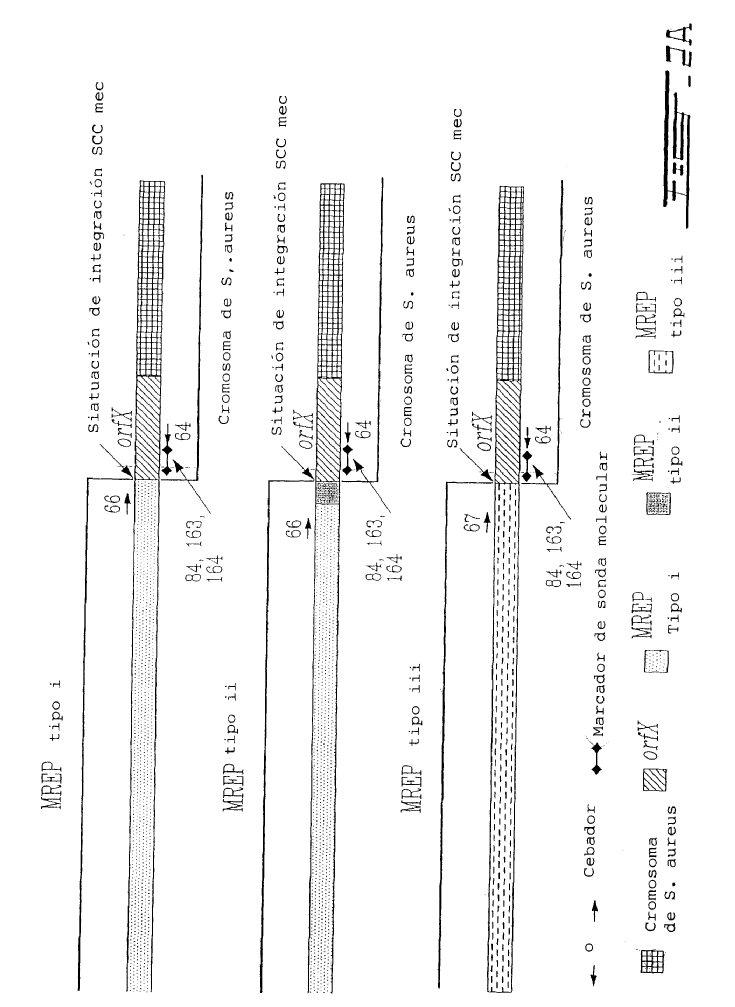
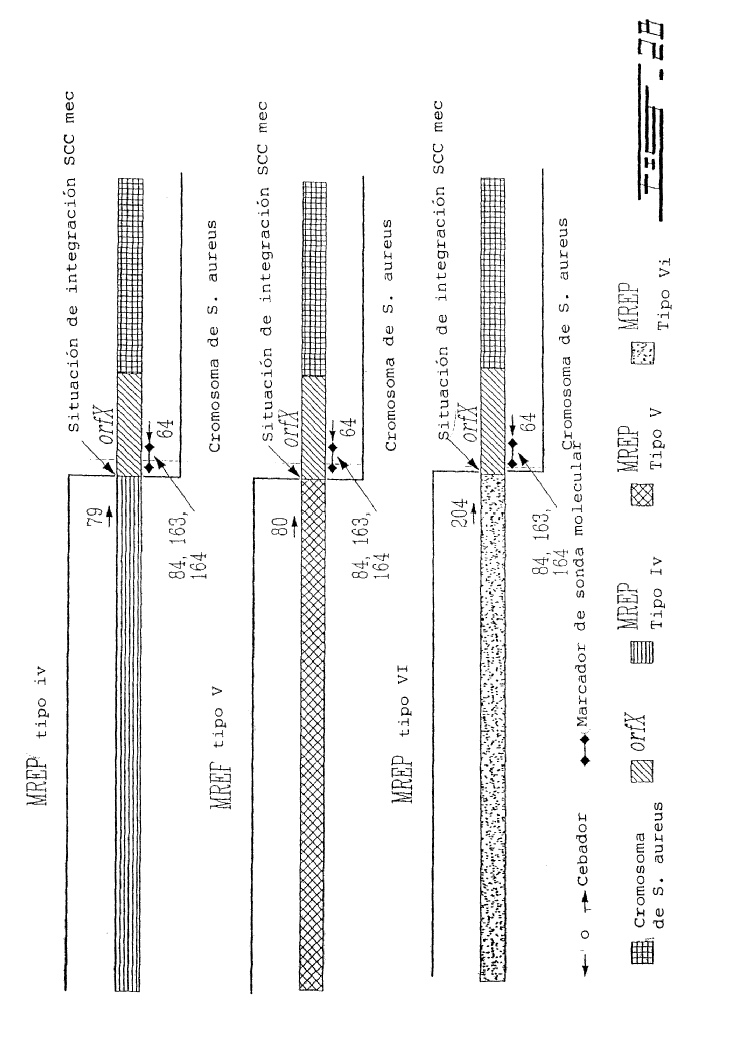
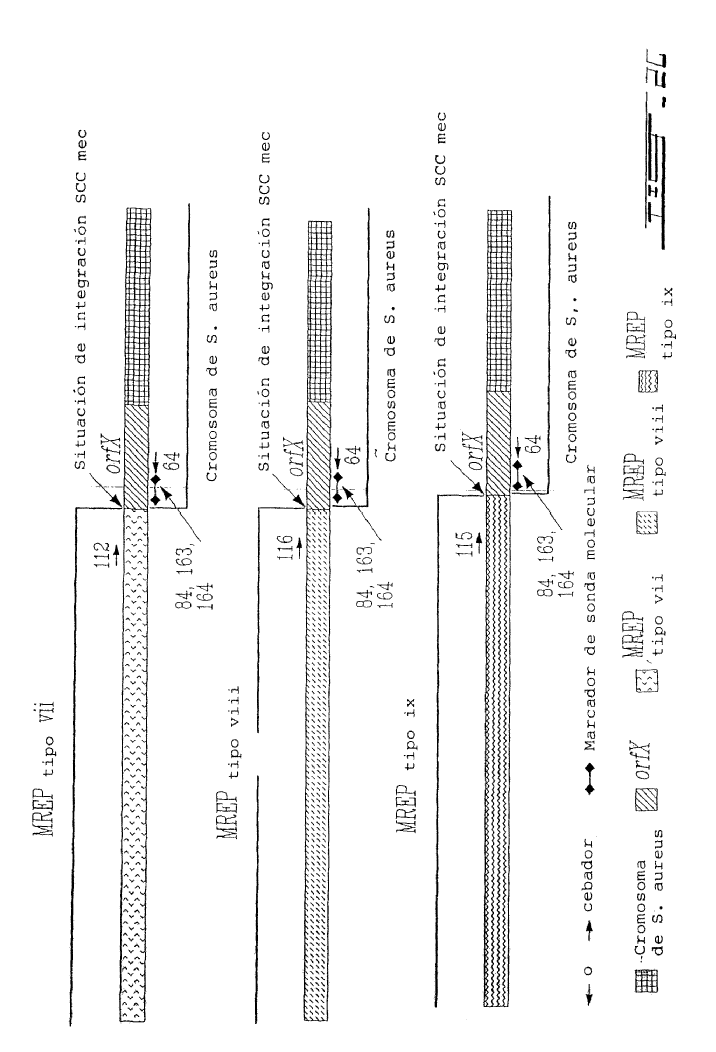
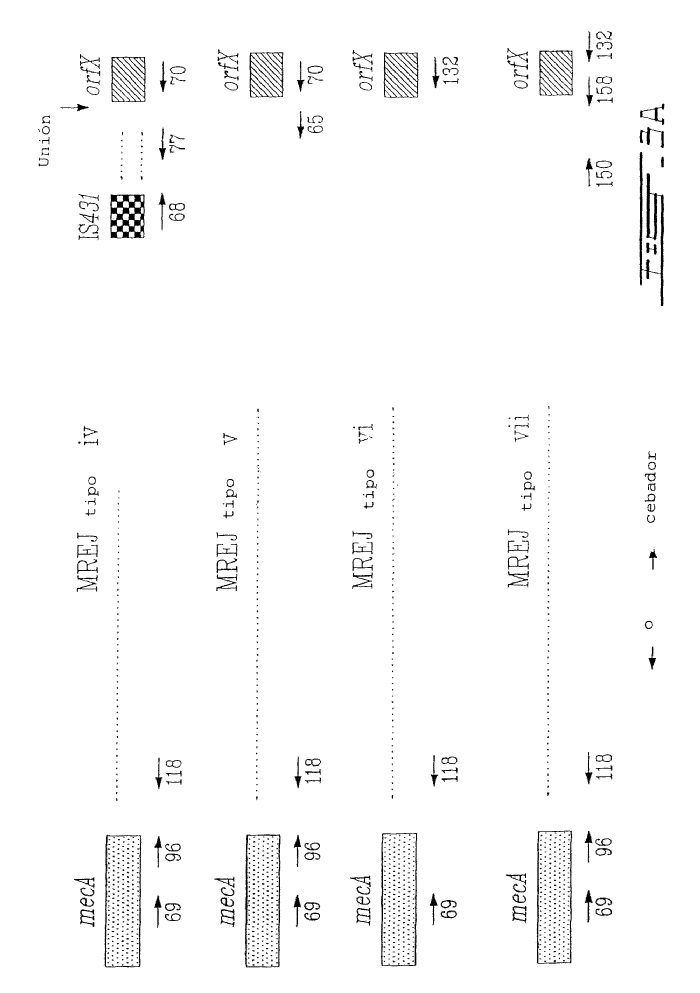
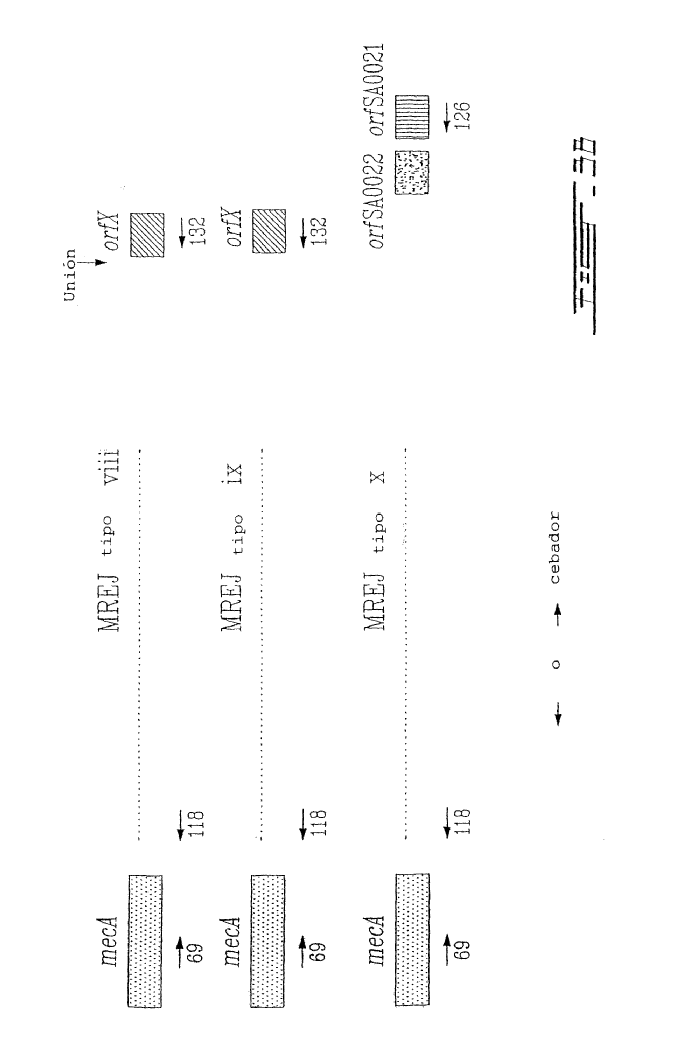
Mediante la determinación de la secuencia nucleotídica de la zona del ADN que rodea al gen mecA de la cepa N315 de S. aureus (aislada en Japón en 1982), Hiramatsu et al. han descubierto que el gen mecA es transportado por un nuevo elemento genético, denominado cromosoma mec de la casete estafilocócica (SCCmec), insertado en el cromosoma. SCCmec es un elemento genético móvil caracterizado por la presencia del terminal invertido y de repeticiones directas, una serie de genes de recombinasa específica para el sitio (ccrA y ccrB), y el complejo génico mecA (Ito et al., 1999, Antimicrob. Agents Chemother. 43:1449-1458; Katayama et al., 2000, Antimicrob. Agents Chemother. 43:1549-1555). El elemento se escinde exactamente en el cromosoma de la cepa N315 de S. aureus y se integra en una secuencia cromosómica específica de S. aureus en la misma orientación a través de la función de una única serie de genes de recombinasa que comprende ccrA y ccrB. Se descubrieron dos elementos genéticos nuevos que compartían propiedades estructurales similares de SCCmec clonando y secuenciando la zona del ADN que rodea al gen mecA de las cepas NCTC 10442 del MRSA (la primera cepa de MRSA se aisló en Inglaterra en 1961) y 85/2082 (una cepa procedente de Nueva Zelanda aislada en 1985). Las tres SCCmec se han denominado tipo I (NCTC 10442), tipo II (N315) y tipo III (85/2082) haciendo referencia al año de aislamiento de las cepas (Ito et al., 2001, Antimicrob. Agents Chemother. 45:1323-1336) (Figura 1). Hiramatsu et al. han descubierto que los ADN del SCCmec están integrados a un sitio específico en el cromosoma de S. aureus sensible a la meticilina (MSSA). En el contexto de la presente invención se caracterizaron las secuencias nucleotídicas de las zonas alrededor de los límites izquierdo y derecho del ADN de SCCmec (es decir attL y attR, respectivamente) así como a los de las zonas alrededor de la secuencia de integración del ADN de SCCmec (es decir attBscc que es la secuencia de acoplamiento del cromosoma bacteriano para el ADN del SCCmec). La secuencia attBscc se situó en el extremo 3' de un nuevo marco de lectura abierto (ORF), orfX. El orfX codifica potencialmente una identidad que comparte polipéptidos de 159 aminoácidos con algunos polipéptidos identificados anteriormente, pero de función desconocida (Ito et al., 1999, Antimicrob. Agents Chemother. 43:1449-1458). Recientemente, un nuevo tipo de SCCmec (tipo IV) se ha sido descrito tanto por Hiramatsu et al. (Ma et al, 2002, Antimicrob. Agents Chemother. 46:1147-1152) como por Oliveira et al. (Oliveira et al, 2001, Microb. Drug Resist. 7:349-360). Las secuencias del extremo derecho del nuevo SCCmec de tipo IV procedente de las cepas CA05 y 8/6-3P de S. aureus publicada por Hiramatsu et al. (Ma et al, 2002, Antimicrob. Agents Chemother. 46:1147-1152) fueron casi idénticos en 2000 nucleótidos a las SCCmec de tipo II de la cepa N315 de S. aureus (Ito et al., 2001, Antimicrob. Agents Chemother. 45:1323-1336). Ninguna secuencia en el extremo derecho del SCCmec de tipo IV está disponible de las cepas HDE288 y PL72 de S. aureus descritas por Oliveira et al. (Oliveira et al, 2001, Microb. Drug Resist. 7:349-360).
Los métodos anteriores utilizados para detectar e identificar MRSA (Saito et al., 1995, J. Clin. Microbiol. 33:2498-2500; Ubukata et al., 1992, J. Clin. Microbiol. 30:1728-1733; Murakami et al., 1991, J. Clin. Microbiol. 29:2240-2244; Hiramatsu et al., 1992, Microbiol. Inmunol. 36:445-453), que se basan en la detección del gen mecA y las secuencias cromosómicas específicas de S. aureus, encontraron dificultad en discriminar el MRSA de los estafilococos negativos a coagulasa (CNS) resistentes a la meticilina porque el gen mecA está ampliamente distribuido tanto en las especies de S. aureus como de CNS (Suzuki et al., 1992, Antimicrob. Agents Chemother. 36:429-434). Hiramatsu et al. (patente US nº 6.156.507) han descrito un ensayo de PCR específico para MRSA utilizando cebadores que pueden hibridarse específicamente a los extremos derechos de los 3 tipos de ADN de SCCmec en combinación con un cebador específico para el cromosoma de S. aureus, que corresponde a la secuencia nucleotídica en el lado derecho de la secuencia de integración SCCmec. Como las secuencias nucleotídicas que rodean la secuencia de integración SCCmec en otras especies estafilocócicas (tales como S. epidermidis y S. haemolyticus) son diferentes de las encontradas en S. aureus, este ensayo de PCR era específico para la detección del MRSA. Este ensayo de PCR suministró también información para el tipado de MREP (posición para el "polimorfismo con el extremo derecho mec") del ADN con SCCmec (Ito et al., 2001, Antimicrob. Agents Chemother. 45:1323-1336; Hiramatsu et al., 1996, J. Infect. Chemother. 2:117-129). Este procedimiento de tipado se aprovecha del polimorfismo en el extremo derecho de los ADN con SCCmec adyacentes a la secuencia de integración entre los tres tipos de SCCmec. El tipo III tiene una sola secuencia nucleotídica mientras que...
Reivindicaciones:
1. Un método para detectar la presencia de una cepa de Staphylococcus aureus resistente a meticilina (MRSA) en una muestra, dicha cepa de MRSA siendo resistente debido a la presencia de un inserto SCCmec conteniendo un gen mecA, dicho SCCmec estando insertado en ácidos nucleicos bacterianos generando por lo tanto un punto de unión en el extremo derecho polimórfico (MREJ), dicho método comprende el paso de hibridación de los ácidos nucleicos de la muestra con varias sondas y/o cebadores, caracterizados, por:
2. El método de la reivindicación 1, en donde los cebadores y/o sondas se eligen para hibridar bajo condiciones de hibridación comunes.
3. El método de la reivindicación 2, en donde el cebador y/o las sondas se disponen, en conjunto en el mismo enclave físico.
4. El método de cualquiera de las reivindicaciones 1 a 3, en donde los cebadores y/o sondas con al menos 10 nucleótidos de longitud y son capaces de hibridarse con los MREJ de tipos de i a iii, tal como se define en cualquiera de las ID de SEC. Nº: 1, 20, 21, 22, 23, 24, 25, 41; 199; 2, 17, 18, 19, 26, 40, 173, 174, 175, 176, 177, 178, 179, 180, 181, 182, 183, 185, 186, 197; 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 104, 184, 198;
y con uno o más de los MREJ de tipos de iv a ix, con ID de SEC Nº: 42, 43, 44, 45, 46, 51; 47, 48, 49, 50; 171; 165, 166; 167; 168.
5. El método de cualquiera de las reivindicaciones de 1 a 4, en donde los cebadores y/o sondas en conjunto se hibridan con dichas ID de SEC Nº de MREJ de tipos de i a ix.
6. El método de cualquiera de las reivindicaciones 1 a 5, en donde dichos cebadores y/o sondas tienen las siguientes secuencias ID de SEC Nº:




7. El método de la reivindicación 6, en donde los pares de cebadores tienen las secuencias de nucleótidos que se definen en la ID de SEC Nº


8. El método de la reivindicación 7, comprendiendo además las sondas con las siguientes secuencias: ID de SEC Nº: 32, 83, 84, 160, 161, 162, 163, 164 para la detección de MREJ de tipos i a ix.
9. El método de cualquiera de las reivindicaciones 6 a 8, en donde dichos cebadores y sondas tienen las siguientes secuencias de nucleótidos:
10. El método de cualquiera de las reivindicaciones de 1 a 8, en donde dichos cebadores y sondas se usan conjuntamente.
11. El método de la reivindicación 9 ó 10, en donde dichas sondas y/o cebadores se usan conjuntamente en el mismo enclave físico.
12. Un método para tipificar un MREJ de una cepa MRSA, que comprende los pasos de: reproducir el método de cualquiera de las reivindicaciones de 1 a 1 con los cebadores y/o sondas específicos para un tipo de MREJ determinado, y detectar un cebador y/o sonda hibridados como indicación de las presencia de un tipo de MREJ determinado.
13. Un ácido nucleico seleccionado de:
14. Un oligonucleótido de al menos 10 nucleótidos de longitud que hibrida con el ácido nucleico de la reivindicación 13 y que hibrida con uno o más MREJ de tipos seleccionados de iv a ix.
15. Un par de oligonucleótidos que tienen las secuencias de nucleótidos definidas en cualquiera de ID de SEC Nº:


16. Un oligonucleótido que tiene la secuencia de nucleótidos definida en cualquiera de la ID de SEC Nº: 32, 83, 84, 160, 161, 162, 163, 164.
17. Una composición en cuestión que comprenda cebadores y/o sondas, las secuencias de nucleótidos de las que al menos tienen 10 nucleótidos de longitud que hibridan con cualquier ácido nucleico definido en la reivindicación 13, y que hibrida con uno o más MREJ de los tipos seleccionados de iv a ix.
18. La composición de la reivindicación 17, que además comprende cebadores y/o sondas, que hibridan con uno o más tipos de MREJ seleccionados de i a iii.
19. La composición de la reivindicación 18 ó 19, en donde los pares de cebadores tienen las secuencias de nucleótidos en la ID de SEC Nº:


20. La composición de la reivindicación 18, que además comprende sondas, cuyas ID de SEC nº son: 32, 83, 84, 160, 161, 162, 163, 164.



Patentes similares o relacionadas:
MICROORGANISMOS MARCADOS Y MÉTODOS PARA MARCAR, del 6 de Febrero de 2012, de DANISCO A/S: Un método para marcar una bacteria que comprende las etapas de: (a) exponer una bacteria original que comprende un locus CRISPR a un bacteriófago […]
DETECCIÓN, IDENTIFICACIÓN Y DIFERENCIACIÓN DE TAXONES EUBACTERIANOS EMPLEANDO UN ENSAYO DE HIBRIDACIÓN, del 13 de Enero de 2012, de INNOGENETICS N.V. ROCHE DIAGNOSTICS GMBH: Un conjunto de dos o tres sondas de polinucleótidos, donde dichas sondas se hibridan específicamente con SEQ ID NO 1 o SEQ ID NO 2, o con su forma de ARN, donde T se […]
MÉTODO DE DIAGNÓSTICO Y DE SEGUIMIENTO DE UNA VAGINOSIS BACTERIANA MEDIANTE CUANTIFICACIÓN MOLECULAR, del 28 de Diciembre de 2011, de UNIVERSITE DE LA MEDITERRANEE, AIX-MARSEILLE II: Método de diagnóstico y de seguimiento in vitro del estado de la flora bacteriana vaginal frente a la presencia de una vaginosis bacteriana […]
IDENTIFICACIÓN DE PATÓGENOS, del 14 de Diciembre de 2011, de AIT AUSTRIAN INSTITUTE OF TECHNOLOGY GMBH: Procedimiento de identificación de patógenos microbianos, preferiblemente patógenos humanos, en una muestra de fluido corporal, que comprende a) proporcionar una muestra de […]
DETECCIÓN DE GÉRMENES ASOCIADOS A PERIODONTITIS, del 9 de Diciembre de 2011, de GREINER BIO-ONE GMBH: Procedimiento para la detección y/o para la determinación de bacterias asociadas a periodontitis de una muestra biológica, que comprende las etapas i) obtención […]
SECUENCIAS GENOMICAS DE CEBADORES Y SONDA PARA LA CUANTIFICACION DE ADENOVIRUS PORCINOS (PADV), del 30 de Junio de 2011, de UNIVERSIDAD DE BARCELONA: Secuencias genómicas de cebadores y sonda para la cuantificación de adenovirus porcinos (PAdV).La invención se refiere a un test sensible para la detección de […]
 OLIGONUCLEÓTIDOS PARA LA DETECCIÓN DE SALMONELLA, del 3 de Junio de 2011, de INSTITUT PASTEUR INSTITUT NATIONAL DE LA SANTE ET DE LA RECHERCHE MEDICALE (INSERM): Par de cebadores oligonucleotídicos caracterizado porque consiste en dos oligonucleótidos de por lo menos 9 nucleótidos que hibridan en condiciones astringentes con la […]
OLIGONUCLEÓTIDOS PARA LA DETECCIÓN DE SALMONELLA, del 3 de Junio de 2011, de INSTITUT PASTEUR INSTITUT NATIONAL DE LA SANTE ET DE LA RECHERCHE MEDICALE (INSERM): Par de cebadores oligonucleotídicos caracterizado porque consiste en dos oligonucleótidos de por lo menos 9 nucleótidos que hibridan en condiciones astringentes con la […]
SECUENCIAS UTILIZADAS PARA LA DETECCIÓN E IDENTIFICACIÓN DE STAPHYLOCOCCUS AUREUS RESISTENTE A LA METICILINA (SARM) DE TIPO AEDM XI, del 16 de Mayo de 2011, de GENEOHM SCIENCES, INC.: Método para la detección de la presencia o ausencia de una cepa de Staphylococcus aureus resistente a la meticilina de tipo xi AEDM caracterizado […]