OLIGONUCLEÓTIDOS PARA LA DETECCIÓN DE SALMONELLA.
Par de cebadores oligonucleotídicos caracterizado porque consiste en dos oligonucleótidos de por lo menos 9 nucleótidos que hibridan en condiciones astringentes con la secuencia nucleotídica que corresponde a la parte Cterminal del gen iagA definda entre las posiciones 1318 y 1755 de la secuencia iagA representada en la figura 1,
siendo dicho par capaz de amplificar una secuencia de ADN o de ADNc de Salmonella enterica y de Salmonella bongori comprendida en la secuencia iagA
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E04078201.
Solicitante: INSTITUT PASTEUR
INSTITUT NATIONAL DE LA SANTE ET DE LA RECHERCHE MEDICALE (INSERM).
Nacionalidad solicitante: Francia.
Dirección: 25-28, RUE DU DOCTEUR ROUX 75724 PARIS CEDEX 15 FRANCIA.
Inventor/es: POPOFF, MICHEL, YVAN, LE GUERN FELLOUS, MURIEL.
Fecha de Publicación: .
Fecha Solicitud PCT: 15 de Enero de 1996.
Clasificación Internacional de Patentes:
- C12Q1/68M10B
Clasificación PCT:
- C07K14/255 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › Salmonella (G).
- C12Q1/68 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C07H21/04 C07 […] › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con desoxirribosilo como radical sacárido.
- C07K14/255 C07K 14/00 […] › Salmonella (G).
- C12P19/34 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda.
PDF original: ES-2360344_T3.pdf




Fragmento de la descripción:
El género Salmonella contiene dos especies, Salmonella enterica, especie dividida en seis subespecies basándose en características bioquímicas y de homología a nivel del ADN, y Salmonella bongori. El género se subdivide en más de 2.000 serovariedades definidas con ayuda de antígenos somáticos y flagelares. Las bacterias del género Salmonella son generalmente patógenas para el animal o para el ser humano. Así se sabe que las Salmonella se encuentran entre los agentes responsables de las intoxicaciones alimentarias más frecuentes en los países desarrollados: por esta razón, son importantes métodos de detección rápidos y fiables de las subespecies de
Salmonella.
Las salmonelas responsables de las toxiinfecciones alimentarias pertenecen principalmente a la subespecie I (también denominada grupo I) de S. enterica.
No obstante, las toxiinfecciones no son las únicas patologías provocadas por infecciones de Salmonella.
Por ejemplo, Salmonella enterica subespecie enterica serovariedad typhi (a continuación denominada Typhi) es el agente que causa la fiebre tifoidea humana.
Teniendo en cuenta la naturaleza de las infecciones provocadas por las salmonelas y la necesidad especialmente de estudiar su presencia en las muestras biológicas obtenidas de los pacientes o en los alimentos, parece indispensable disponer de medios rápidos y sensibles para detectar su presencia.
Los métodos habituales de cultivo ampliamente utilizados hasta la fecha para la detección de las salmonelas requieren un tiempo importante y no están adaptados por ejemplo para hacer un seguimiento de la contaminación de productos alimentarios. Para vencer las desventajas de estos métodos, ya se han propuesto varios métodos basados en técnicas de biología molecular tales como pruebas de hibridación y las pruebas de reacción en cadena de la polimerasa. Se han utilizado diferentes sondas de ADN en varios protocolos de hibridación y de PCR para detectar las subespecies de Salmonella en la alimentación. Sin embargo, ninguna de estas técnicas es completamente satisfactoria, ya que las secuencias utilizadas no se conocen completamente o no están exclusivamente presentes en el género Salmonella y de esta forma, pueden conducir a reacciones cruzadas entre la sonda y secuencias de ADN de otras enterobacterias o pueden conducir a un gran número de falsos negativos o de falsos positivos.
Los inventores han buscado medios que permiten la detección específica y sensible del conjunto de las salmonelas de las especies S. enterica y/o S. Bongori. Desde este punto de vista, se han interesado por la cepa de Salmonella enterica subespecie enterica serovariedad typhi (S. typhi) y por el gen que participa en la invasión de células por S. typhi.
Además, han definido determinadas condiciones que permiten la detección específica de grupos determinados de salmonelas, por ejemplo, bacterias del grupo I.
En el estado anterior de la técnica, ya se ha demostrado que la cepa Typhi puede adherirse a monocapas de células HeLa y de entrar en estas células (Yabuuchi et al, 1986). Sin embargo, hasta la fecha, no se han identificado claramente los determinantes genéticos implicados en este proceso de adhesión y de entrada en las células. Elsinghorst et al. (1989) han clonado un fragmento cromosómico de Typhi, que confiere a bacterias de tipo Escherichia coli la capacidad de penetrar en las células Henle 407. Recientemente, se han identificado y clonado otra región cromosómica implicada en la invasión de las células HeLa por la cepa Typhi Ty2 (Popoff y Dion, 1990).
Los inventores de la presente solicitud han identificado en un fragmento de ADN de 2,4 kb de S. typhi contenido en la secuencia HindIII de 7,9 kb descrita por Popoff y Dion (1990), regiones susceptibles de participar en la actividad de invasión de Salmonella enterica subespecie enterica serovariedad Typhi en células, y en participar en cultivos celulares de tipo HeLa, siendo estas regiones susceptibles además de ser utilizadas en reacciones para la realización de un diagnóstico generalizado de todos los representantes de las especies S. enterica y/o S. bongori o probablemente en condiciones de detección particulares, para el diagnóstico específico del grupo I de S. enterica.
Los inventores han identificado una secuencia denominada IagA y una secuencia denominada IagB y las han caracterizado por su participación en la invasión celular manifestándose durante una infección por Salmonella enterica subespecie enterica serovariedad Typhi.
La especificidad de estas secuencias en el seno de S. typhi ha hecho que los inventores propongan su utilización para definir unos medios para el diagnóstico de una infección por S. typhi, incluso para el diagnóstico de una infección por Salmonella de las especies S. enterica y/o S. bongori o en determinados casos para poner en evidencia S. enterica de grupos específicos.
Estos medios que se pueden utilizar para el diagnóstico de una infección por Salmonella enterica y/o Salmonella bongori comprenden unos oligonucleótidos susceptibles de ser utilizados en reacciones de amplificación de secuencias de nucleótidos, por ejemplo, reacciones en cadena de la polimerasa. La solicitud describe asimismo unas sondas para la detección de ácidos nucleicos de S. enterica y/o S. bongori, siendo estos ácidos nucleicos, si procede, unos fragmentos amplificados.
La invención tiene asimismo por objeto un kit y un método de detección de la presencia de Salmonella enterica y Salmonella bongori en muestras biológicas y por ejemplo, en productos alimentarios o en cualquier muestra que sea el objeto de un diagnóstico clínico. Otros métodos y kits de detección son específicos de las cepas del grupo I de S. enterica.
Estos otros métodos permiten, por el contrario, investigar la presencia de bacterias S. enterica o S. bongori del género Salmonella. El género Salmonella incluye así seis subespecies o grupos I, II, III, IV, V o VI. Las subespecies I, II, III, IV y VI pertenecen a la especie S. enterica y la subespecie V pertenece a la especie S. bongori.
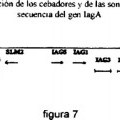
La solicitud describe asimismo las secuencias de nucleótidos que participan en la invasión de células por Salmonella enterica subespecie enterica serovariedad Typhi, caracterizadas porque se trata de una de las secuencias iagA o iagB comprendidas respectivamente entre los nucleótidos 97 y 1755 de la secuencia representada en la figura 1 (IagA) y entre los nucleótidos 1776 y 2255 de la secuencia representada en la figura 1 (IagB).
La solicitud describe asimismo unas secuencias de nucleótidos modificadas con respecto a iagA o iagB pero que presentan sin embargo las mismas propiedades al tratarse de la invasión de las células, o que se hibridan en condiciones astringentes con una de dichas secuencias.
La presente solicitud describe asimismo unas proteínas IagA e IagB que responden a las secuencias presentadas en la figura 1 o unas variantes de estas secuencias obtenidas por mutación, deleción o adición de aminoácidos por cuanto que la secuencia así obtenida es reconocida por unos anticuerpos dirigidos contra una de dichas secuencias IagA o IagB.
De manera general, la solicitud describe cualquier secuencia de aminoácidos codificada por los genes iagA e iagB representados en la figura 1.
La solicitud describe por otra parte cualquier fragmento de una de estas secuencias, en particular cualquier fragmento en forma purificada, suficiente para conservar en S. typhi sus propiedades de adhesión y de infección de las células, y en particular de las células HeLa en cultivo.
El procedimiento de infección de las células HeLa en cultivo es el procedimiento habitual que se ha descrito en particular en la solicitud de patente internacional publicada con el número WO 92/01056.
La invención se refiere a unos medios para la detección de la presencia de S. enterica y S. bongori y en su caso, para la cuantificación de S. enterica y S. bongori en muestras biológicas.
Por muestra biológica, se entiende cualquier muestra extraída para la realización de análisis in vitro en el animal o en el ser humano o extraída a partir de productos alimenticios con independencia de la naturaleza o a partir de cualquier medio líquido, sólido o gaseoso susceptible de contener los agentes patogénicos buscados.
La invención tiene por objeto en este contexto, un par de cebadores tal como se define en la reivindicación 1.
Reivindicaciones:
1. Par de cebadores oligonucleotídicos caracterizado porque consiste en dos oligonucleótidos de por lo menos 9 nucleótidos que hibridan en condiciones astringentes con la secuencia nucleotídica que corresponde a la parte C-terminal del gen iagA definda entre las posiciones 1318 y 1755 de la secuencia iagA representada en la figura 1, siendo dicho par capaz de amplificar una secuencia de ADN o de ADNc de Salmonella enterica y de Salmonella bongori comprendida en la secuencia iagA.
2. Par de cebadores según la reivindicación 1, caracterizado porque las condiciones de hibridación astringentes son 50ºC en la mezcla de reacción siguiente: KCl 50 mM, Tris-HCl 10mM pH 8,3, MgCl2 1,5 mM, dCTP, dATP y dGTP 125 µM, 250 µM de dUTP, 25 pmoles de cada uno de los cebadores, 1 unidad de Uracilo N Glucosilasa y 1U de Taq.
3. Kit para la detección de la presencia de Salmonella enterica y/o de Salmonella bongori en muestras biológicas, que comprende:
- un par de cebadores oligonucleotídicos según la reivindicación 1 ó 2, -una sonda para la detección de los fragmentos amplificados, y -los reactivos necesarios para la realización de la reacción de amplificación.
4. Utilización de un par de cebadores según la reivindicación 1 para la amplificación de secuencias de nucleótidos de bacterias del género Salmonella especies enterica y bongori.
5. Utilización de un par de cebadores oligonucleotídicos según la reivindicación 1, para la detección tras la amplificación del ADN genómico o del ADNc, de bacterias del género Salmonella especies enterica y bongori.
6. Utilización según la reivindicación 4 ó 5, en la que las condiciones de hibridación astringentes son 50ºC en la mezcla de reacción siguiente: KCl 50 mM, Tris-HCl 10mM pH 8,3, MgCl2 1,5 mM, dCTP, dATP y dGTP 125 µM, 250 µM de dUTP, 25 pmoles de cada uno de los cebadores, 1 unidad de Uracilo N Glucosilasa y 1U de Taq.
Patentes similares o relacionadas:
MICROORGANISMOS MARCADOS Y MÉTODOS PARA MARCAR, del 6 de Febrero de 2012, de DANISCO A/S: Un método para marcar una bacteria que comprende las etapas de: (a) exponer una bacteria original que comprende un locus CRISPR a un bacteriófago […]
DETECCIÓN, IDENTIFICACIÓN Y DIFERENCIACIÓN DE TAXONES EUBACTERIANOS EMPLEANDO UN ENSAYO DE HIBRIDACIÓN, del 13 de Enero de 2012, de INNOGENETICS N.V. ROCHE DIAGNOSTICS GMBH: Un conjunto de dos o tres sondas de polinucleótidos, donde dichas sondas se hibridan específicamente con SEQ ID NO 1 o SEQ ID NO 2, o con su forma de ARN, donde T se […]
MÉTODO DE DIAGNÓSTICO Y DE SEGUIMIENTO DE UNA VAGINOSIS BACTERIANA MEDIANTE CUANTIFICACIÓN MOLECULAR, del 28 de Diciembre de 2011, de UNIVERSITE DE LA MEDITERRANEE, AIX-MARSEILLE II: Método de diagnóstico y de seguimiento in vitro del estado de la flora bacteriana vaginal frente a la presencia de una vaginosis bacteriana […]
IDENTIFICACIÓN DE PATÓGENOS, del 14 de Diciembre de 2011, de AIT AUSTRIAN INSTITUTE OF TECHNOLOGY GMBH: Procedimiento de identificación de patógenos microbianos, preferiblemente patógenos humanos, en una muestra de fluido corporal, que comprende a) proporcionar una muestra de […]
DETECCIÓN DE GÉRMENES ASOCIADOS A PERIODONTITIS, del 9 de Diciembre de 2011, de GREINER BIO-ONE GMBH: Procedimiento para la detección y/o para la determinación de bacterias asociadas a periodontitis de una muestra biológica, que comprende las etapas i) obtención […]
SECUENCIAS GENOMICAS DE CEBADORES Y SONDA PARA LA CUANTIFICACION DE ADENOVIRUS PORCINOS (PADV), del 30 de Junio de 2011, de UNIVERSIDAD DE BARCELONA: Secuencias genómicas de cebadores y sonda para la cuantificación de adenovirus porcinos (PAdV).La invención se refiere a un test sensible para la detección de […]
SECUENCIAS UTILIZADAS PARA LA DETECCIÓN E IDENTIFICACIÓN DE STAPHYLOCOCCUS AUREUS RESISTENTE A LA METICILINA (SARM) DE TIPO AEDM XI, del 16 de Mayo de 2011, de GENEOHM SCIENCES, INC.: Método para la detección de la presencia o ausencia de una cepa de Staphylococcus aureus resistente a la meticilina de tipo xi AEDM caracterizado […]
Vacunas de Salmonella no tifoidea, del 23 de Noviembre de 2016, de GSK Vaccines Institute for Global Health S.r.l: Un polipéptido inmunogénico que comprende o consiste en: (i) un polipéptido que es una OmpD de Salmonella no tifoidea, o un fragmento inmunogénico de la misma que tiene […]