DISPOSITIVO INTERFASE DIGITAL PARA LA INTEGRACION DEL MODO DE IMAGEN EN ESPECTROSCOPIO DE FUERZA MONOMOLECULAR.
Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular.
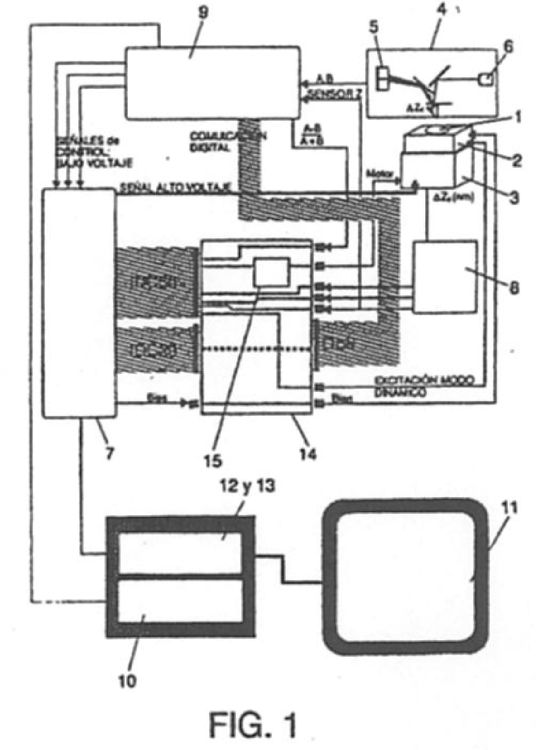
Cuenta con un cabezal de detección de AFM (4) conectado a un controlador de espectroscopia (9) que se une a una tarjeta de obtención de datos (10) instalada en un ordenador (11) unido a un bloque procesador DSP (12) que conecta con un bloque de conversión DAC-ADC (13). La muestra a analizar (1) se monta en un elemento oscilante (2) colocado sobre un posicionador 3D (3) empujado por un motor y unido a un sensor de posición (8) con sensores en tres direcciones. Un controlador de imagen (7) conecta con la tarjeta (10), el bloque de conversión (13), el sensor de posición (8), el posicionador (3), el elemento oscilante (2), el cabezal de detección (4) y el controlador de espectroscopia (9), facilitando el conexionado una caja integradora. El cabezal de detección (4) se ubica en la parte superior de una mesa antivibraciones
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P200701859.
Solicitante: CONSEJO SUPERIOR DE INVESTIGACIONES CIENTIFICAS
UNIVERSIDAD AUTONOMA DE MADRID.
Nacionalidad solicitante: España.
Provincia: MADRID.
Inventor/es: CARRION VAZQUEZ,MARIANO SIXTO, VALBUENA JIMENEZ,ALEJANDRO, GOMEZ HERRERO,JULIO.
Fecha de Solicitud: 2 de Julio de 2007.
Fecha de Publicación: .
Fecha de Concesión: 18 de Enero de 2010.
Clasificación Internacional de Patentes:
- G01N13/16
Clasificación PCT:
- G01Q60/24 FISICA. › G01 METROLOGIA; ENSAYOS. › G01Q TECNICAS O APARATOS DE SONDA DE BARRIDO; APLICACIONES DE TECNICAS DE SONDA DE BARRIDO, p. ej. MICROSCOPIA POR SONDA DE BARRIDO [SMP]. › G01Q 60/00 Tipos particulares de microscopía por sonda de barrido [SPM] o aparatos empleados; Componentes esenciales al efecto. › AFM [Microscopía de Fuerza Atómica] o aparatos empleados, p. ej. sondas AFM.
Fragmento de la descripción:
Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular.
Objeto de la invención
La presente invención, tal y como se expresa en el enunciado de esta memoria descriptiva, se refiere a un dispositivo interfase digital para la integración del modo de imagen en la espectroscopia de fuerza monomolecular, cuya finalidad esencial consiste en proporcionar una herramienta sencilla, fiable, estable y de fácil implementación con la característica novedosa de añadir capacidades de formación de imágenes a un procedimiento standard muy usado en nanomecánica de proteínas. Dicha herramienta o instrumento se consigue básicamente mediante la interconexión de un controlador de imagen y otros bloques funcionales electrónicos a un microscopio de fuerza atómica (AFM, de sus siglas en inglés) y con determinados programas informáticos. El instrumento resultante combina las potentes capacidades de la última generación de dispositivos de espectroscopia de fuerza con toda la variedad de modos de formación de imágenes disponibles: por contacto, dinámico y por saltos. Usando un sistema modelo de poliproteínas (dominio I27 de la titina) se demuestra la capacidad de formación de imágenes, nanoposicionamiento y medición de fuerza casi simultáneas del dispositivo de la invención.
En la mayoría de estudios sobre nanomecánica de proteínas (medición de sus propiedades mecánicas a escala atómica) se utiliza el microscopio de fuerza atómica (AFM) en su modalidad de medición de fuerzas, es decir actuando como espectroscopio de fuerzas. Mediante la presente invención se añade la capacidad de formación de imágenes al dispositivo AFM en su modalidad de espectroscopio de fuerza y se integran programas de análisis potentes y disponibles para ambas modalidades. Mediante el dispositivo de la invención, único en su género es posible lograr un análisis de espectroscopia de fuerza y la formación de imágenes para moléculas individuales de manera casi simultánea y con una alta resolución.
Además, mediante la herramienta proporcionada por el dispositivo de la invención se pretende facilitar el desarrollo de un sistema de funcionalización universal muy necesario para permitir un mejor control de la muestra y una eficacia mejorada de inmovilización de proteínas necesaria para realizar los análisis con AFM.
Antecedentes de la invención
El microscopio de fuerza atómica (AFM) es un aparato que permite medir propiedades mecánicas de los materiales con resolución atómica [1]. La principal aplicación de este instrumento en biología ha sido la formación de imágenes superficiales a alta resolución de estructuras biológicas en su entorno acuoso natural [2]. Además, de esto se puede obtener información mecánica importante de la muestra usando el AFM en la denominada configuración de espectroscopia de fuerza [3].
El fundamento básico del AFM es la medición de la deflexión de un fleje a través de un láser reflejado en la superficie de este fleje a la vez que se desplaza la muestra a analizar en las tres direcciones del espacio (gracias a la actuación de un posicionador piezo-eléctrico) para lograr así la interacción de la punta fina del fleje con la muestra en las tres dimensiones. La deflexión de este fleje o sensor se detecta a través de la medida en el voltaje de un fotodiodo dual, de manera que cuando el fleje está en equilibrio la cantidad de luz sobre las dos partes del fotodiodo es la misma y la fuerza (diferencia de voltaje generador en cada sector del fotodiodo) será O. La interacción de la punta del fleje con la superficie de la molécula hace que el mismo se flexiona, provocando que el fotodiodo detecte más luz en un sector que en otro. De esta manera se obtiene una señal en voltaje que está relacionada con la fuerza ejercida en la punta por la muestra bajo estudio. Otra parte básica de un microscopio de fuerzas es la señal de alto voltaje enviada a un posicionador piezo-eléctrico para mover la muestra bajo la punta y poder escanear la superficie. Por su parte, en un microscopio de fuerzas convencional la unidad generadora de alto voltaje que envía las señales al posicionador piezo-eléctrico y el controlador de dicho posicionador pueden encontrarse integrados en un mismo bloque o no. Así, por ejemplo el sistema de control Dulcinea (Nanotec Electrónica, S.L.) integra ambos elementos, mientras el sistema inicial desde el que se ha desarrollado la presente invención [7], mantenía ambos elementos separados, en este caso una controladora JRC Instruments y un amplificador de alto voltaje de Physik Instruments. El último componente básico es un ordenador provisto de tarjetas de adquisición de datos y de un software específico para procesar digitalmente las señales de la fuerza normal (la proveniente del fotodiodo) y las rampas que van al amplificador y después al piezoeléctrico. Este procesado permite representar los datos en bruto (espectro de fuerzas) o visualizar la topografía de la muestra como una imagen.
Puesto que el AFM estaba originalmente diseñado para registrar curvas de fuerza-extensión incluye un único posicionador piezoeléctrico, por lo que la tecnología más avanzada en el campo de los "espectrómetros de fuerza" se basa en el diseño "por encargo" de AFMs especializados en la extensión de las moléculas en una única dimensión, lo que permite su análisis individual [4].
En esta denominada modalidad de espectroscopia de fuerza de moléculas individuales (SMFS, de sus siglas en inglés), el AFM es capaz de medir fuerzas con una sensibilidad de decenas de piconewtons y cambios de longitud en las moléculas con resolución nanométrica, en una escala de tiempo en el rango de milisegundos. Estos instrumentos han permitido el análisis de eventos de despliegue y repliegue de moléculas individuales en proteínas modulares [5] y, con el uso de poliproteínas recombinantes (que permiten una identificación clara de moléculas individuales), en módulos proteicos individuales [6] así como en proteínas no modulares. Por consiguiente, la aplicación de la SMFS al estudio de las proteínas se ha extendido rápidamente de manera que ha surgido un nuevo campo de investigación, denominado "nanomecánica de proteínas". Es evidente sin embargo que estos avances también tienen potencial en el análisis de moléculas no proteicas, por lo que podríamos hablar de nanomecánica molecular.
Tras sus primeras aplicaciones al estudio de las proteínas [5, 6] la SMFS ha evolucionado gracias a una segunda generación de instrumentos que incorpora un posicionador 3D compacto con resolución subnanométrica (aproximadamente 0,5 nm), el modo de fuerza constante y un potente software, permitiendo funcionalidades tales como el funcionamiento semiautomático, el reconocimiento automático de patrones periódicos de poliproteínas y el establecimiento de protocolos específicos para despliegue y repliegue de proteínas. Estas características y funciones se han integrado actualmente mediante un paquete de software específico basado en IGRO Pro, que es de acceso público [7]. Si bien no existe propiamente un dispositivo para SMFS con estas características en el mercado, si lo están todos sus compo-nentes, de manera que diversos grupos ya han establecido esta configuración de manera satisfactoria [8, 9, 10, 11].
Actualmente los análisis de tipo SMFS deben afrontar tres importantes restricciones: la falta de conocimiento del estado inicial de la muestra antes del experimento, la aleatoriedad en la "recogida" o selección de moléculas para cada observación y el que esta "recogida" tenga una eficacia reducida, pues a menudo no es posible seleccionar moléculas individuales. Asimismo, debe tomarse en consideración que, tras la inmovilización sobre una superficie, necesaria para su análisis mediante el AFM, las moléculas biológicas, y en particular las proteínas pueden desnaturalizarse en cierto grado [12]. Por lo tanto, en un experimento de SMFS normal, la muestra (generalmente una proteína purificada por cromatografía de afinidad) puede estar constituida por una población heterogénea de especies moleculares, que incluye moléculas individuales (nativas, parcialmente desnaturalizadas, completamente desnaturalizadas y plegadas incorrectamente), complejos supramoleculares, agregados y moléculas contaminantes. Si bien el uso de poliproteínas (en las que una única molécula proteica incluye diversas proteínas con funcionalidades individuales) es muy útil para realizar análisis de módulos proteicos individuales e inferir la identificación de moléculas individuales, su uso no siempre puede excluir todas estas posibles fuentes de error.
Reivindicaciones:
1. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, que cuenta con un cabezal de detección de AFM (microscopio de fuerza atómica) de vista superior (4) provisto de fotodiodo (5) y láser (6), y que se aplica a una muestra a analizar (1) ubicada en un posicionador piezo-eléctrico 3D (3); caracterizado porque dicho cabezal de detección (4) se conecta con un controlador de la función de espectroscopia de fuerza (9) unido a una o más tarjetas de obtención de datos (10) que se instalan en un ordenador (11) unido a un bloque procesador de señal digital (DSP) (12) que conecta con un bloque de conversión digital-analógico/analógico-digital (13); en tanto que la muestra a analizar (1) se dispone sobre un elemento piezo- eléctrico oscilante (2) que se coloca en la parte superior del posicionador piezo-eléctrico 3D (3) y que hace oscilar la muestra (1) para poder obtener imágenes en modo dinámico, empujando al posicionador (3) un motor acoplado a un tornillo micrométrico para facilitar y automatizar la aproximación entre la punta del cabezal (4) y la muestra (1); conectándose este posicionador (3) a un sensor de posición (8) que cuenta con sensores capacitivos en tres direcciones para conocer con gran precisión la posición del posicionador (3) y poder hacer correcciones de linearización del desplazamiento del mismo; incluyéndose además un controlador de imagen (7) que conecta con la tarjeta de obtención de datos (10), con el bloque de conversión (13) y con el sensor de posición (8); conectando además este controlador de imagen (7) con el posicionador 3D (3), con el elemento oscilante (2) y con el cabezal de detección (4); existiendo también conexiones entre este controlador de imagen (7) y el controlador de la función de espectroscopia de fuerza (9); facilitándose el conexionado mediante una caja integradora (14) que permite que las electrónicas de los dos controladores (7 y 9) puedan actuar sobre la parte mecánica del AFM consistente en el cabezal del microscopio (4) y el posicionador piezo-eléctrico (3); y disponiéndose el cabezal de detección (4) en la parte superior de una mesa de aislamiento de vibraciones.
2. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, según la reivindicación 1, caracterizado porque el cabezal de detección (4) es un cabezal multimodo de AFM, fotodetector de cuatro cuadrantes que se utiliza como fotodetector de dos semicírculos en el dispositivo.
3. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, según la reivindicación 1, caracterizado porque en el cabezal de detección (4) el sistema láser original se encuentra sustituido por un sistema de haces láser más estrecho y más estable.
4. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, según la reivindicación 1, caracterizado porque el posicionador piezo-eléctrico 3D (3) es un posicionador piezo-eléctrico de bucle cerrado de varios ejes equipado con un sensor capacitivo rápido de la posición del accionador que permite una resolución subnanométrica.
5. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, según la reivindicación 1, caracterizado porque el controlador de imagen (7) incluye amplificadores de alto voltaje cuya entrada consiste exclusivamente en la salida analógica de los DACs del bloque de conversión (13); mientras que las señales analógicas que genera la tarjeta de obtención de datos (10) son digitalizadas antes de ser tenidas en cuenta por el bloque procesador (DSP) (12).
6. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, según la reivindicación 1, caracterizado porque los movimientos piezo-eléctricos del elemento oscilante (2) están codirigidos por el controlador de la función de espectroscopia de fuerza (9) y por el controlador de imagen (7); introduciéndose las señales de bajo voltaje de la tarjeta de obtención de datos (10) en el bloque procesador (DSP) (12) a través de tres canales diferentes del convertidor analógico-digital ADC del bloque de conversión (13).
7. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, según la reivindicación 1, caracterizado porque el elemento oscilante (2) incluye unas bias que posibilitan experimentos con potenciales en superficie, medidas de corriente, electroquímica y otros.
8. Dispositivo interfase digital para la integración del modo de imagen en espectroscopio de fuerza monomolecular, según la reivindicación 1, caracterizado porque la referida caja integradora (14) incluye un buffer (15) para el aludido motor.
Patentes similares o relacionadas:
 METODO DE UTILIZACION DE UN MICROSCOPIO DE FUERZAS ATOMICAS Y MICROSCOPIO, del 1 de Diciembre de 2007, de CONSEJO SUPERIOR INVESTIGACIONES CIENTIFICAS: Método de utilización de un microscopio de fuerzas atómicas y microscopio.#El método que comprende realizar como mínimo una excitación bi-modal de una micropalanca […]
METODO DE UTILIZACION DE UN MICROSCOPIO DE FUERZAS ATOMICAS Y MICROSCOPIO, del 1 de Diciembre de 2007, de CONSEJO SUPERIOR INVESTIGACIONES CIENTIFICAS: Método de utilización de un microscopio de fuerzas atómicas y microscopio.#El método que comprende realizar como mínimo una excitación bi-modal de una micropalanca […]
INSTRUMENTO DE CARACTERIZACION ELECTRICA A ESCALA NANOMETRICA., del 1 de Abril de 2006, de UNIVERSITAT AUTONOMA DE BARCELONA: Comprende un microscopio de fuerzas atómicas (AFM) que incluye un dispositivo de barrido, al menos una punta, un soporte para la muestra, y medios […]
 PROCEDIMIENTO DE SECUENCIACION DE POLINUCLEOTIDOS, del 1 de Junio de 2008, de MEDICAL BIOSYSTEMS LTD.: Un procedimiento para determinar la secuencia de un polinucleótido, que comprende las etapas de: (i) poner en contacto una enzima de tratamiento de polinucleótidos, […]
PROCEDIMIENTO DE SECUENCIACION DE POLINUCLEOTIDOS, del 1 de Junio de 2008, de MEDICAL BIOSYSTEMS LTD.: Un procedimiento para determinar la secuencia de un polinucleótido, que comprende las etapas de: (i) poner en contacto una enzima de tratamiento de polinucleótidos, […]
MÉTODO INTEGRADO QUE UTILIZA MICROSCOPÍA DE FUERZA ATÓMICA PARA DETERMINAR SIMULTÁNEAMENTE LAS PROPIEDADES BIOMECÁNICAS (COMO VISCOELASTICIDAD) DE O CÉLULAS VIVAS Y TOPOGRAFÍA DE BIOMATERIALES, del 27 de Junio de 2019, de PONTIFICIA UNIVERSIDAD CATÓLICA DE CHILE: La presente invención corresponde a un método para determinar biocompatibilidad entre células vivas y biomateriales, utilizando Microscopía de Fuerza […]
PROCESO POR MEDIO DE MICROSCOPÍA DE FUERZA ATÓMICA PARA EL ANÁLISIS MASIVO FÍSICO Y MECÁNICO EN MATERIALES, ARREGLOS DE BIOMATERIALES Y ESTRUCTURAS, del 13 de Junio de 2019, de INSTITUTO POLITÉCNICO NACIONAL: La presente invención se refiere a un proceso implementado por computadora que consiste en etapas de fijado preciso, manipulación y mediciones masivas por microscopía […]