Método de preparación de bibliotecas de polinucleótidos molde.
Un método para generar una biblioteca de moléculas polinucleotídicas molde que tienen secuencias comunes en sus extremos 5' y secuencias comunes en sus extremos 3',
comprendiendo el método:
ligar polinucleótidos adaptadores con apareamiento erróneo idénticos en ambos extremos de cada uno de uno o más dúplex de polinucleótido diana para formar una o más construcciones adaptador-diana, en el que cada adaptador con apareamiento erróneo se forma a partir de dos cadenas polinucleotídicas hibridadas que forman un complejo biomolecular que comprende al menos una región bicatenaria y una región no apareada, y
realizar una reacción de extensión con cebador inicial en la que un oligonucleótido cebador se hibrida con una parte de adaptador de cada una de las construcciones adaptador-diana y se extiende por adición secuencial de nucleótidos para formar productos de extensión complementarios a al menos una cadena de cada una de las construcciones adaptador-diana,
en el que los productos de extensión, y opcionalmente productos de amplificación derivados de los mismos, proporcionan en su conjunto una biblioteca de moléculas polinucleotídicas molde que tienen secuencias comunes en sus extremos 5' y secuencias comunes en sus extremos 3';
en el que los dúplex de polinucleótido diana que van a ligarse son una mezcla compleja de fragmentos de ADN genómico que representan un genoma completo o sustancialmente completo, y en el que la biblioteca de moléculas polinucleotídicas molde generadas es representativa del genoma completo o sustancialmente completo.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2006/004055.
Solicitante: ILLUMINA CAMBRIDGE LIMITED.
Nacionalidad solicitante: Reino Unido.
Dirección: Chesterford Research Park Little Chesterford Saffron Walden Essex CB10 1XL REINO UNIDO.
Inventor/es: SMITH,GEOFFREY PAUL, RIGATTI,ROBERTO, GORMLEY,NIALL ANTHONY, BENTLEY,DAVID.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2450047_T3.pdf
Fragmento de la descripción:
Método de preparación de bibliotecas de polinucleótidos molde
Campo de la invención [0001] La invención se refiere a un método de preparación de una biblioteca de polinucleótidos molde y también al uso de la biblioteca de moldes en métodos de amplificación de ácidos nucleicos en fase sólida. En particular, la invención se refiere a un método de preparación de una biblioteca de polinucleótidos molde que tienen secuencias comunes en sus extremos 5’ y en sus extremos 3’.
Antecedentes de la invención [0002] El desarrollo de fármacos de biología molecular y farmacológica actual hace un uso intensivo de análisis de ácidos nucleicos. Las áreas más conflictivas son las de secuenciación del genoma completo, detección de polimorfismo de un solo nucleótido, exploración y control de la expresión de genes, que típicamente requieren análisis de grandes cantidades de ácido nucleico.
Un área de la tecnología que revolucionó el estudio de los ácidos nucleicos fue el desarrollo de las técnicas de amplificación de ácidos nucleicos, tales como la reacción en cadena de la polimerasa (PCR) . Las reacciones de amplificación, tales como PCR, pueden permitir al usuario amplificar específica y selectivamente un ácido nucleico diana particular de interés a partir de una mezcla compleja de ácidos nucleicos. Sin embargo, existe una necesidad constante de técnicas de amplificación de ácidos nucleicos que permitan la amplificación simultánea de mezclas complejas de moldes de diversa secuencia, tales como fragmentos de ADN genómico (por ejemplo, amplificación de “genoma completo”) o bibliotecas de ADNc, en una única reacción de amplificación.
La amplificación por PCR no puede producirse en ausencia de hibridación de cebadores de amplificación directos e inversos para secuencias de unión a cebador en el molde a amplificar en las condiciones de las etapas de hibridación de la reacción PCR, es decir, si hay insuficiente complementariedad entre cebadores y molde. Un cierto conocimiento previo de la secuencia del molde se requiere por tanto antes de poder realizarse una reacción de PCR para amplificar un molde específico, salvo que se usen cebadores al azar con una pérdida consecuente de especificidad. El usuario debe normalmente conocer la secuencia de al menos los sitios de unión a cebador en el molde por adelantado para que puedan diseñarse los cebadores apropiados, aunque la secuencia restante del molde sea desconocida. La necesidad de conocimiento previo de la secuencia del molde aumenta la complejidad y el coste de la amplificación por PCR de mezclas complejas de moldes, tales como fragmentos de ADN genómico.
Los documentos WO 98/44151 y WO 00/18957 describen métodos de formación de series de polinucleótidos basadas en amplificación de ácidos nucleicos en “fase sólida”, que es análoga a una reacción en cadena de la polimerasa en la que los productos de amplificación están inmovilizados sobre un soporte sólido para formar series comprendidas por agrupaciones de ácido nucleico o “colonias”. Cada agrupación o colonia de dicha serie se forma a partir de una pluralidad de cadenas de polinucleótidos idénticas inmovilizadas y una pluralidad de cadenas de polinucleótidos complementarias inmovilizadas idénticas. Las series así formadas se denominan generalmente en el presente documento “series agrupadas” y sus características generales se entenderán haciendo referencia a los documentos WO 98/44151 o WO 00/18957.
Como se ha indicado anteriormente, los métodos de amplificación en fase sólida de los documentos WO 98/44151 y WO 00/18957 son esencialmente una forma de la reacción en cadena de la polimerasa realizada sobre un soporte sólido. Como cualquier reacción PCR, estos métodos requieren el uso de cebadores de amplificación directos e inversos (que pueden ser idénticos o diferentes) que pueden hibridarse con un molde a amplificar. En los métodos de los documentos WO 98/44151 y WO 00/18957 ambos cebadores se inmovilizan sobre el soporte sólido en el extremo 5’. Se conocen otras formas de amplificación en fase sólida en las que solo un cebador está inmovilizado y el otro está presente en la solución libre (Mitra, R. D y Church, G. M., Nucleic Acids Research, 1999, Vol.27, Nº 24) .
En común con todas las técnicas de PCR, la amplificación por PCR en fase sólida requiere el uso de cebadores de amplificación directos e inversos que incluyen secuencias de nucleótidos “específicas de molde” que pueden hibridarse con secuencias en el molde a amplificar, o su complemento, en condiciones de las etapas de hibridación de la reacción PCR. Las secuencias en el molde con el que los cebadores se hibridan en condiciones de la reacción PCR pueden denominarse en el presente documento secuencias “de unión a cebador”.
Determinadas realizaciones de los métodos descritos en los documentos WO 98/44151 y WO 00/18957 utilizan cebadores “universales” para amplificar moldes que comprenden una parte molde variable que se desea amplificar flanqueada en 5’ y 3’ por secuencias de unión a cebador común o “universal”. Los cebadores directo e inverso “universales” incluyen secuencias que pueden hibridarse con las secuencias de unión a cebador “universal” en la construcción molde. La parte molde variable puede en sí misma ser de secuencia conocida, desconocida o parcialmente desconocida. Esta estrategia tiene la ventaja de que no es necesario diseñar un par específico de cebadores para cada molde a amplificar; pueden utilizarse los mismos cebadores para la amplificación de diferentes moldes siempre que cada molde se modifique por la adición de las mismas secuencias de unión a cebador universal
en sus extremos 5’ y 3’. La secuencia molde variable puede por lo tanto ser cualquier fragmento de ADN de interés.
Una estrategia análoga puede usarse para amplificar una mezcla de moldes, tal como una pluralidad o biblioteca de moléculas de ácido nucleico molde (por ejemplo, fragmentos de ADN genómico) , usando un solo par de cebadores directos e inversos universales siempre que cada molécula molde en la mezcla se modifique por la adición de las mismas secuencias de unión de cebador universal.
Dichas estrategias de “cebador universal” para amplificación por PCR, y en particular amplificación por PCR en fase sólida, son ventajosas ya que permiten que puedan realizarse múltiples moléculas molde de secuencia igual
o diferente, conocida o desconocida, a amplificar en una única reacción de amplificación, que puede realizarse en un soporte sólido que lleva un solo par de cebadores “universales”. La amplificación simultánea de una mezcla de moldes de diferentes secuencias por PCR requeriría por otro lado una pluralidad de pares de cebadores, siendo cada par complementario a cada molde exclusivo en la mezcla. La generación de una pluralidad de pares cebadores para cada molde individual no es una acción viable para mezclas complejas de moldes.
La adición de secuencias de cebador universal sobre los extremos de los moldes a amplificar por PCR puede conseguirse mediante diversos métodos conocidos por los expertos en la técnica. Por ejemplo, un cebador universal que consiste en una secuencia universal en su extremo 5’ y una secuencia degenerada en su extremo 3’ puede usarse en una PCR (DOP-PCR, por ejemplo PNAS 1996 vol. 93 páginas 14676-14679) para amplificar
fragmentos al azar a partir de un molde complejo o una mezcla compleja de moldes. La parte 3’ degenerada del
cebador se hibrida en posiciones al azar sobre ADN y puede extenderse para generar una copia del molde que tiene la secuencia universal en su extremo 5’.
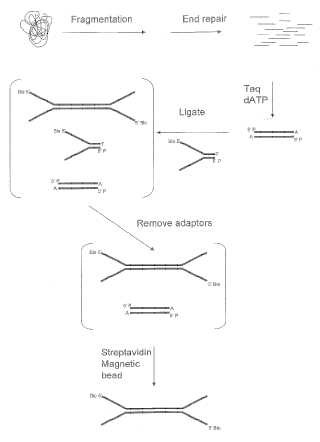
Como alternativa, pueden ligarse adaptadores que contengan secuencias de cebador universal en los extremos de los moldes. Los adaptadores pueden mono o bicatenarios. Sin son bicatenarios, estos pueden tener extremos solapantes que son complementarios a los extremos solapantes de las moléculas molde que se han generado con una endonucleasa de restricción. Como alternativa, los adaptadores bicatenarios pueden ser romos, en cuyo caso los moldes también tienen extremos romos. Los extremos romos de los moldes pueden haberse formado durante un proceso para cizallar el ADN en fragmentos, o pueden haberse formado mediante una reacción de reparación de extremos, como conocen los expertos en la técnica.
Pueden usarse un solo adaptador o dos adaptadores diferentes en una reacción de ligación con moldes. Si un molde se ha manipulado de tal manera que sus extremos son iguales, es decir, ambos son... [Seguir leyendo]
Reivindicaciones:
1. Un método para generar una biblioteca de moléculas polinucleotídicas molde que tienen secuencias comunes en sus extremos 5’ y secuencias comunes en sus extremos 3’, comprendiendo el método:
ligar polinucleótidos adaptadores con apareamiento erróneo idénticos en ambos extremos de cada uno de uno o más dúplex de polinucleótido diana para formar una o más construcciones adaptador-diana, en el que cada adaptador con apareamiento erróneo se forma a partir de dos cadenas polinucleotídicas hibridadas que forman un complejo biomolecular que comprende al menos una región bicatenaria y una región no apareada, y realizar una reacción de extensión con cebador inicial en la que un oligonucleótido cebador se hibrida con una parte de adaptador de cada una de las construcciones adaptador-diana y se extiende por adición secuencial de nucleótidos para formar productos de extensión complementarios a al menos una cadena de cada una de las construcciones adaptador-diana, en el que los productos de extensión, y opcionalmente productos de amplificación derivados de los mismos, proporcionan en su conjunto una biblioteca de moléculas polinucleotídicas molde que tienen secuencias comunes en sus extremos 5’ y secuencias comunes en sus extremos 3’; en el que los dúplex de polinucleótido diana que van a ligarse son una mezcla compleja de fragmentos de ADN genómico que representan un genoma completo o sustancialmente completo, y en el que la biblioteca de moléculas polinucleotídicas molde generadas es representativa del genoma completo o sustancialmente completo.
2. El método de acuerdo con la reivindicación 1, en el que el oligonucleótido cebador es un cebador con cola que comprende una secuencia con cola no hibridante en el extremo 5’.
3. El método de acuerdo con la reivindicación 1 o 2, en el que la reacción de extensión con cebador inicial comprende:
a) hibridar un primer oligonucleótido cebador con una parte de adaptador de cada una de las construcciones adaptador-diana, siendo el primer cebador un cebador con cola que comprende una cola 5’ no hibridante, b) extender el primer cebador por adición secuencial de nucleótidos para formar productos de extensión complementarios a al menos una cadena de cada una de la construcciones adaptador-diana, y c) someter los productos obtenidos en la etapa b) a condiciones desnaturalizantes, separando de este modo los productos de extensión de cadenas de las construcciones adaptador-diana.
4. El método de la reivindicación 3, que comprende la etapa adicional de hibridar segundos oligonucleótidos cebadores con los productos de extensión y extender los segundos cebadores para formar cadenas complementarias a los productos de extensión.
5. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 4, en el que el primer cebador utilizado en la reacción de extensión con cebador inicial se hibrida con una región no apareada de los adaptadores.
6. El método de acuerdo con la reivindicación 4, en el que la reacción de extensión con cebador inicial se realiza como parte de una reacción en cadena de la polimerasa y los productos de amplificación de la reacción PCR se recogen para proporcionar una biblioteca de moléculas polinucleotídicas molde que tienen secuencias comunes en
sus extremos 5’ y 3’.
7. Un método de acuerdo con la reivindicación 6, en el que la reacción en cadena de la polimerasa se realiza usando primeros cebadores de oligonucleótido que pueden hibridarse con una región no apareada de la parte de adaptador de una primera cadena de las construcciones adaptador-diana y segundos cebadores de oligonucleótido que pueden hibridarse con una región de las cadenas extendidas producidas por extensión de los primeros cebadores de oligonucleótido, siendo esta región complementaria a la región no apareada de la parte de adaptador en una segunda cadena de las construcciones adaptador-diana.
8. El método de acuerdo con la reivindicación 7, en el que los segundos cebadores también son cebadores con cola que comprenden una parte de cola en 5’ que no hibrida con los adaptadores con apareamiento erróneo.
9. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 8, en el que los polinucleótidos adaptadores con apareamiento erróneo son adaptadores bifurcados formados por hibridación de cadenas polinucleotídicas primera y segunda parcialmente complementarias, en el que una secuencia de 5 o más nucleótidos consecutivos en
el extremo 3’ de la primera cadena es complementaria a una secuencia de 5 o más nucleótidos consecutivos en el extremo 5’ de la segunda cadena de tal manera que una región bicatenaria de 5 o más pares de bases consecutivos se forma hibridando las dos cadenas y en el que una secuencia de al menos 10 nucleótidos consecutivos en el
extremo 5’ de la primera cadena y una secuencia de al menos 10 nucleótidos consecutivos en el extremo 3’ de la segunda cadena no son complementarias de tal manera que la región no apareada de al menos 10 nucleótidos consecutivos en cada cadena permanece como una forma monocatenaria cuando la región bicatenaria se hibrida.
10. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 8, en el que los dúplex de polinucleótido diana son fragmentos de un genoma completo.
11. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 8, en el que los dúplex de polinucleótido diana son ADNc bicatenarios.
12. Un método de amplificación de ácidos nucleicos en fase sólida de moléculas polinucleotídicas molde que comprende:
preparar una biblioteca de moléculas polinucleotídicas molde que tienen secuencias comunes en sus extremos 5’ y secuencias comunes en sus extremos 3’ usando el método de acuerdo con una cualquiera de las reivindicaciones 1 a 8 y realizar una reacción de amplificación de ácido nucleico en fase sólida en el que dichas moléculas polinucleotídicas molde se amplifican.
13. Uso de una biblioteca de moléculas polinucleotídicas molde preparadas de acuerdo con el método de una cualquiera de las reivindicaciones 1 a 11 como un molde para amplificación en fase sólida.
14. Uso de una biblioteca de moléculas polinucleotídicas molde preparadas de acuerdo con el método de la reivindicación 10 como un molde para amplificación del genoma completo.
15. Un kit para su uso en la preparación de una biblioteca de moléculas polinucleotídicas molde que tienen secuencias comunes en su extremo 5’ y secuencias comunes en sus extremos 3’, comprendiendo el kit polinucleótidos adaptadores bifurcados como se define en la reivindicación 9 y uno o más cebadores oligonucleotídicos que pueden hibridarse con los polinucleótidos adaptadores con apareamiento erróneo en el que
los cebadores oligonucleotídicos son cebadores con cola que comprenden una parte en cola 5’ que no hibrida con el
adaptador bifurcado.
16. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 12, o el uso de acuerdo con una cualquiera de las reivindicaciones 13 o 14, o el kit de acuerdo con la reivindicación 15, en el que al menos una de dichas secuencias comunes comprende la secuencia de la SEC ID Nº 1, o la secuencia de la SEC ID Nº 2.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]