Péptidos modificados que actúan como agentes terapéuticos.
Una composición de la Fórmula (X1)a-F1-(X2)b y sus multímeros,
donde: F1 es un dominio Fc; X1 y X2 se selecciona cada uno de manera independiente de -(L1)c-P1, -(L1)c-P1-(L2)d-P2, -(L1)c-P1-(L2)d-P2-(L3)e-P3, y -(L1)c-P1-(L2)d-P2-(L3)e-P3-(L4)f-P4 P1, P2, P3, y P4 son cada una secuencias aleatorizadas de manera independiente de péptidos farmacológicamente activos; L1, L2, L3 y L4 son cada uno de manera independiente enlazadores; y a, b, c, d, e, y f, son cada uno de manera independiente 0 o 1, con la condición de que al menos uno de a y b es 1, y donde "péptido" se refiere a moléculas de 2 a 40 aminoácidos y donde ni X1 ni X2 es una proteína nativa.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US1999/025044.
Solicitante: AMGEN INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: ONE AMGEN CENTER DRIVE THOUSAND OAKS, CA 91320-1799 ESTADOS UNIDOS DE AMERICA.
Inventor/es: BOONE, THOMAS, CHARLES, FEIGE, ULRICH, DR., LIU,CHUAN-FA, CHEETHAM,JANET.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07K19/00 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › Péptidos híbridos (Inmoglobulinas híbridas compuestas solamente de inmoglobulinas C07K 16/46).
- C12N1/21 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 1/00 Microorganismos, p.ej. protozoos; Composiciones que los contienen (preparaciones de uso médico que contienen material de protozoos, bacterias o virus A61K 35/66, de algas A61K 36/02, de hongos A61K 36/06; preparación de composiciones de uso médico que contienen antígenos o anticuerpos bacterianos, p. ej. vacunas bacterianas, A61K 39/00 ); Procesos de cultivo o conservación de microorganismos, o de composiciones que los contienen; Procesos de preparación o aislamiento de una composición que contiene un microorganismo; Sus medios de cultivo. › modificados por la introducción de material genético extraño.
- C12N15/62 C12N […] › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Secuencias de ADN que codifican proteínas de fusión.
- C12N15/70 C12N 15/00 […] › Vectores o sistemas de expresión especialmente adaptados a E. coli.
- C12R1/19 C12 […] › C12R SISTEMA DE INDEXACION ASOCIADO A LAS SUBCLASES C12C - C12Q, RELATIVO A LOS MICROORGANISMOS. › C12R 1/00 Microorganismos. › Escherichia coli.

Fragmento de la descripción:
Péptidos modificados como agentes terapéuticos.
Antecedentes de la invención
Las proteínas recombinantes son una clase emergente de agentes terapéuticos. Dichos productos terapéuticos recombinantes han dado lugar a avances en la formulación y modificación química de las proteínas. Dichas modificaciones pueden proteger las proteínas terapéuticas, básicamente bloqueando su exposición a enzimas proteolíticas. Las modificaciones de las proteínas también pueden aumentar la estabilidad, el tiempo de circulación y la actividad biológica de las proteínas terapéuticas. Un artículo de estudio que describe la modificación de las proteínas y las proteínas de fusión es Francis (1992), Focus on Growth Factors 3:4-10 (Mediscript, Londres), que se incorpora a la presente como referencia.
Una modificación útil es la combinación con el dominio "Fc" de un anticuerpo. Los anticuerpos comprenden dos partes funcionalmente independientes, un dominio variable conocido como "Fab", que se fija al antígeno, y un dominio constante conocido como "Fc", que se une a funciones efectoras tales como la activación del complemento y el ataque por parte de células fagocíticas. Un Fc tiene una larga vida media en suero, mientras que un Fab es de vida corta. Capon et al. (1989), Nature 337: 525-31. Cuando forma una estructura junto con una proteína terapéutica, un dominio Fc puede aportar una vida media más larga o incorporar funciones tales como la fijación al receptor de Fc, la fijación a la proteína A, la fijación del complemento y quizás incluso la transferencia placentaria. Id. La Tabla 1 resume el uso de las fusiones de Fc que se conocen en la técnica.
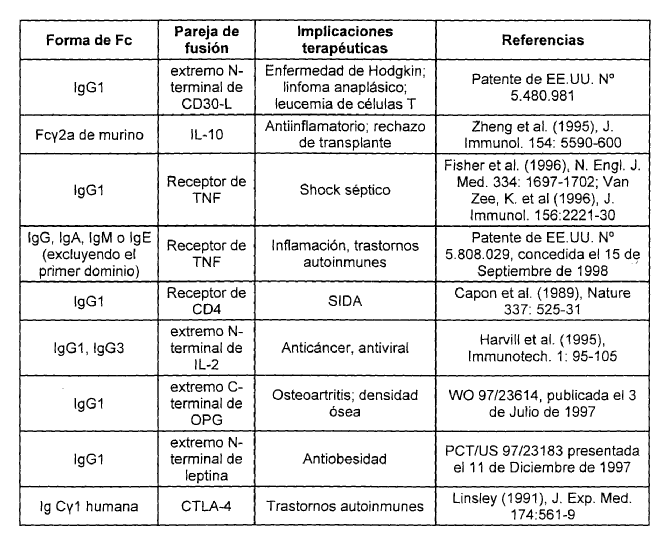
Un abordaje un tanto diferente al desarrollo de agentes terapéuticos es la selección de bibliotecas de péptidos. La interacción de un ligando de proteína con su receptor con frecuencia se produce en una interfase relativamente grande. Sin embargo, como se ha demostrado en el caso de la hormona del crecimiento humano y su receptor, solamente algunos de los restos clave en la interfase contribuyen a la mayor parte de la energía de fijación. Clackson et al. (1995), Science 267: 383-6. La mayor parte del ligando de proteína meramente exhibe los epítopos de fijación en la topología correcta o sirve a funciones no relacionadas con la vinculación. En consecuencia, las moléculas que poseen sólo longitud "peptídica" (2 a 40 aminoácidos) pueden vincularse a la proteína receptora de un determinado ligando de proteína grande. Dichos péptidos pueden imitar la bioactividad del ligando de proteína grande ("agonistas péptidos") o, mediante una vinculación competitiva, inhibir la bioactividad del ligando de proteína grande ("antagonistas péptidos").
Las bibliotecas de péptidos que exhiben fagos han surgido como un método poderoso para identificar tales agonistas y antagonistas peptídicos. Véase, por ejemplo, Scott et al. (1990), Science 249: 386; Devlin et al. (1990), Science 249: 404; Pat. de EE.UU. Nº 5.223.409, concedida el 29 de Junio de 1993; Pat. de EE.UU. Nº 5.733.731, concedida el 31 de Marzo de 1998; Pat. de EE.UU. Nº 5.498.530, concedida el 12 de Marzo de 1996; Pat. de EE.UU. Nº 5.432.018, concedida el 11 de Julio de 1995; Pat. de EE.UU. Nº 5.338.665, concedida el 16 de Agosto de 1994; Pat. de EE.UU. Nº 5.922.545, concedida el 13 de Julio de 1999; documentos WO 96/40987, publicado el 19 de Diciembre de 1996; y WO 98/15833, publicado el 16 de Abril de 1998 (cada una de las cuales se incorpora como referencia). En dichas bibliotecas, se exhiben secuencias de péptidos aleatorias mediante la fusión con proteínas de cubierta de fago filamentoso. Normalmente, los péptidos exhibidos son eluidos por afinidad contra un dominio extracelular de un receptor inmovilizado por anticuerpo. Los fagos retenidos pueden enriquecerse con rondas sucesivas de purificación y repropagación por afinidad. Se puede seguir la secuencia de los mejores péptidos de vinculación para identificar los restos clave dentro de una o más familias de péptidos relacionadas estructuralmente. Véase, p. ej., Cwirla et al. (1997), Science 276: 1696-9, donde se identificaron dos familias distintas. Las secuencias de péptidos también pueden sugerir qué restos pueden reemplazarse con seguridad mediante barrido con alanina o por mutagénesis en el nivel del ADN. Se pueden crear bibliotecas de mutagénesis y seleccionarse para optimizar más aún la secuencia de los mejores enlazadores. Lowman (1997), Ann. Rev. Biophys. Biomol. Struct. 26: 401-24.
El análisis estructural de la interacción proteína-proteína también puede usarse para sugerir péptidos que imitan la actividad de fijación de los ligandos de proteína grande. En un análisis de tal naturaleza, la estructura cristalina puede sugerir la identidad y la relativa orientación de los restos críticos del ligando de proteína grande, a partir de los cuales se puede diseñar un péptido. Véase, p. ej. Takasaki et al. (1997), Nature Biotech. 15: 1266-70. Estos métodos de análisis también pueden utilizarse para investigar la interacción entre una proteína receptora y péptidos seleccionados por exhibición de fagos, lo cual puede sugerir modificación ulterior de los péptidos para aumentar la afinidad de fijación.
Hay otros métodos que compiten con la exhibición de fagos en la investigación de péptidos. Una biblioteca de péptidos puede fusionarse al extremo terminal carboxilo del represor de Iac y expresarse en E. coli. Otro método basado en E. coli permite la exhibición en la membrana externa celular por fusión con una lipoproteína asociada a peptidoglicano (PAL). De aquí en más en la presente, estos métodos y otros relacionados se denominarán colectivamente "exhibición en E. coli". En otro método, la traducción de ARN aleatorio se detiene antes de la liberación del ribosoma, lo que da como resultado una biblioteca de polipéptidos con su ARN asociado todavía ligado. De aquí en más en la presente, este método y otros relacionados se denominarán colectivamente "exhibición ribosómica". Otros métodos emplean la unión química de péptidos al ARN; véase, por ejemplo, Roberts & Szostak (1997), Proc. Natl. Acad. Sci. USA, 94: 12297-303. De aquí en más en la presente, este método y otros relacionados se denominarán colectivamente "selección de ARN-péptidos". Se han desarrollado bibliotecas de péptidos derivados químicamente en las que los péptidos son inmovilizados sobre materiales estables, no biológicos, tales como varillas de polietileno o resinas permeables a solventes. Otra biblioteca de péptidos derivados químicamente utiliza la fotolitografía para escanear péptidos inmovilizados sobre portaobjetos de vidrio. De aquí en más en la presente, este método y otros relacionados se denominarán colectivamente "selección de péptidos químicos". La selección de péptidos químicos puede resultar ventajosa porque permite usar D-aminoácidos y otros análogos no naturales, así como elementos no peptídicos. Tanto los métodos biológicos como los químicos están analizados en Wells & Lowman (1992), Curr. Opin. Biotechnol. 3: 355-62.
En concepto, se pueden descubrir miméticos peptídicos de cualquier proteína utilizando exhibición de fagos y los otros métodos mencionados más arriba. Estos métodos han sido utilizados para el mapeo de epítopos, para la identificación de aminoácidos críticos en las interacciones proteína-proteína, y como bases para el descubrimiento de nuevos agentes terapéuticos. P. ej., Cortese et al. (1996), Curr. Opin. Biotech. 7: 616-21. En la actualidad las bibliotecas de péptidos se están usando con mucha más frecuencia en estudios inmunológicos, tales como el mapeo de epítopos. Kreeger (1996), The Scientist 10(13): 19-20.
En la presente es de especial interés el uso de las bibliotecas de péptidos y de otras técnicas en el descubrimiento de péptidos farmacológicamente activos. Una serie de tales péptidos identificados en la técnica están resumidos en la Tabla 2. Los péptidos aparecen descritos en las publicaciones enumeradas, cada una de las cuales se incorpora a la presente como referencia. Se describe la actividad farmacológica de los péptidos, y en muchos casos aparece a continuación un término abreviado para el mismo entre paréntesis. Algunos de estos péptidos han sido modificados...
Reivindicaciones:
1. Una composición de la Fórmula
y sus multímeros, donde:
F1 es un dominio Fc;
X1 y X2 se selecciona cada uno de manera independiente de -(L1)c-P1, -(L1)c-P1-(L2)d-P2, -(L1)c-P1-(L2)d-P2-(L3)e-P3, y -(L1)c-P1-(L2)d-P2-(L3)e-P3-(L4)f-P4
P1, P2, P3, y P4 son cada una secuencias aleatorizadas de manera independiente de péptidos farmacológicamente activos;
L1, L2, L3 y L4 son cada uno de manera independiente enlazadores; y
a, b, c, d, e, y f, son cada uno de manera independiente 0 o 1, con la condición de que al menos uno de a y b es 1, y donde "péptido" se refiere a moléculas de 2 a 40 aminoácidos y donde ni X1 ni X2 es una proteína nativa.
2. La composición de acuerdo con la reivindicación 1 de las Fórmulas
o
3. La composición de acuerdo con la reivindicación 1 de la Fórmula
4. La composición de acuerdo con la reivindicación 1 de la Fórmula
5. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 4, donde F1 es un dominio Fc de IgG.
6. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 4, donde F1 es un dominio Fc de IgG 1.
7. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 4, donde F1 comprende la secuencia de SEQ ID NO: 2.
8. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 7, donde P1, P2, P3, y P4 son cada uno secuencias peptídicas antagonistas de IL-1 aleatorizadas de manera independiente.
9. La composición de acuerdo con la reivindicación 8, donde la secuencia peptídica antagonista de IL-1 se selecciona de las SEQ ID NOs: 212, 907, 908, 909, 910, 917 y 979.
10. La composición de acuerdo con la reivindicación 8, donde la secuencia peptídica antagonista de IL-1 se selecciona de las SEQ ID NOs: 213 a 271, 671 a 906, 911 a 916 y 918 a 1023.
11. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 7, donde P1, P2, P3, y P4 son cada uno secuencias peptídicas miméticas de EPO aleatorizadas de manera independiente.
12. La composición de acuerdo con la reivindicación 11, donde la secuencia peptídica mimética de EPO se selecciona de la Tabla 5.
13. La composición de acuerdo con la reivindicación 11, que comprende una secuencia seleccionada de las SEQ ID NOs: 83, 84, 85, 124, 419, 420, 421 y 461.
14. La composición de acuerdo con la reivindicación 11, que comprende una secuencia seleccionada de las SEQ ID NOs: 339 y 340.
15. La composición de acuerdo con la reivindicación 11, que comprende una secuencia seleccionada de las SEQ ID NOs: 20 y 22.
16. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 7, donde P1, P2, P3, y P4 son cada uno secuencias peptídicas miméticas de TPO aleatorizadas de manera independiente.
17. La composición de acuerdo con la reivindicación 16, donde la secuencia peptídica mimética de TPO se selecciona de la Tabla 6.
18. La composición de acuerdo con la reivindicación 16, que comprende una secuencia seleccionada de las SEQ ID NOs: 6 y 12.
19. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 7, donde P1, P2, P3 y P4 son cada uno secuencias peptídicas antagonistas de TNF aleatorizadas de manera independiente.
20. La composición de acuerdo con cualquiera de las reivindicaciones 1 a 7, donde P1, P2, P3 y P4 son cada uno secuencias peptídicas miméticas de GCSF aleatorizadas de manera independiente.
21. Un ADN que codifica una composición de acuerdo con cualquiera de las reivindicaciones 1 a 20.
22. Un vector de expresión que comprende el ADN de la reivindicación 21.
23. Una célula huésped que comprende el vector de expresión de la reivindicación 22.
24. La célula de la reivindicación 23, donde la célula es una célula de E. coli.
25. Una composición farmacéutica que comprende una composición de acuerdo con cualquiera de las reivindicaciones 1 a 20 junto con un diluyente, conservante, solubilizante, emulsionante, adyuvante y/o portador, aceptable para uso farmacéutico.
26. Una composición de acuerdo con cualquiera de las reivindicaciones 1 a 20 para usar como agente terapéutico o profiláctico.
27. Un proceso para preparar un compuesto farmacológicamente activo, que consiste en
donde "péptido" se refiere a moléculas de 2 a 40 aminoácidos.
28. El proceso de la reivindicación 27, donde el péptido se selecciona en un proceso que comprende seleccionar de una biblioteca de exhibición de fagos, una biblioteca de exhibición de E. coli, una biblioteca ribosomal, o una biblioteca de péptidos químicos.
29. El proceso de la reivindicación 27 o 28, donde el péptido es un péptido mimético de EPO aleatorizado.
30. El proceso de la reivindicación 27 o 28, donde el péptido es un péptido mimético de TPO aleatorizado.
31. El proceso de la reivindicación 27 o 28, donde el péptido es un péptido antagonista de IL-1 aleatorizado.
32. El proceso de la reivindicación 27 o 28, donde el péptido es un péptido mimético de GCSF aleatorizado.
33. El proceso de la reivindicación 27 o 28, donde el péptido es un péptido antagonista de TNF aleatorizado.
34. El proceso de la reivindicación 27 o 28, donde el péptido se selecciona de las Tablas 4 a 20.
35. El proceso de cualquiera de las reivindicaciones 27 a 34, donde el compuesto preparado es de la Fórmula
y sus multímeros, donde:
F1 es un dominio Fc;
X1 y X2 se selecciona cada uno de manera independiente de -(L1)c-P1,-(L1)c-P-(L1)d-P2, -(L1)c-P1-(L2)d-P2-(L3)e-P3, y -(L1)c-P1-(L2)d-P2-(L3)e-P3-(L4)f-P4
P1, P2, P3, y P4 son cada una secuencias aleatorizadas de manera independiente de péptidos farmacológicamente activos;
L1, L2, L3 y L4 son cada uno de manera independiente enlazadores; y
a, b, c, d, e, y f, son cada uno de manera independiente 0 o 1, con la condición de que al menos uno de a y b es 1, y donde ni X1 ni X2 es una proteína nativa.
36. El proceso de la reivindicación 35, donde el compuesto preparado es de las Fórmulas
o
37. El proceso de la reivindicación 35, donde el compuesto preparado es de las Fórmulas
o
38. El proceso de acuerdo con cualquiera de las reivindicaciones 27 a 37, donde F1 es un dominio Fc de IgG.
39. El proceso de acuerdo con cualquiera de las reivindicaciones 27 a 37, donde F1 es un dominio Fc de IgG 1.
40. El proceso de acuerdo con cualquiera de las reivindicaciones 27 a 37, donde F1 comprende la secuencia de SEQ ID NO: 2.
Patentes similares o relacionadas:
Cadena ligera de enteroquinasa modificada, del 22 de Julio de 2020, de NOVO NORDISK A/S: Un análogo de la cadena ligera de la enteroquinasa bovina que comprende una secuencia de aminoácidos establecida en la SEQ ID NO: 1, en donde dicho análogo comprende […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Antígenos de coagulasa estafilocócica y métodos para su uso, del 13 de Mayo de 2020, de UNIVERSITY OF CHICAGO: Una composición inmunógena que comprende al menos dos dominios 1-2 de coagulasa estafilocócica diferentes, en donde cada uno de los al menos dos dominios […]
Reconocimiento de unión a diana celular mediante un agente bioactivo usando transferencia de energía de resonancia de bioluminiscencia intracelular, del 6 de Mayo de 2020, de PROMEGA CORPORATION: Un sistema de ensayo que comprende: (a) una biblioteca de agentes bioactivos, cada uno de los cuales está fijado a un fluoróforo; (b) una diana celular fusionada a […]
Utilización diagnóstica de un polipéptido de fusión que comprende una proteína vírica y un enzima MGMT, del 15 de Abril de 2020, de INSTITUT PASTEUR: Utilización in vitro de un polipéptido de fusión que comprende una proteína vírica y i) el enzima 6-metilguanina-ADN-metiltransferasa (MGMT, EC 2.1.1.63) o un homólogo […]
Etiqueta de epítopo y método de detección, captura y/o purificación de polipéptidos etiquetados, del 15 de Abril de 2020, de ChromoTek GmbH: Péptido epítopo aislado que tiene de 12 a 25 aminoácidos, en donde la secuencia de aminoácidos comprende una secuencia según se define en SEQ ID NO: 32 (X1X2RX4X5AX7SX9WX11X12), […]