PROCEDIMIENTO PARA LA CARACTERIZACIÓN DE POLINUCLEÓTIDOS.
Procedimiento para determinar la secuencia de un polinucleótido diana,
que comprende las etapas siguientes: (i)tratar una muestra de un polinucleótido diana bicatenario para crear regiones protuberantes en cada extremo, una de las cuales debe secuenciarse, presentando cada región protuberante un número definido de bases, (ii)dividir la muestra en compartimientos de reacción y poner en contacto cada muestra separada con una secuencia polinucleótida de señal bicatenaria diferente y un polinucleótido adaptador bicatenario correspondiente, representando cada secuencia de señal una secuencia polinucleótida específica de la misma longitud que la de la región protuberante que debe secuenciarse, y que comprende una región protuberante que permite la hibridación y la ligación con el extremo del polinucleótido diana situado enfrente del que se está secuenciando, y comprendiendo cada adaptador una región protuberante que presenta una secuencia complementaria a la secuencia representada por la secuencia de señal, hibridándose el adaptador con la región protuberante en la muestra diana que debe secuenciarse únicamente en el caso de que la región protuberante del adaptador sea complementaria a la región protuberante que se está secuenciando, (iii)llevar a cabo la reacción de polimerasa de la muestra o muestras utilizando cebadores que se hibridan con los extremos de la secuencia de señal y de la secuencia adaptadora, en la que el producto de la reacción de polimerasa comprende un sitio de restricción que permite el corte del adaptador para formar una nueva región protuberante que debe secuenciarse, opcionalmente mediante la repetición de las etapas (i) a (iii) utilizando enzimas de restricción para crear las regiones protuberantes, e (iv)identificar qué secuencias de señal se encuentran presentes en los productos amplificados, y en qué orden, para determinar de este modo la secuencia del polinucleótido diana
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2004/001673.
Solicitante: LINGVITAE AS.
Nacionalidad solicitante: Noruega.
Dirección: TRIMVEIEN 6, A562 0372 OSLO NORUEGA.
Inventor/es: LEXOW, PREBEN, RAGNHILDSTVEIT,ERLEND.
Fecha de Publicación: .
Fecha Solicitud PCT: 16 de Abril de 2004.
Fecha Concesión Europea: 10 de Febrero de 2010.
Clasificación Internacional de Patentes:
- C12Q1/68D2E1
- C12Q1/68E4
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
Fragmento de la descripción:
Procedimiento para la caracterización de polinucleótidos.
Campo de la invención
La presente invención se refiere a procedimientos para convertir un polinucleótido diana en cadenas de información legible, para la determinación posterior de la secuencia del polinucleótido diana.
Antecedentes de la invención
Los avances en el estudio de las moléculas han sido impulsados, en parte, por la mejora de las tecnologías utilizadas para caracterizar las moléculas o las reacciones biológicas de las mismas. En particular, el estudio de los ácidos nucleicos ADN y ARN se ha beneficiado de las tecnologías en desarrollo utilizadas para el análisis de secuencias y del estudio de los sucesos de hibridación.
El procedimiento principal utilizando en general para la secuenciación de ADN a gran escala es el procedimiento de terminación de cadena. Este procedimiento fue desarrollado por primera vez por Sanger y Coulson (Sanger et al., Proc. Natl. Acad. Sci. USA 74:5463-5467, 1977) y se basa en la utilización de derivados dideoxi de los cuatro nucleótidos que se incorporan en la cadena polinucleótida naciente en una reacción de polimerasa. Tras la incorporación, los derivados dideoxi terminan la reacción de polimerasa y seguidamente los productos se separan mediante electroforesis en gel y se analizan para revelar la posición en la que se ha incorporado en la cadena el derivado dideoxi particular.
Aunque dicho procedimiento ha sido utilizado ampliamente y produce resultados fiables, se reconoce que resulta lento, laborioso y caro.
El documento US-A-5.302.509 da a conocer un procedimiento para secuenciar un polinucleótido inmovilizado sobre un soporte sólido. El procedimiento se basa en la incorporación de bases A, G, C y T 3-bloqueadas que presentan un marcaje fluorescente diferente al del polinucleótido inmovilizado, en presencia de ADN polimerasa. La polimerasa incorpora una base complementaria al polinucleótido diana, pero el grupo 3'-bloqueante impide adiciones posteriores. El marcaje de la base incorporada seguidamente puede determinarse y eliminarse el grupo bloqueante mediante corte químico, permitiendo que se produzca la polimerización posterior. Sin embargo, la necesidad de eliminar los grupos bloqueantes de esta manera requiere mucho tiempo y debe realizarse con elevada eficiencia.
El documento WO-A-00/39333 describe un procedimiento para secuenciar un polinucleótido mediante la conversión de la secuencia de un polinucleótido diana en un segundo polinucleótido que presenta una secuencia definida e información posicional contenidas en el mismo. La información de secuencia de la diana se dice que se encuentra "magnificada" en el segundo polinucleótido, permitiendo una mayor facilidad para distinguir entre las bases individuales de la molécula diana. Esto se consigue utilizando "etiquetas magnificadoras", que son secuencias de ácidos nucleicos predeterminadas. Cada una de las bases adenina, citosina, guanina y timina en la molécula diana se encuentra representada por una etiqueta magnificadora individual, convirtiendo la secuencia diana original en una secuencia magnificada. A continuación, las técnicas convencionales pueden utilizarse para determinar el orden de las etiquetas magnificadoras, determinando de esta manera la secuencia específica del polinucleótido diana.
En un procedimiento de secuenciación preferente, cada etiqueta magnificadora comprende un marcaje, por ejemplo un marcaje fluorescente, que seguidamente puede identificarse y utilizarse para caracterizar la etiqueta magnificadora.
Aunque el procedimiento dado a conocer en la presente publicación de patente presenta muchas ventajas, todavía existe una necesidad de procedimientos mejoradas para secuenciar los polinucleótidos diana.
Sumario de la invención
Según un primer aspecto de la presente invención, un procedimiento para identificar características específicas de un polinucleótido diana presente en una muestra comprende las etapas siguientes:
El procedimiento de la presente invención permite determinar múltiples características de un polinucleótido diana de una manera simple que puede automatizarse fácilmente. En la forma de realización preferida, el procedimiento se basa en la incorporación de dos moléculas polinucleótidas en aquellas dianas que presentan la característica específica, permitiendo llevar a cabo una reacción de amplificación, incrementado selectivamente de esta manera el número de polinucleótidos diana con la característica deseada y eliminando efectivamente aquellas dianas que no presentan dicha característica. El procedimiento resulta particularmente adecuado para determinar la secuencia de un polinucleótido diana.
Según un segundo aspecto de la invención, un procedimiento para determinar la secuencia de un polinucleótido diana comprende las etapas siguientes:
Descripción de los dibujos
La invención se describe haciendo referencia a las figuras adjuntas, en las que:
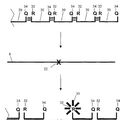
la figura 1 es una representación esquemática de una secuencia de señal, que ilustra la conversión de parte de un polinucleótido diana en una secuencia de señal definida,
la figura 2 es una representación esquemática de un experimento de secuenciación de un polinucleótido que se ha llevado a cabo utilizando la presente invención,
la figura 3 es una representación esquemática de la utilización de diferentes compartimientos con secuencias de señal específicas asociadas en un procedimiento de conversión,
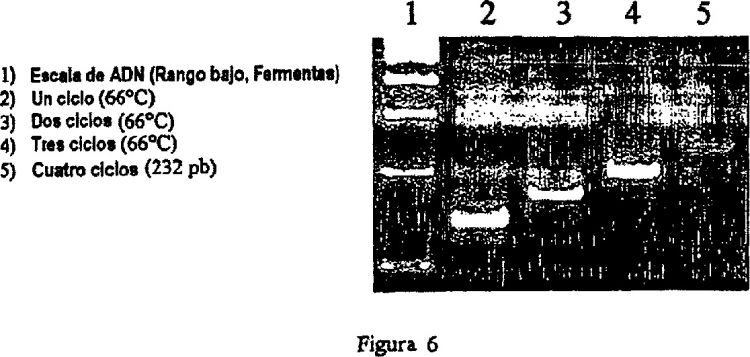
la figura 4 muestra los resultados de la conversión, llevando a cabo tres ciclos para formar una serie de secuencias de señal que representan la secuencia de bases de un polinucleótido diana,
la figura 5 es una representación esquemática de un experimento de secuenciación de un polinucleótido, que se ha llevado a...
Reivindicaciones:
1. Procedimiento para determinar la secuencia de un polinucleótido diana, que comprende las etapas siguientes:
2. Procedimiento según la reivindicación 1, en el que la región protuberante que se liga a la secuencia de señal presenta por lo menos 3 bases.
3. Procedimiento según la reivindicación 1 ó 2, en el que la región protuberante que debe secuenciarse presenta 4 bases.
4. Procedimiento según cualquiera de las reivindicaciones anteriores, en el que la combinación de la totalidad de las secuencias representadas por la secuencia de señal en cada compartimiento de reacción corresponde a la totalidad de las permutaciones de una secuencia que comprende el número de bases en la región protuberante que debe secuenciarse.
5. Procedimiento según cualquiera de las reivindicaciones anteriores, en el que el sitio de restricción es específico para una enzima de clase II.
6. Procedimiento según la reivindicación 5, en el que la enzima es SfaNI o EarI.
7. Procedimiento según cualquiera de las reivindicaciones anteriores, en el que la reacción de polimerasa se lleva a cabo utilizando metil-dCTP como sustituto de dCTP.
8. Procedimiento según cualquiera de las reivindicaciones anteriores, en el que el adaptador se inmoviliza sobre un material de soporte.
9. Procedimiento según cualquiera de las reivindicaciones anteriores, en el que secuencias de señal secuenciales, y opcionalmente los adaptadores secuenciales, comprenden sitios de reconocimiento para diferentes oligonucleótidos cebadores.
Patentes similares o relacionadas:
 MÉTODO PARA LA DETECCIÓN DE UNA ALTERACIÓN EN UN ÁCIDO NUCLÉICO, del 18 de Febrero de 2011, de GENZYME CORPORATION: Un método para detectar una alteración en un ácido nucleico diana que se sospecha que está en una muestra biológica, comprendiendo el método las etapas […]
MÉTODO PARA LA DETECCIÓN DE UNA ALTERACIÓN EN UN ÁCIDO NUCLÉICO, del 18 de Febrero de 2011, de GENZYME CORPORATION: Un método para detectar una alteración en un ácido nucleico diana que se sospecha que está en una muestra biológica, comprendiendo el método las etapas […]
AMPLIFICACION DE ACIDOS NUCLEICOS MEDIANTE EMULSION DE PERLAS, del 11 de Mayo de 2010, de 454 CORPORATION: Método para la amplificación de uno o más ácidos nucleicos sobre una perla, que comprende las etapas siguientes: (a) formar una emulsión […]
MÉTODO DE ANÁLISIS DE ÁCIDO NUCLEICO PARA ANALIZAR EL PATRÓN DE METILACIÓN DE ISLAS CPG EN DIFERENTES MUESTRAS, del 14 de Febrero de 2012, de ORYZON GENOMICS, S.A.: Método para determinar la metilación de un ácido nucleico que comprende las siguientes etapas: a) fragmentación de una muestra de ADN genómico con al menos un enzima de […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]