Métodos y dispositivos para la síntesis de ácidos nucleicos.
Un método para producir al menos un polinucleótido que tiene una secuencia predefinida,
comprendiendo el método:
a. proporcionar al menos una primera y una segunda pluralidad de oligonucleótidos monocatenarios unidos al soporte, en donde cada primera y segunda pluralidades de oligonucleótidos tiene una secuencia predefinida y se unen a una característica discreta de un soporte, cada primera pluralidad de oligonucleótidos unidos al soporte comprende una región de secuencia en su extremo 5' complementario a una región de secuencia de un extremo 5' de la segunda pluralidad de oligonucleótidos unidos al soporte;
b. generar al menos una primera y una segunda pluralidades de oligonucleótidos de construcción complementarios a la primera y la segunda pluralidades de oligonucleótidos unidos al soporte en una reacción de extensión de la cadena, cada primera pluralidad de oligonucleótidos de construcción comprende una región de secuencia en su extremo 3' que es complementaria a una región de secuencia de un extremo 3' de la segunda pluralidad de oligonucleótidos de construcción;
c. proporcionar una pluralidad de oligonucleótidos monocatenarios de anclaje unidos al soporte en una característica seleccionada, en donde el extremo 5' de la pluralidad de un primer oligonucleótido de anclaje es el mismo que una región de secuencia de la primera pluralidad de oligonucleótidos unidos al soporte;
d. hibridar al menos la primera y la segunda pluralidades de oligonucleótidos de construcción a la pluralidad de oligonucleótidos de anclaje; y
e. ligar al menos la primera y la segunda pluralidades de oligonucleótidos de construcción, generando de ese modo al menos un polinucleótido.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2011/060243.
Solicitante: Gen9, Inc.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 840 MEMORIAL DRIVE CAMBRIDGE, MA 02139 ESTADOS UNIDOS DE AMERICA.
Inventor/es: SCHINDLER,DANIEL, JACOBSON,JOSEPH, KUNG,LI-YUN A, WILSON,ANDREW KIRK, RAMU,SENTHIL, HUDSON,MIKE.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/66 C12N 15/00 […] › Métodos generales para insertar un gen en un vector para formar un vector recombinante, utilizando la escisión y la unión; Utilización de "linkers" no funcionales o de adaptadores, p. ej. "linkers" que contienen la secuencia para una endonucleasa de restricción.
- C12P19/34 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
PDF original: ES-2548400_T3.pdf

Fragmento de la descripción:
Métodos y dispositivos para la síntesis de ácidos nucleicos
Campo de la invención
Los métodos y aparatos proporcionados en el presente documento se refieren a la síntesis y ensamblaje de ácidos nucleicos y bibliotecas de ácido nucleico que tienen una secuencia predefinida. Más particularmente, se proporcionan métodos y aparatos para la síntesis de polinucleótidos diana en un soporte sólido y para la selección de los polinucleótidos diana.
Antecedentes
El empleo de las técnicas de química de ADN recombinante, es hoy en día común para las secuencias de ADN que se van a replicar y amplificar a partir de la naturaleza y después desensamblar en partes del componente. Según las partes del componente, las secuencias se recombinan o se vuelven a ensamblar después en nuevas secuencias de ADN. Sin embargo, la dependencia en las secuencias disponibles de forma natural limita significativamente las posibilidades que pueden estudiar los investigadores. Mientras que en la actualidad es posible para las secuencias cortas de ADN sintetizarse directamente a partir de nucleósidos individuales, ha sido poco factible en general construir directamente grandes segmentos o ensamblajes de polinucleótidos, es decir, secuencias polinucleótidas más largas de aproximadamente 400 pares de bases.
Se puede realizar la síntesis de oligonucleótidos mediante síntesis personalizadas masivamente paralelas en microchips (Zhou et al. (2004) Nucleic Acids Res. 32:5409; Fodor et al. (1991) Science 251:767) . Sin embargo, los microchips actuales tienen áreas de superficie muy bajas y por tanto sólo pueden producirse pequeñas cantidades de oligonucleótidos. Cuando se liberan en la solución, los oligonucleótidos se presentan en concentraciones picomolares o inferiores por secuencia, concentraciones que no son suficientemente elevadas para impulsar las reacciones de cebado bimoleculares de manera eficiente. Los métodos actuales para ensamblar ácidos nucleicos requieren que se amplifiquen los oligonucleótidos de microchips antes del ensamblaje. Como tal, sigue habiendo una necesidad de mejora de métodos y dispositivos rentables para el ensamblaje de genes de alta fidelidad y la producción de un gran número de secuencias polinucleótidas.
El documento WO2004/024886 informa sobre la síntesis in vitro y el ensamblaje de polinucleótidos de longitud génica, largos basado en un ensamblaje de múltiples oligonucleótidos más cortos sintetizados in situ en una 35 plataforma de micromatrices. El documento WO2005/059096 informa sobre la producción de moléculas de ácido nucleico largas con un control del usuario preciso en el contenido de la secuencia. El documento WO2010/025310 informa sobre métodos para sintetizar y/o ensamblar al menos un producto polinucleótido que tiene una secuencia predefinida a partir de una pluralidad de oligonucleótidos diferentes. El documento DE114343591 informa sobre la producción de elementos funcionales oligoméricos o poliméricos (EF) mediante el enlace de al menos dos elementos de diseño (ED) . El documento WO01/88173 informa sobre métodos de preparación de un polinucleótido que tiene al menos 200 nucleótidos en una dirección 5' a 3' o 3' a 5' ligando una pluralidad de oligonucleótidos, cuyo ensamblaje, representa la secuencia nucleótida del polinucleótido deseado. El documento WO99/47536 informa sobre un método recursivo para sintetizar moléculas de ácido nucleico que implica ciclos de unión de moléculas de ácido nucleico a una molécula de ácido nucleico preparada. El documento EP2175021 informa sobre la síntesis de 45 hebras dobles de ácido nucleico de una secuencia opcional en un soporte que tiene un área de superficie que contiene una pluralidad de áreas de reacción individual. El documento WO00/29616 informa sobre métodos de ensamblaje de un polinucleótido en un soporte sólido realizando etapas de hibridación, ligamiento, y extensión. El documento WO00/75368 informa sobre métodos para la síntesis en fragmentos de ADN en una matriz sólida. Los documentos EP1314783 y EP1411122 informan sobre una molécula de ácido nucleico monocatenario para su uso en la preparación de ácido nucleico, que comprende una secuencia que corresponde al sitio de reconocimiento de una enzima de restricción tipo IIS, y una secuencia arbitraria pero definida de nucleótidos. El documento WO2006/076679 informa sobre la producción de proteínas con características deseadas mediante la expresión y exploración de ácidos nucleicos codificantes. El documento WO2006/044956 informa sobre métodos de fabricación de ADN sintético mediante el ensamblaje multiplexado de oligonucleótidos sintéticos. El documento WO99/14318 55 informa sobre la síntesis química completa y el ensamblaje de genes y genomas. El documento WO2007/010252 y el documento WO2008/041002 informan sobre la secuenciación por pares de una plantilla polinucleótida bicatenaria.
Sumario
Los aspectos de la invención se refieren a métodos y aparatos para preparar y/o ensamblar polímeros de alta fidelidad. En el presente documento se proporciona también dispositivos y métodos para procesar reacciones de ensamblaje de ácido nucleico y ácidos nucleicos ensamblados. Es objeto de la presente invención proporcionar métodos económicos, prácticos para sintetizar ácidos nucleicos personalizados.
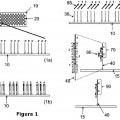
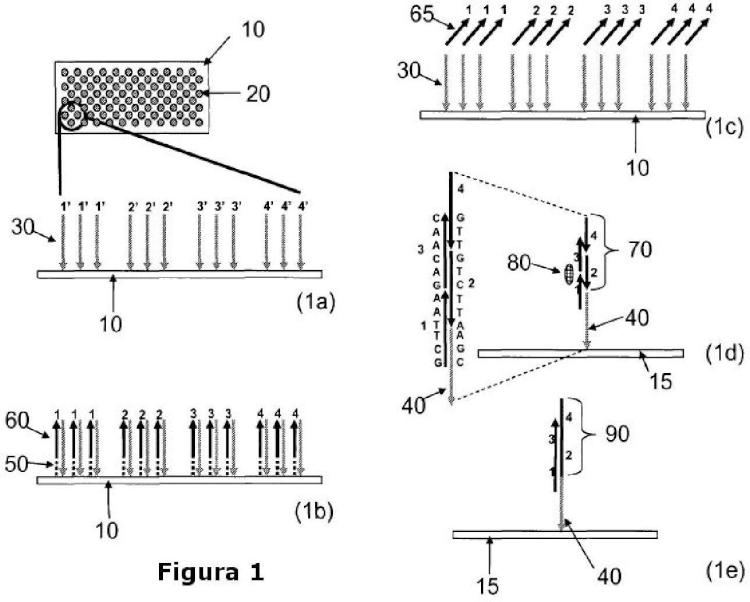
Los aspectos de la invención se refieren a métodos y dispositivos para producir un polinucleótido que tiene una secuencia predeterminada en un soporte sólido. En algunas realizaciones, se proporcionan pluralidades de
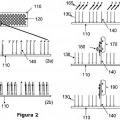
oligonucleótidos monocatenarios unidos al soporte en características diferentes de un soporte sólido, cada pluralidad de oligonucleótidos tiene una secuencia predefinida, siendo cada pluralidad unida a una característica discreta diferente del soporte. En algunas realizaciones, cada pluralidad de oligonucleótidos comprende una región de secuencia en su extremo 3' que es complementaria a una región de secuencia de un extremo 3' de otro oligonucleótido y en la que la primera pluralidad de oligonucleótidos tiene un extremo 5' que es complementario a un extremo 5' de un primer oligonucleótido monocatenario de anclaje. En algunas realizaciones, la pluralidad de oligonucleótidos unidos al soporte se inmoviliza en el soporte. En algunas realizaciones la pluralidad de oligonucleótidos unidos al soporte se sintetiza en el soporte sólido. En otras realizaciones, la pluralidad de oligonucleótidos unidos al soporte se deposita en el soporte sólido. En algunas realizaciones, el soporte es un dispositivo de micromatrices.
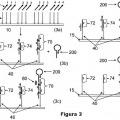
Según algunas realizaciones, se proporcionan al menos una primera y una segunda pluralidad de oligonucleótidos monocatenarios unidos al soporte, en la que cada primera y segunda pluralidad de oligonucleótidos tienen una secuencia predefinida y se unen a una característica discreta del soporte. En algunas realizaciones, cada primera 15 pluralidad de oligonucleótidos comprende una región de secuencia en su extremo 3' que es complementaria a una región de secuencia de un extremo 3' de la segunda pluralidad de oligonucleótidos. En algunas realizaciones, se proporciona una pluralidad de oligonucleótidos monocatenarios de anclaje unidos al soporte, en la que el extremo 5' de la pluralidad del primer oligonucleótido de anclaje es igual que una región de secuencia de la primera pluralidad de oligonucleótidos unidos al soporte. Al menos se generan una primera y una segunda pluralidad de oligonucleótidos de construcción complementarias a la primera y segunda pluralidad de oligonucleótidos unidos al soporte en una reacción de extensión de la cadena. Los oligonucleótidos de construcción pueden hibridarse en la pluralidad de oligonucleótidos de anclaje en una característica seleccionada. Al menos se ligan la primera y la segunda pluralidad de oligonucleótidos de construcción, generando de este modo al menos un polinucleótido que tiene una secuencia predefinida. En algunas realizaciones, al menos se disocian la primera y segunda pluralidad de 25 oligonucleótidos de construcción a partir de al menos la primera y segunda pluralidad de oligonucleótidos unidos al soporte. En algunas realizaciones, la primera pluralidad de oligonucleótidos de construcción se transfiere de una primera característica a una característica seleccionada y la segunda pluralidad de oligonucleótidos de construcción se transfiere de una segunda característica a la característica seleccionada, en la que la característica seleccionada comprende una pluralidad de oligonucleótidos monocatenarios de anclaje unidos al soporte. En algunas realizaciones, la característica seleccionada se encuentra en el mismo soporte que la primera y la segunda característica. En otras realizaciones, la característica... [Seguir leyendo]
Reivindicaciones:
1. Un método para producir al menos un polinucleótido que tiene una secuencia predefinida, comprendiendo el método: 5
a. proporcionar al menos una primera y una segunda pluralidad de oligonucleótidos monocatenarios unidos al soporte, en donde cada primera y segunda pluralidades de oligonucleótidos tiene una secuencia predefinida y se unen a una característica discreta de un soporte, cada primera pluralidad de oligonucleótidos unidos al soporte comprende una región de secuencia en su extremo 5' complementario a una región de secuencia de un extremo 5' de la segunda pluralidad de oligonucleótidos unidos al soporte;
b. generar al menos una primera y una segunda pluralidades de oligonucleótidos de construcción complementarios a la primera y la segunda pluralidades de oligonucleótidos unidos al soporte en una reacción de extensión de la cadena, cada primera pluralidad de oligonucleótidos de construcción comprende una región de secuencia en su extremo 3' que es complementaria a una región de secuencia de un extremo 3' de la segunda
pluralidad de oligonucleótidos de construcción;
c. proporcionar una pluralidad de oligonucleótidos monocatenarios de anclaje unidos al soporte en una característica seleccionada, en donde el extremo 5' de la pluralidad de un primer oligonucleótido de anclaje es el mismo que una región de secuencia de la primera pluralidad de oligonucleótidos unidos al soporte;
d. hibridar al menos la primera y la segunda pluralidades de oligonucleótidos de construcción a la pluralidad de oligonucleótidos de anclaje; y
e. ligar al menos la primera y la segunda pluralidades de oligonucleótidos de construcción, generando de ese modo al menos un polinucleótido.
2. El método de la reivindicación 1, en el que al menos la primera y la segunda pluralidades de oligonucleótidos de construcción se disocian de al menos la primera y la segunda pluralidades de oligonucleótidos unidos al soporte y comprende además, transferir la primera pluralidad de oligonucleótidos de construcción a partir de una primera característica a una característica seleccionada y transferir la segunda pluralidad de oligonucleótidos de construcción de una segunda característica a la característica seleccionada, en donde la característica seleccionada comprende la pluralidad de oligonucleótidos monocatenarios de anclaje unidos al soporte, en donde la característica seleccionada está en el mismo soporte que la primera y la segunda características o en donde la característica seleccionada está en un soporte diferente de la primera y la segunda características.
3. El método según una cualquiera de las reivindicaciones anteriores, que comprende adicionalmente:
proporcionar una tercera pluralidad de oligonucleótidos monocatenarios unidos al soporte, en donde cada tercera pluralidad de oligonucleótidos unidos al soporte tiene una secuencia predefinida y se une a una tercera característica discreta del soporte, cada tercera pluralidad de oligonucleótidos unidos al soporte comprende una región de secuencia en su extremo 3' que es complementaria a una región de secuencia de un extremo 3' de la segunda pluralidad de oligonucleótidos unidos al soporte; generar una tercera pluralidad de oligonucleótidos de construcción complementarios a la tercera pluralidad de oligonucleótidos unidos al soporte en una reacción de extensión de cadena; hibridar al menos la primera, segunda y tercera pluralidades de oligonucleótidos de construcción a la pluralidad de oligonucleótidos de anclaje en la característica seleccionada; y 45 ligar al menos la primera, segunda y tercera pluralidades de oligonucleótidos de construcción, produciendo de ese modo al menos un polinucleótido.
4. El método según una cualquiera de las reivindicaciones anteriores, en el que cada pluralidad de oligonucleótidos unidos al soporte tiene un sitio de unión al cebador en su extremo 3' y comprende además la hibridación de un cebador a al menos las primera y segunda pluralidades de oligonucleótidos unidos al soporte en condiciones que favorecen la extensión del cebador, formando de este modo duplexos del producto de extensión.
5. El método según una cualquiera de las reivindicaciones anteriores, en el que la pluralidad de oligonucleótidos unidos al soporte se sintetiza en el soporte sólido, en el que la pluralidad de oligonucleótidos unidos al soporte se
deposita en el soporte sólido o en el que la pluralidad de oligonucleótidos unidos al soporte se inmoviliza en el soporte a través de su extremo 3'.
6. El método según una cualquiera de las reivindicaciones anteriores, en el que el soporte es un dispositivo de micromatrices.
7. El método según cualquiera de las reivindicaciones anteriores, que comprende:
proporcionar pluralidades N de oligonucleótidos monocatenarios unidos al soporte generando de este modo una primera pluralidad de oligonucleótidos de construcción que comprende en su extremo 3' una región de secuencia 65 que es complementaria a una región de secuencia en el extremo 3' de un segundo oligonucleótido de construcción, y una pluralidad de oligonucleótidos de construcción N que comprende en su extremo 3' una región
de secuencia que es complementaria a una región de secuencia de una pluralidad de oligonucleótidos de construcción (N-1) ; y proporcionar una pluralidad de oligonucleótidos de anclaje que comprende en su extremo 5' una secuencia que es la misma que una región de secuencia de la primera pluralidad de oligonucleótidos unidos al soporte.
8. El método de la reivindicación 7, en el que se generan las pluralidades N de oligonucleótidos de construcción complementarios a los oligonucleótidos monocatenarios unidos al soporte y en el que las pluralidades N de los oligonucleótidos de construcción abarcan la secuencia completa del polinucleótido sin espacios.
9. El método según una cualquiera de las reivindicaciones anteriores, en el que la región de secuencia del extremo 3' de la primera pluralidad de oligonucleótidos unidos al soporte es idéntica a la región del extremo 5' de los oligonucleótidos de anclaje.
10. El método de la reivindicación 4, que disocia además los duplexos de extensión liberando de este modo al 15 menos una primera y una segunda pluralidad de oligonucleótidos de construcción.
11. El método de la reivindicación 4, en el que la secuencia del cebador comprende al menos un uracilo y comprende además eliminar el cebador utilizando una mezcla de uracilo-ADN glicosilasa (UAG) y ADN glicosilasaliasa endonucleasa VIII.
12. El método de la reivindicación 1, que comprende además
(i) proporcionar un soporte con una primera pluralidad de oligonucleótidos de anclaje unidos al soporte, en donde el extremo 5' de la primera pluralidad de oligonucleótidos de anclaje es complementario al extremo 5' de una primera pluralidad de oligonucleótidos y una segunda pluralidad de oligonucleótidos de anclaje unidos al soporte, en donde el extremo 5' del segundo oligonucleótido de anclaje es complementario a un oligonucleótido de construcción N terminal;
(ii) sintetizar una pluralidad de polinucleótidos bicatenarios unidos al soporte que comprende un saliente monocatenario 5', la pluralidad de secuencias polinucleótidas comprende la secuencia polinucleótida predefinida, en donde el saliente monocatenario 5' de la secuencia polinucleótida predefinida comprende la secuencia de oligonucleótido de construcción N terminal y el extremo 3' monocatenario de la secuencia polinucleótida que comprende la primera secuencia oligonucleótida;
(iii) hibridar la pluralidad de polinucleótidos sintetizados con los primeros oligonucleótidos de anclaje; y (iv) someter los polinucleótidos sintetizados a condiciones de hibridación para hibridar el saliente 5' del polinucleótido que tiene un oligonucleótido de construcción N terminal en el extremo 5' de la segunda pluralidad de oligonucleótidos de anclaje, seleccionando de este modo los polinucleótidos que tienen la secuencia predefinida.
13. El método de la reivindicación 12, que comprende uno o más de los siguientes:
degradar las secuencias polinucleótidas que tienen un extremo 3' o 5' libre; liberar los polinucleótidos que tienen la secuencia predefinida desde el soporte, en donde se liberan los polinucleótidos utilizando una endonucleasa tipo II o en donde los polinucleótidos se liberan utilizando una mezcla de uracilo-ADN glicosilasa (UAG) y ADN glicosilasa-ligasa endonucleasa VIII.
14. El método de la reivindicación 12, en el que la primera pluralidad de oligonucleótidos de anclaje está separada de la segunda pluralidad de oligonucleótidos de anclaje por una distancia correspondiente a la longitud del polinucleótido predefinido.
15. El método de la reivindicación 14, en el que el soporte comprende además oligonucleótidos monocatenarios separados unidos al soporte para fijar la distancia entre el primer y el segundo oligonucleótidos de anclaje, y en donde la distancia entre el primer y el segundo oligonucleótidos de anclaje depende de una concentración del primer y del segundo oligonucleótidos de anclaje y de la concentración del oligonucleótido separador.
16. El método de la reivindicación 12, en el que la pluralidad de polinucleótidos bicatenarios unidos al soporte se sintetiza mediante extensión de cadena de la polimerasa utilizando como plantillas una pluralidad de oligonucleótidos monocatenarios unidos al soporte.
17. Una matriz de ácido nucleico, que comprende:
a. un soporte sólido;
b. una pluralidad de características discretas asociadas al soporte sólido, en donde cada característica comprende una pluralidad de oligonucleótidos unidos al soporte que tiene una secuencia predefinida, en donde la primera pluralidad de oligonucleótidos unidos al soporte comprende en su extremo 5' una región de secuencia
que es complementaria a una región de secuencia en el extremo 5' de una segunda pluralidad de oligonucleótidos unidos al soporte, en donde una pluralidad de oligonucleótidos unidos al soporte N comprende
en su extremo 5' una secuencia complementaria a la región de secuencia del extremo 5' de una pluralidad de oligonucleótidos unidos al soporte (N-1) ; y c. al menos una primera pluralidad de oligonucleótidos de anclaje que comprende en su extremo 5' una secuencia que es idéntica a una región de secuencia de la primera pluralidad de oligonucleótidos unidos al 5 soporte.
18. La matriz de ácidos nucleicos de la reivindicación 17, que comprende además una segunda pluralidad de oligonucleótidos de anclaje unidos al soporte, en donde el extremo 5' del segundo oligonucleótido de anclaje es idéntico al extremo 3' de la pluralidad de oligonucleótidos unidos al soporte N.
19. Un método para producir una pluralidad de polinucleótidos que tiene una secuencia predefinida, comprendiendo el método:
(a) proporcionar un primer y un segundo soportes que tienen una pluralidad de características, en donde cada característica comprende una pluralidad de oligonucleótidos unidos al soporte diferentes que tienen una secuencia predefinida diferente;
(b) generar una primera y una segunda pluralidades de oligonucleótidos de construcción diferentes que tienen secuencias predefinidas diferentes utilizando como plantillas la pluralidad de oligonucleótidos unidos al soporte, la primera y la segunda pluralidades de oligonucleótidos de construcción tienen secuencias complementarias de extremo 3;
(c) proporcionar un soporte que comprende una pluralidad de características, en donde cada característica comprende oligonucleótidos monocatenarios de anclaje unidos al soporte, en donde el extremo 5' de cada pluralidad de oligonucleótidos de anclaje es complementario al extremo 5' de la primera pluralidad de oligonucleótidos de construcción;
(d) hibridar la primera pluralidad de oligonucleótidos de construcción con los oligonucleótidos de anclaje;
(e) hibridar la segunda pluralidad de oligonucleótidos de construcción con la primera pluralidad de oligonucleótidos de construcción;
(f) hibridar opcionalmente de forma secuencial una tercera pluralidad de oligonucleótidos de construcción con la segunda pluralidad de oligonucleótidos de construcción; y
(g) ligar las pluralidades de oligonucleótidos de construcción.
20. El método de la reivindicación 19, en el que se aplican uno o más de los siguientes:
cada pluralidad de oligonucleótidos de construcción que define cada polinucleótido predefinido se sintetiza en un soporte diferente; la pluralidad de diferentes polinucleótidos se ensambla en diferentes características de soporte que comprenden los oligonucleótidos de anclaje unidos al soporte; la etapa de generar la pluralidad de los primeros oligonucleótidos de construcción comprende hibridar una secuencia del cebador que tiene al menos un uracilo con la primera pluralidad de oligonucleótidos unidos al soporte en condiciones que favorecen la extensión del cebador y eliminar el cebador utilizando una mezcla de uracilo-ADN glicosilasa (UAG) y ADN glicosilasa-ligasa endonucleasa VIII.
21. El método de la reivindicación 19, que comprende:
(i) proporcionar un primer soporte que comprende una pluralidad de características, en el que cada característica comprende oligonucleótidos monocatenarios de anclaje unidos al soporte, en el que el extremo 5' de cada pluralidad de oligonucleótidos de anclaje es complementario al extremo 5' de una primera pluralidad de oligonucleótidos de construcción;
(ii) proporcionar un segundo soporte que tiene una pluralidad de características, en el que cada característica comprende una pluralidad de oligonucleótidos monocatenarios de plantilla unidos al soporte, cada pluralidad de oligonucleótidos unidos al soporte tiene una secuencia predefinida diferente; generar una primera pluralidad de oligonucleótidos de construcción por extensión de la polimerasa, utilizando como plantillas la pluralidad de oligonucleótidos unidos al soporte;
(iii) colocar el primer y el segundo soportes, de modo que cada característica del segundo soporte se alinee a 55 una característica correspondiente del primer soporte;
(iv) liberar la primera pluralidad de oligonucleótidos de construcción en una solución en condiciones que favorecen la hibridación de la primera pluralidad de oligonucleótidos de construcción con la pluralidad de oligonucleótidos de anclaje correspondientes; y
(v) repetir opcionalmente las etapas (ii) - (iv) con un tercer soporte que comprende una segunda pluralidad de oligonucleótidos de construcción, la segunda y la tercera pluralidad de oligonucleótidos de construcción tienen secuencias complementarias del extremo 3'.
22. El método de la reivindicación 21, en el que se aplican uno o más de los siguientes:
la etapa de generar la pluralidad de los primeros oligonucleótidos de construcción comprende hibridar una secuencia del cebador que tiene al menos un uracilo con la primera pluralidad de oligonucleótidos unidos al
soporte en condiciones que favorecen la extensión del cebador y eliminar el cebador utilizando una mezcla de uracilo-ADN glicosilasa (UAG) y ADN glicosilasa-ligasa endonucleasa VIII; en donde cada característica del segundo soporte comprende una pluralidad de oligonucleótidos, en donde la pluralidad de oligonucleótidos comprende al menos dos poblaciones de oligonucleótidos que tienen diferentes secuencias predefinidas, al menos dos poblaciones de oligonucleótidos tienen secuencias complementarias del extremo 3'; en donde el segundo soporte se coloca arriba y orientado al primer soporte; en donde la etapa de liberación de la primera pluralidad de oligonucleótidos de construcción en solución permite la difusión de la primera pluralidad de oligonucleótidos hacia los oligonucleótidos de anclaje;
en donde la estequiometría de la primera pluralidad de oligonucleótidos de construcción es superior a la estequiometría de los oligonucleótidos de anclaje; y/o en donde el tercer soporte comprende una pluralidad de polinucleótidos inmovilizados por hibridación con una pluralidad de oligonucleótidos de anclaje.
23. El método según cualquiera de las reivindicacione.
2. 22, que comprende además ligar los oligonucleótidos de anclaje y la segunda pluralidad de oligonucleótidos de construcción.
24. El método de la reivindicación 21, en el que la etapa de liberar la primera pluralidad de oligonucleótidos de construcción en solución se realiza en presencia de una membrana porosa que permite una difusión vertical sustancial de los oligonucleótidos de construcción hacia los oligonucleótidos de anclaje, en donde la membrana porosa reduce la difusión lateral de los oligonucleótidos de construcción.
25. El método de la reivindicación 21, en el que la etapa de liberar las dos poblaciones de oligonucleótidos en solución permite la hibridación de la primera población de oligonucleótidos de construcción con la segunda población de oligonucleótidos de construcción y la hibridación de la primera población de oligonucleótidos con el oligonucleótido de anclaje.
26. El método de la reivindicación 25, en el que la solución comprende una ligasa.
27. El método según cualquiera de las reivindicacione.
2. 26, en el que la pluralidad de polinucleótidos se inmoviliza en el apoyo o está en solución.
28. El método según una cualquiera de las reivindicacione.
2. 23, en el que se comprende además la exposición de la pluralidad de polinucleótidos a un componente que reconoce y escinde un mal apareamiento en condiciones 35 adecuadas para la escisión de los polinucleótidos bicatenarios que contienen un nucleótido mal apareado.
29. El método de la reivindicación 28, en el que el componente que reconoce y escinde el mal apareamiento comprende una endonucleasa de mal apareamiento y/o la endonucleasa específica de mal apareamiento es una enzima CEL I.
30. El método de la reivindicación 17, que comprende además una tercera pluralidad de oligonucleótidos unidos al soporte que tiene una secuencia predefinida, en donde la tercera pluralidad de oligonucleótidos unidos al soporte comprende en su extremo 3' una región de secuencia que es complementaria a una región de secuencia en el extremo 3' de una segunda pluralidad de oligonucleótidos unidos al soporte.
Patentes similares o relacionadas:
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Método para romper un ácido nucleico y añadir un adaptador por medio de transposasa y reactivo, del 1 de Julio de 2020, de MGI Tech Co., Ltd: Un metodo para romper un acido nucleico y anadir un adaptador por medio de una transposasa, que comprende las siguientes etapas: interrumpir […]
Vacuna subunitaria contra Mycoplasma spp., del 1 de Julio de 2020, de Agricultural Technology Research Institute: Una composición para prevenir una infección por Mycoplasma spp., que comprende: un principio activo, que comprende una proteína de PdhA; y un adyuvante […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Proteínas de captura de la superficie celular recombinantes, del 13 de Mayo de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para detectar y aislar células que producen altos niveles de una proteína heterodimérica que tiene una primera subunidad y una segunda subunidad, […]
Etiquetado y evaluación de una secuencia diana, del 13 de Mayo de 2020, de RhoDx, Inc: Un método para modificar un ácido nucleico, que comprende: (a) poner en contacto un ácido nucleico de cadena sencilla con una actividad de transferasa […]
Preparación de bibliotecas de ácido nucleico marcado usando protocolo de adición en un solo tubo, del 13 de Mayo de 2020, de ILLUMINA, INC: Un método para preparar una biblioteca de fragmentos de ácido nucleico marcados que comprende: (a) poner en contacto una célula individual […]