PROMOTORES AOX1 MUTANTES.
Un promotor de alcohol oxidasa 1 (AOX1) de Pichia pastoris mutante del promotor de AOX1 de Pichia pastoris de tipo salvaje (SEQ ID No.
1) que comprende al menos una mutación en los nucleótidos 694 a 723 (-260 a -231) del Seq ID No. 1 para su expresión elevada en condiciones de des-represión en relación con el promotor de AOX1 de tipo salvaje
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E10155180.
Solicitante: TECHNISCHE UNIVERSITÄT GRAZ
VTU HOLDING GMBH.
Nacionalidad solicitante: Austria.
Dirección: RECHBAUERSTRASSE, 12 8010 GRAZ AUSTRIA.
Inventor/es: GLIEDER, ANTON, HARTNER,Franz.
Fecha de Publicación: .
Fecha Solicitud PCT: 23 de Febrero de 2006.
Clasificación Internacional de Patentes:
- C12N15/81A
Clasificación PCT:
- C12N15/00 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K).
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
PDF original: ES-2358086_T3.pdf
Fragmento de la descripción:
Promotores AOX1 mutantes.
La presente invención se refiere a promotores de AOX1 de Pichia pastoris mutantes.
S. cerevisiae ha dominado (y todavía domina) el uso científico y biotecnológico como modelo de organismo y sistema de producción eucariótico. En el último siglo, ha aumentado sumamente el atractivo de otra levadura: la levadura de fisión Schizosaccharomyces pombe. Por su atributo para reproducirse solamente por medio de fisión S. pombe adquirió una atención excepcional como organismo modelo y hoy en día es la especie de levadura más intensamente estudiada en términos de genética molecular y biología celular, junto con S. cerevisiae. Entre las más de 700 especies de levadura diferentes conocidas hasta la fecha, las dos levaduras mencionadas antes pueden proporcionar solamente un conjunto limitado de atributos interesantes para aplicaciones tecnológicas y científicas. Desde los años 70 u 80 se han investigado más y más especies de levadura con características excepcionales para la biotecnología y la investigación. Estas levaduras denominadas no convencionales (NCY) o levaduras no Saccharomyces (en este caso el término Saccharomyces incluye la levadura Schizosaccharomyces pombe) se desarrollan por varias razones: poseen importancia médica como Candida albicans o relevancia tecnológica como Yarrowia lipolytica y Kluyveromyces lactis que tienen la capacidad de crecer sobre sustratos concretos (p. ej. n-alcanos, lactosa). Por ejemplo el patógeno fúngico humano más común C. albicans se estudia extensamente para revelar la naturaleza de los factores de virulencia implicados en la patogénesis, convirtiéndose por lo tanto en el organismo modelo para las levaduras patogénicas. Otro grupo bien establecido de NCY son las levaduras metilotróficas Pichia pastoris y Hansenula polymorpha (Pichia angusta) que son superiores a S. cerevisiae en términos de producción de proteína recombinante y estudios de biogénesis de peroxisomas. Estos son solamente los miembros más prominentes de las levaduras no convencionales que tienen todavía atractivo tecnológico académico. Hasta la fecha otras numerosas especies también tienen un interés particular y este grupo aumentará rápidamente en los próximos años.
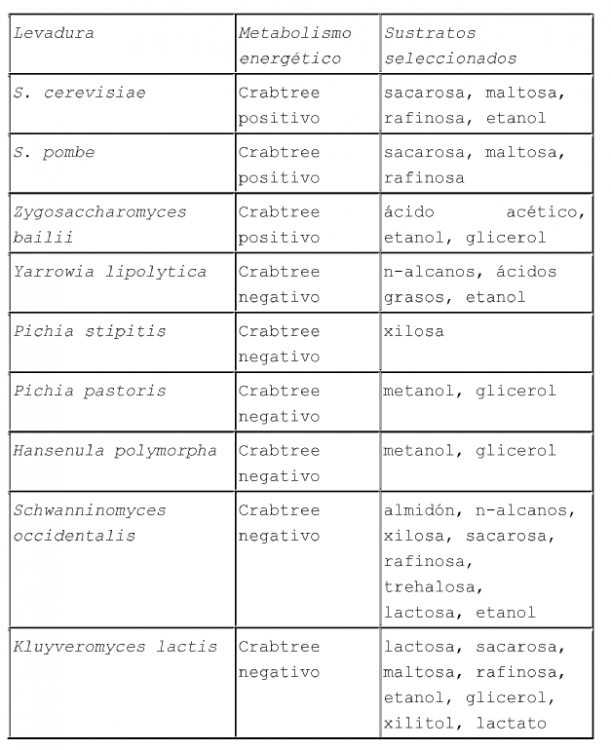
Los azúcares, la clase de moléculas más abundante en la naturaleza, son utilizados por todas las levaduras conocidas. Aunque hay grandes diferencias en la aceptación del sustrato entre especie y especie (véase la Tabla 1), la conversión de glucosa 6-fosfato o fructosa 6-fosfato en piruvato es un tema común en su metabolismo. De cualquier manera, el equipamiento enzimático para la ruta glicolítica varía significativamente entre las diferentes levaduras. Si bien en S. cerevisiae la mayoría de las enzimas son conocidas y están caracterizadas, al menos parcialmente, en las NCY solamente se describieron unas pocas enzimas. Algunas de las funciones necesarias para la glicólisis están mediadas por numerosos genes/enzimas en algunas levaduras, especialmente aquellas que juegan un papel adicional en el control o la regulación del metabolismo y/o que están en un punto de ramificación como glucoquinasa/hexoquinasa, fosfofructoquinasa y gliceraldehído 3-fosfato deshidrogenasa. Normalmente, las isoenzimas se regulan diferencialmente indicando diversas funciones bajo pre-requisitos medioambientales cambiantes. Algunos de los genes que codifican enzimas glicolíticas son constitutivos y altamente expresados, p. ej. el gen PGK1 (fosfoglicerato quinasa) de S. cerevisiae o GAP (gliceraldehído 3-fosfato deshidrogenasa) de P. pastoris mientras otras enzimas son estrictamente reguladas como el gen de la ENO1 (enolasa) de S. cerevisiae.
(Tabla pasa a página siguiente)
El destino del piruvato en el metabolismo varía significativamente entre las especies de levadura y las condiciones de cultivo. En S. cerevisiae y otras levaduras denominadas Crabtree positivas, la respiración es inhibida por la glucosa y azúcares afines. Esto conduce a la transformación de piruvato vía piruvato descarboxilasa en etanol y CO2, incluso con grandes cantidades de oxígeno, lo que también es conocido como fermentación. En las levaduras Crabtree negativas, a la que pertenecen la mayoría de las NCY, la transformación de piruvato en etanol se produce solamente en condiciones anaerobias. En condiciones aerobias el piruvato se oxida a CO2 vía piruvato deshidrogenasa y ciclo de los ácidos tricarboxílicos (TCA). El ciclo de los TCA tiene un interés excepcional para el metabolismo celular debido al hecho de que esta es la única vía de oxidación de los azúcares a CO2. La oxidación a CO2 da como resultado la producción de NADH, que se utiliza para la producción de energía. Además, los intermedios del ciclo de los TCA son las principales fuentes de metabolitos con fines biosintéticos. Debido a la eliminación de intermedios el ciclo de los TCA debe ser recargado para mantenerse en funcionamiento. Las principales reacciones anapleróticas en levaduras son la piruvato carboxilasa y el ciclo del glioxilato. La primera es la ruta principal cuando se desarrolla sobre amonio como única fuente de nitrógeno mientras el último es necesario cuando se desarrolla sobre fuentes de carbono con menos de 3 átomos de carbono. En contraste con este eminente interés no se conoce apenas nada acerca de los genes o enzimas implicados en el ciclo de los TCA en las NCY. El NADH generado por las reacciones catabólicas, ya sea en el citosol o en las mitocondrias, tiene que ser re-oxidado a NAD+ para mantener las reacciones en funcionamiento. En las levaduras Crabtree negativas (p. ej. Pichia pastoris) en condiciones aerobias el NADH es re-oxidado principalmente a través de la cadena respiratoria. La situación es significativamente diferente en las levaduras Crabtree positivas como S. cerevisiae donde coexisten respiración y fermentación. Cuando se desarrolla sobre glucosa en condiciones aerobias, la respiración es reprimida por la glucosa y se produce la fermentación. En estas condiciones el NAD+ es regenerado por la formación de etanol (NADH producido por glicólisis) o glicerol. La respiración en levaduras difiere del paradigma animal de esta ruta descrito en cualquier libro de texto de bioquímica. Primero, algunas levaduras, como S. cerevisiae y Kluyveromyces lactis, carecen del complejo I de la cadena respiratoria. En estas levaduras la regeneración de NAD+ se realiza sin bombear protones a través de la membrana mitocondrial interna por las NADH deshidrogenasas externa e interna. La segunda diferencia principal, encontrada en las levaduras Crabtree negativas, hongos y plantas, es una ruta de respiración alternativa paralela al complejo III y IV de la cadena del citocromo. Esta respiración alternativa está mediada por la denominada oxidasa alternativa que transfiere electrones directamente desde la reserva de ubiquinona a oxígeno sin bombear protones a través de la membrana mitocondrial interna.
El NADPH para fines biosintéticos se produce en la porción oxidativa de la ruta de las pentosas fosfato (PPP). Otros metabolitos muy importantes proporcionados por esta ruta son la ribosa 5-fosfato y la eritrosa 4-fosfato, necesarias para la síntesis de ácidos nucleicos y cofactores de nucleótidos y para la síntesis de aminoácidos aromáticos, respectivamente. Todavía existen muchos vacíos en la información sobre los genes y sus correspondientes enzimas implicados en la PPP en las levaduras no convencionales. Se aislaron unas pocas enzimas de Candida utilis, S. pombe y K. lactis. La caracterización composicional y cinética reveló varias diferencias entre estas enzimas. Debido a la carencia de información, no se puede estimar la influencia de la PPP en estas levaduras pero se ha demostrado que p. ej. los mutantes para la fosfoglucosa isomerasa de K. lactis, que carecen de glicólisis, son capaces de crecer en medio con glucosa, en contraste con S. cerevisiae. Esta observación indica que la capacidad de la ruta de las pentosas fosfato en K. lactis es suficiente para el crecimiento sobre glucosa como fuente de carbono. En levaduras metilotróficas, se pudo encontrar una transcetolasa adicional... [Seguir leyendo]
Reivindicaciones:
1. Un promotor de alcohol oxidasa 1 (AOX1) de Pichia pastoris mutante del promotor de AOX1 de Pichia pastoris de tipo salvaje (SEQ ID No. 1) que comprende al menos una mutación en los nucleótidos 694 a 723 (-260 a -231) del Seq ID No. 1 para su expresión elevada en condiciones de des-represión en relación con el promotor de AOX1 de tipo salvaje.
2. El promotor de acuerdo con la reivindicación 1, caracterizado porque el promotor comprende adicionalmente una mutación de los nucleótidos 170 a 239 (-784 a -719) y/o los nucleótidos 729 a 763 (-225 a -191) del Seq ID No. 1 y/o de un sitio de unión al factor de transcripción (TFBS) y/o de al menos una mutación seleccionada del grupo que consiste en los nucleótidos 170 a 191 (-784 a -763), los nucleótidos 192 a 213 (-762 a -741), nucleótidos 192 a 210 (-762 a -744), los nucleótidos 207 a 209 (-747 a -745), los nucleótidos 214 a 235 (-740 a -719), los nucleótidos 304 a 350 (-650 a -604), los nucleótidos 364 a 393 (-590 a -561), los nucleótidos 434 a 508 (-520 a -446), los nucleótidos 509 a 551 (-445 a -403), los nucleótidos 552 a 560 (-402 a -394), los nucleótidos 585 a 617 (-369 a -337), los nucleótidos 621 a 660 (-333 a -294), los nucleótidos 625 a 683 (-329 a -271), los nucleótidos 736 a 741 (-218 a -213), los nucleótidos 737 a 738 (-217 a -216), los nucleótidos 726 a 755 (-228 a -199), los nucleótidos 784 a 800 (-170 a -154) o los nucleótidos 823 a 861 (-131 a -93).
3. El promotor de acuerdo con la reivindicación 1 o 2, caracterizado porque la mutación es una deleción, una sustitución, una inserción y/o una inversión.
4. El promotor de acuerdo con la reivindicación 2 o 3, caracterizado porque el sitio de unión al factor de transcripción (TFBS) se selecciona del grupo que consiste en Hap1, Hsf, Hap234, abaA, Stre, Rap1, Adr1, Mat1MC, Gcr1 y QA-1F, donde el sitio de unión al factor de transcripción (TFBS) Hap1 comprende preferiblemente los nucleótidos 54 a 58 del Seq ID No. 1, Hsf los nucleótidos 142 a 149 y 517 a 524 del Seq ID No. 1, Hap234 los nucleótidos 196 a 200, 206 a 210 y 668 a 672 del Seq ID No. 1, abaA los nucleótidos 219 a 224 del Seq ID No. 1, Stre los nucleótidos 281 a 285 del Seq ID No. 1, Rap1 los nucleótidos 335 a 339 del Seq ID No. 1, Adr1 los nucleótidos 371 a 377 del Seq ID No.1, Mat1MC los nucleótidos 683 a 687 del Seq ID No. 1, Gcr1 los nucleótidos 702 a 706 del Seq ID No. 1 y QA-1F los nucleótidos 747 a 761 del Seq ID No. 1.
5. Una molécula de ácido nucleico que comprende al menos un promotor de alcohol oxidasa 1 (AOX1) de Pichia pastoris mutante de acuerdo con una cualquiera de las reivindicaciones 1 a 4 y al menos un ácido nucleico que codifica una proteína (péptido) o un ácido nucleico funcional, caracterizado porque dicho promotor y dicho ácido nucleico están conectados operablemente entre sí formando una casete de expresión con una única copia o múltiples copias.
6. Un vector que comprende un promotor de alcohol oxidasa 1 (AOX1) de Pichia pastoris mutante de acuerdo con una cualquiera de las reivindicaciones 1 a 4 o una molécula de ácido nucleico de acuerdo con la reivindicación 5.
7. Una célula que comprende al menos un promotor de la alcohol oxidasa 1 (AOX1) de Pichia pastoris mutante de acuerdo con una cualquiera de las reivindicaciones 1 a 4, al menos un fragmento de ácido nucleico de acuerdo con la reivindicación 5 o al menos un vector de acuerdo con la reivindicación 6.
8. Una célula de acuerdo con reivindicación 7, caracterizada porque dicha célula es una célula eucariótica, en particular una célula de levadura, preferiblemente una célula de levadura metilotrófica, seleccionada preferiblemente del grupo que consiste en Candida, Hansenula, Pichia y Torulopsis, en particular una célula de Pichia pastoris.
9. Un kit para la expresión de una proteína seleccionada que comprende
10. Un kit de acuerdo con la reivindicación 9, caracterizado porque dicha célula es una célula de levadura, preferiblemente una célula de levadura metilotrófica, seleccionada preferiblemente del grupo que consiste en Candida, Hansenula, Pichia y Torulopsis, en particular una célula de Pichia pastoris.
11. Un método para la expresión de una proteína, un péptido o un ácido nucleico funcional recombinantes en una célula que comprende las siguientes etapas:
12. El método de acuerdo con la reivindicación 11, caracterizado porque dicha célula es una célula de levadura, preferiblemente una célula de levadura metilotrófica, seleccionada preferiblemente del grupo que consiste en Candida, Hansenula, Pichia y Torulopsis, en particular una célula de Pichia pastoris.
13. El uso de una molécula de ácido nucleico de acuerdo con la reivindicación 5, un vector de acuerdo con la reivindicación 6 o una célula de acuerdo con una cualquiera de las reivindicaciones 7 u 8 para la expresión de una proteína, péptido o ácido nucleico funcional.
14. Un método para el aislamiento de clones de superexpresión que comprende las etapas de:
15. Un método de acuerdo con la reivindicación 14, caracterizado porque el marcador selectivo es un antibiótico, preferiblemente Zeocina o geneticina.
16. Un método de acuerdo con la reivindicación 14 o 15, caracterizado porque el marcador selectivo es Zeocina y el de resistencia marcador es el gen sh ble.
17. Un método de acuerdo con una cualquiera de las reivindicaciones 14 a 16, caracterizado porque la célula es una célula de levadura, preferiblemente una célula de levadura metilotrófica, preferiblemente seleccionada del grupo que consiste en Candida, Hansenula, Pichia y Torulopsis, en particular una célula de Pichia pastoris.
18. Un método de acuerdo con una cualquiera de las reivindicaciones 11 a 17, caracterizado porque la fuente de carbono no represora se selecciona del grupo que consiste en alanina, manitol, sorbitol, trehalosa, lactosa y sus combinaciones.
19. Un método de acuerdo con una cualquiera de las reivindicaciones 11 a 18, caracterizado porque la molécula de ácido nucleico o el vector son introducidos en la célula mediante transformación, preferiblemente electroporación o transformación química, o mediante fusión de protoplastos, o mediante bombardeo de partículas.
Patentes similares o relacionadas:
 USO DE LA QUIMOSINA RECOMBINANTE ACTIVA, del 11 de Mayo de 2011, de UNIVERSIDAD DE SANTIAGO DE COMPOSTELA: Uso de la quimosina recombinante activa.Uso del cuajo, sin activación previa, obtenido a partir de una célula hospedadora que contiene un vector de expresión […]
USO DE LA QUIMOSINA RECOMBINANTE ACTIVA, del 11 de Mayo de 2011, de UNIVERSIDAD DE SANTIAGO DE COMPOSTELA: Uso de la quimosina recombinante activa.Uso del cuajo, sin activación previa, obtenido a partir de una célula hospedadora que contiene un vector de expresión […]
 PROMOTORES MUTANTES DE AOX 1, del 5 de Abril de 2011, de TECHNISCHE UNIVERSITÄT GRAZ VTU HOLDING GMBH: Un promotor mutante de alcohol oxidasa 1 (AOX1) de Pichia pastoris del promotor de AOX1 de Pichia pastoris de tipo silvestre (SEC ID. Nº: 1) que comprende al […]
PROMOTORES MUTANTES DE AOX 1, del 5 de Abril de 2011, de TECHNISCHE UNIVERSITÄT GRAZ VTU HOLDING GMBH: Un promotor mutante de alcohol oxidasa 1 (AOX1) de Pichia pastoris del promotor de AOX1 de Pichia pastoris de tipo silvestre (SEC ID. Nº: 1) que comprende al […]
Biblioteca de péptidos y su uso, del 8 de Julio de 2020, de DAIICHI SANKYO COMPANY, LIMITED: Una biblioteca de péptidos que comprende una pluralidad de péptidos diferentes en la que los péptidos comprenden cada uno una secuencia de aminoácidos […]
Sistemas de conservación y procesamiento de espermatozoides, del 17 de Junio de 2020, de XY, LLC: Un procedimiento para producir una muestra de esperma de mamífero no humano adecuada para la fertilización in vitro, que incluye la etapa de: […]
Ratón nuligénico para Pint que muestra un fenotipo asociado a envejecimiento prematuro, del 10 de Junio de 2020, de REGENERON PHARMACEUTICALS, INC.: Un ratón cuyo genoma comprende una inactivación de un locus del ARN no codificante largo (ARNlnc) Pint endógeno, en donde la inactivación (i) da como resultado que el […]
COMPOSICIÓN A BASE DE BIOPOLÍMEROS RECOMBINANTES Y USOS DE LA MISMA COMO BIOTINTA, del 23 de Abril de 2020, de UNIVERSIDAD DE VALLADOLID: La presente invención se refiere a composiciones que comprenden biopoiímeros recombinantes formados por combinaciones de monómeros de tipo "Recombinámeros […]
Composiciones para tratar MPSI, del 22 de Abril de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Un vector que lleva un casete de expresión que comprende un gen de la alfa-L-iduronidasa humana (hIDUA) que tiene la secuencia de nucleótidos de SEQ ID NO: […]
Método para la producción de L-serina usando microorganismos modificados por ingeniería genética deficientes para las vías de degradación de la serina, del 22 de Abril de 2020, de CysBio ApS: Una bacteria que pertenece a la familia Enterobacteriaceae que se ha modificado para inactivar los genes sdaA, sdaB, tdcG y glyA.