AMPLIFICACIÓN UNIVERSAL DE RNA FRAGMENTADO.
Un método de procesamiento de RNA fragmentado que comprende una multiplicidad de especies de RNA obtenidas de una muestra de tejido fijada e incrustada en parafina para análisis de la expresión génica,
que comprende los pasos de: (a) poliadenilación del RNA fragmentado, y (b) conversión del RNA poliadenilado fragmentado obtenido en el paso (a) en cDNA
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2004/043163.
Solicitante: GENOMIC HEALTH, INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 301 PENOBSCOT DRIVE REDWOOD CITY, CA 94063 ESTADOS UNIDOS DE AMERICA.
Inventor/es: KIEFER, MICHAEL, C., HOYT,Kenneth,W.
Fecha de Publicación: .
Fecha Solicitud PCT: 21 de Diciembre de 2004.
Clasificación Internacional de Patentes:
- C12N15/10D
Clasificación PCT:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12N15/10 C12N 15/00 […] › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania.
PDF original: ES-2360113_T3.pdf





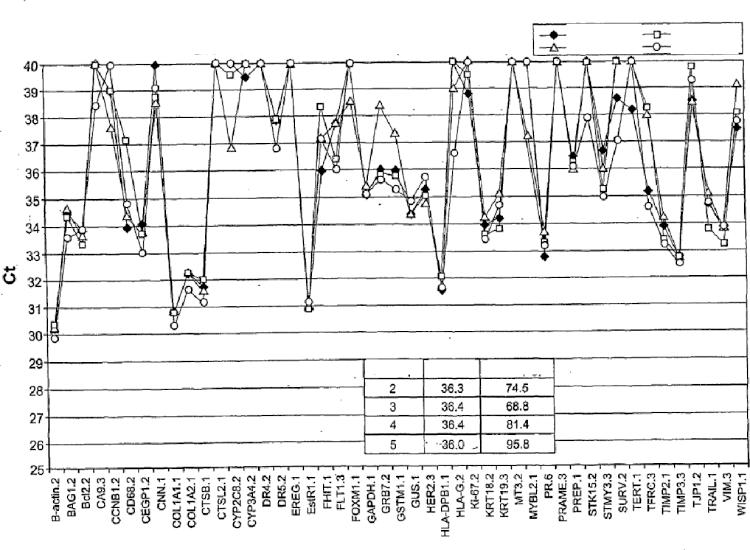
Fragmento de la descripción:
Campo de la invención
La presente invención se refiere a métodos de preparación de RNA para determinación del perfil de la expresión génica por una diversidad de métodos. Los métodos de la invención son particularmente útiles para amplificación universal de RNA, con inclusión de RNA en el cual una o más especies de RNA está(n) fragmentada(s) y/o bloqueada(s) en su término 3', tal como se obtiene a partir de tejido fijado incrustado en parafina (FPET). Los métodos son útiles también para detectar especies de RNA que carecen de poliadenilación. Adicionalmente, se proporcionan métodos RT-PCR mejorados para determinación del perfil útiles en la expresión génica.
Detección de la Técnica Afín
La determinación del perfil de la expresión génica está adquiriendo una importancia creciente tanto en investigación biológica como en la práctica clínica. La determinación del perfil de la expresión génica ha sido utilizada para clasificar diversos tipos de cáncer (véase v.g. Golub et al., Science 286:531-537 (1999); Bhattacharjae et al., Proc. Natl. Acad. Sci. USA 98:13790-13795 (2001); Chen-Hsiang et al., Bioinformatics 17 (Suppl. 1):S316-S322 (2001); Ramaswamy et al., Proc. Natl. Acad. Sci. USA 98:15149-15154 (2001); Martin et al., Cancer Res. 60:2232-2238 (2000); West et al., Proc. Natl. Acad. Sci. USA 98:11462-11467 (2001); Sorlie et al., Proc. Natl. Acad. Sci. USA 98:10869-10874 (2001); Yan et al., Cancer Res. 61:8375-8380 (2001)), y para predecir el desenlace clínico del cáncer, tal como cáncer de mama (Van't Veer et al., Nature 415: 530-536 (2002)) y cáncer de pulmón (Beer et al., Nat. Med. 8: 816-24 (2002)).
Los métodos utilizados más comúnmente conocidos en la técnica para la cuantificación de la expresión de mRNA en una muestra incluyen transferencia Northern e hibridación in situ (Parker & Barnes, Methods in Molecular Biology 106:247-283 (1999)); ensayos de protección de RNasas (Hod, Biotechniques 13:852-854 (1992)); microrredes (Schena et al., Proc. Natl. Acad. Sci. USA 93(2):106-149 (1996)), y reacción en cadena de polimerasa con transcriptasa inversa (RT-PCR) (Weis et al., Trends in Genetics 8:263-264 (1992)). Alternativamente, pueden emplearse anticuerpos que pueden reconocer dúplex específicos, con inclusión de dúplex de DNA, dúplex de RNA, y dúplex híbridos DNA-RNA
o dúplex DNA-proteína. De éstos, debido a su sensibilidad, reproducibilidad, e intervalo dinámico amplio, la RT-PCR en tiempo real está convirtiéndose en el método de elección para determinación del perfil con alta capacidad y expresión exacta.
En muchas situaciones en las que es potencialmente útil la determinación del perfil de la expresión génica, existe material insuficiente para análisis sin amplificación previa del RNA. Dado que el RNA no puede servir como molde para la PCR, el primer paso en la determinación del perfil de la expresión génica por RT-PCR es la transcripción inversa del molde de RNA en cDNA, seguida por su amplificación exponencial en una reacción PCR. La conversión de mRNA en cDNA se realiza típicamente por cebado con oligo dT del mRNA en presencia de la enzima transcriptasa inversa (RT). Este paso, sin embargo, no es eficaz si la fuente de mRNA es una muestra de tejido fijada incrustada en parafina, que puede haber estado archivada durante hasta 10-20 años, y en la cual el RNA está fuertemente degradado (Lewis et al.,
J. Pathol. 195: 66-71 (2001)).
Adicionalmente, si bien Frolova L et al., Nucleic Acids Research, enero 1978, vol. 5, no. 1, 10 enero 1978 (197801), páginas 285-295, describen "Enzymatic synthesis of DNA complementary to mitochondrial mRNA via reverse transcription", Emtage J S et al., Nucleic Acids Research. abril 1979, vol. 6, no. 4, abril 1979 (1979-04), páginas 1221-1239, describen "Polyadenylation and reverse transcription of influenza viral RNA", Hell A et al., Biochimica et Biophysica Acta, vol. 442, no. 1, 1976, páginas 37-49, describen "Synthesis of DNA complementary to human ribosomal RNA poly adenylated", y Amara Rama Rao et al., Nucleic Acids Research, Oxford University Press, Surrey, GB, vol. 25, no. 17, 1997, páginas 3465-3470, describen "Specific polyadenylation and purification of total messenger RNA from Escherichia coli", ninguna de dichas referencias describe los métodos para preparación de RNA de la presente invención.
Dado que las muestras de FPET son la fuente más disponible generalmente de RNA para determinación del perfil de la expresión génica en oncología clínica y debido a que las muestras archivadas de FPET son una fuente importante de RNA para la determinación del perfil de la expresión génica en oncología de investigación, existe una necesidad clínica de métodos que hagan posible y mejoren la eficacia de determinación del perfil de la expresión génica utilizando tales muestras de tejido.
Sumario de la Invención
La presente invención proporciona un método de preparación de muestras que permite una amplificación global incluso de muestras de RNA muy pequeñas o muy fragmentadas. El método de la invención mejora la sensibilidad de los métodos de análisis de RNA, con inclusión de RT-PCR y redes de hibridación. Adicionalmente, los métodos de la invención permiten la medida de los niveles de mRNA de todos los genes expresados con inclusión de especies de mRNA fragmentadas y/o bloqueadas en muestras de tejido archivadas incrustadas en parafina. Este método permite también la medida de mRNA no-poliadenilado tales como histonas y RNAs no codificantes, tales como microRNAs (miRNAs). La invención puede incluir adicionalmente un paso de transcripción inversa mejorado y un paso de PCR modificado que aumenta la sensibilidad de la RT-PCR utilizado para determinación del perfil de la expresión génica de las muestras de RNA fragmentadas.
De acuerdo con un primer aspecto de la presente invención, se proporciona un método de procesamiento de RNA fragmentado como se especifica en la reivindicación 1.
Típicamente, el tamaño de la especie de RNA en el RNA fragmentado está comprendido entre aproximadamente 20 bases y aproximadamente 2000 bases, de modo más frecuente entre aproximadamente 50 y aproximadamente 300 bases.
La poliadenilación puede, por ejemplo, realizarse con poliA-polimerasa de E. coli.
Dado que al menos algunas especies de RNA dentro del RNA fragmentado pueden estar bloqueadas en sus términos 3', el método de la invención puede incluir adicionalmente un paso de desbloqueo. El desbloqueo puede realizarse utilizando reactivos convencionales, tales como, por ejemplo, con una enzima fosfatasa, v.g. fosfatasa alcalina de ternero (CIP), fosfatasa alcalina bacteriana, fosfatasa alcalina de camarón, o variantes de las mismas, o con una polinucleótido-quinasa (PNK), v.g. polinucleótido-quinasa T4 (T4 PNK), o variantes de las mismas.
En una realización, el RNA poliadenilado fragmentado obtenido en el paso (a) anterior se convierte en cDNA por tratamiento con una transcriptasa inversa e iniciadores oligo-dT, donde los iniciadores oligo-dT pueden contener opcionalmente una secuencia promotora de RNA-polimerasa (v.g. el promotor de RNA-polimerasa T7).
En otra realización, antes de convertir en cDNA el RNA poliadenilado fragmentado obtenido en el paso (a) anterior, se elimina el agente de poliadenilación, tal como CIP o PNK.
En una realización adicional, el RNA poliadenilado fragmentado se convierte en cDNA sin eliminación previa del agente de poliadenilación, tal como CIP, o PNK.
En otra realización adicional, el RNA poliadenilado fragmentado se enriquece, v.g., por eliminación de secuencias de rRNA, antes de la conversión en cDNA y subsiguientemente en cDNA bicatenario.
En otra realización adicional, el RNA poliadenilado fragmentado se inmoviliza antes de la conversión en un cDNA.
En una realización particular, el RNA poliadenilado fragmentado se hibrida a un formato de perlas en fase sólida. Si se desea, el RNA poliadenilado fragmentado inmovilizado se enriquece antes de la conversión en cDNA. El enriquecimiento puede comprender la eliminación de secuencias de rRNA por hibridación a oligonucleótidos de rRNA complementarios inmovilizados en perlas.
En otra realización adicional, el RNA es un mRNA obtenido de una muestra de tumor, donde el tumor puede ser cáncer, tal como, por ejemplo, cáncer de mama, cáncer de colon, cáncer de pulmón, cáncer de próstata, cáncer hepatocelular,... [Seguir leyendo]
Reivindicaciones:
1. Un método de procesamiento de RNA fragmentado que comprende una multiplicidad de especies de RNA obtenidas de una muestra de tejido fijada e incrustada en parafina para análisis de la expresión génica, que comprende los pasos de:
(a) poliadenilación del RNA fragmentado, y
(b) conversión del RNA poliadenilado fragmentado obtenido en el paso (a) en cDNA.
2. El método de la reivindicación 1, en el que el tamaño de las especies de RNA en el RNA fragmentado está comprendido entre aproximadamente 20 bases y aproximadamente 2000 bases; en particular, en el que el tamaño medio de las especies de RNA contenidas en el RNA fragmentado está comprendido entre aproximadamente 50 y aproximadamente 300 bases.
3. El método de la reivindicación 1, en el que la poliadenilación se efectúa con poliA-polimerasa de E. coli.
4. El método de la reivindicación 1 que comprende adicionalmente el paso de desbloquear los términos 3' de las especies de RNA fragmentado antes del paso (a) con un agente desbloqueante.
5. El método de la reivindicación 4, en el que dicho agente desbloqueante es una enzima fosfatasa, en particular, en el que dicha fosfatasa se selecciona del grupo constituido por fosfatasa alcalina de ternero (CIP), fosfatasa alcalina bacteriana, fosfatasa alcalina de camarón, y variantes de las mismas; o, en el que dicho agente desbloqueante es una polinucleotido-quinasa (PNK), o una variante de la misma, en particular, en el que dicha polinucleotido-quinasa es polinucleotido-quinasa T4 (T4 PNK), o una variante de la misma.
6. El método de la reivindicación 4, en el que dicho agente desbloqueante se elimina antes de realizar el paso de poliadenilación (a); o, en el que el paso de poliadenilación (a) se realiza sin eliminación previa del agente desbloqueante, en particular, en el que dicho agente desbloqueante es una polinucleotido-quinasa (PNK).
7. El método de la reivindicación 1, en el que el RNA poliadenilado fragmentado obtenido en el paso (a) se convierte en cDNA por tratamiento con una transcriptasa inversa e iniciadores oligo-dT.
8. El método de la reivindicación 7, en el que dicha transcriptasa inversa se selecciona del grupo constituido por transcriptasa inversa del virus de la mieloblastosis aviar (AMV-RT), transcriptasa inversa del virus de la leucemia murina de Moloney (MMLV-RT), y transcriptasas inversas recombinantes heterodímeras expresadas en E. coli.
9. El método de la reivindicación 8, en el que dichos iniciadores oligo-dT contienen una secuencia promotora de RNA-polimerasa, en particular, en el que dicho promotor es un promotor de RNA-polimerasa T7.
10. El método de la reivindicación 7, en el que dicho RNA poliadenilado fragmentado obtenido en el paso (a) se inmoviliza antes de la conversión en cDNA; en particular, en el que dicha inmovilización se realiza sobre perlas; más particularmente, en el que el RNA poliadenilado fragmentado inmovilizado se enriquece antes de la conversión en cDNA; de modo muy particular, en el que el enriquecimiento comprende eliminación de secuencias de rRNA por hibridación a oligonucleótidos de rRNA complementario inmovilizados en perlas.
11. El método de la reivindicación 7, en el que dicha muestra de tejido procede de un tumor, más particularmente, en el que dicho tumor es cáncer; muy particularmente, en el que dicho cáncer se selecciona del grupo constituido por cáncer de mama, cáncer de colon, cáncer de pulmón, cáncer de próstata, cáncer hepatocelular, cáncer gástrico, cáncer pancreático, cáncer cervical, cáncer de ovario, cáncer de hígado, cáncer de vejiga, cáncer del tracto urinario, cáncer de tiroides, cáncer renal, carcinoma, melanoma, y cáncer de cerebro.
12. El método de la reivindicación 9, que comprende adicionalmente el paso de (c) amplificación por PCR utilizando una o más especies de cDNA presentes en el cDNA obtenido en el paso (b) como molde; particularmente, en el que la amplificación PCR comprende 40 ciclos, y los cinco primeros ciclos se llevan a cabo a una temperatura de reasociación/extensión más baja, en particular, en el que los dos primeros ciclos se realizan a una temperatura de reasociación/extensión más baja; o, en el que la amplificación PCR comprende 40 ciclos, y los primeros dos a cinco ciclos se realizan a una temperatura de aproximadamente 40ºC a 58ºC, en particular, en el que dichos ciclos iniciales se llevan a cabo a una temperatura de aproximadamente 50ºC.
13. El método de la reivindicación 9 que comprende adicionalmente los pasos de:
(d) conversión del cDNA obtenido en el paso (b) en DNA bicatenario; y
(e) amplificación del RNA por sometimiento del DNA bicatenario obtenido en el paso (d) a transcripción in vitro con una RNA-polimerasa para obtener RNA complementario (cRNA) amplificado.
14. El método de la reivindicación 13, en el que en el paso (d) cDNA se convierte en DNA bicatenario utilizando RNasa H y DNA-polimerasa I.
15. El método de la reivindicación 13, en el que el cRNA amplificado del paso (e) se utiliza directamente como molde en un ensayo de determinación del perfil de expresión génica por RT-PCR.
16. El método de la reivindicación 13, en el que el paso (e) incluye marcación del cRNA amplificado con un marcador detectable; en particular, en el que el marcador detectable es biotina o un marcador fluorescente.
17. El método de la reivindicación 13, que comprende someter el cRNA amplificado obtenido en el paso (e) a hibridación en una microrred.
18. El método de la reivindicación 1, en el que el RNA poliadenilado fragmentado obtenido en el paso (a) se convierte en cDNA por tratamiento con una transcriptasa inversa e iniciadores inversos extendidos, y el cDNA obtenido se amplifica por PCR utilizando iniciadores PCR directos e inversos no extendidos y sondas, diseñados sobre la base de amplicones diana.
19. El método de la reivindicación 18, en el que los iniciadores inversos extendidos se extienden 10, en particular 20, más particularmente 30 bases adicionales en el amplicón que el iniciador PCR inverso específico del gen.
20. El método de la reivindicación 18, en el que los iniciadores inversos extendidos se extienden en el amplicón dentro de una base del iniciador PCR directo.
21. Un método de procesamiento RNA que comprende una multiplicidad de especies de RNA obtenidas de una muestra de tejido fijada e incrustada en parafina para análisis de la expresión génica, que comprende los pasos de:
(a) poliadenilación de dicho RNA; y
(b) conversión del RNA poliadenilado en cDNA por transcriptasa inversa e iniciadores oligo dT u oligo dT-T7.
22. El método de la reivindicación 21, en el que dicho RNA comprende especies de RNA con términos 3' bloqueados; comprendiendo en particular desbloquear dichas especies de RNA bloqueadas antes del paso (a).
23. El método de la reivindicación 21 que comprende adicionalmente el paso de utilizar el cDNA cebado con oligo dT directamente para análisis de la expresión génica; en particular, en el que dicho análisis de la expresión génica se efectúa por TaqMan®.
24. El método de la reivindicación 21 que comprende adicionalmente el paso de conversión del cDNA cebado con oligo dT-T7 en DNA bicatenario con DNA-polimerasa I y RNAsa H, y amplificación del DNA bicatenario con RNApolimerasa T7 y rNTPs; o que comprende adicionalmente el paso de conversión del cDNA cebado con oligo dT-T7 en DNA bicatenario, y amplificación del DNA bicatenario con RNA-polimerasa T7 y rNTPs biotinilados.
25. Un método de procesamiento de RNA que comprende una multiplicidad de especies de RNA obtenidas de una muestra de tejido fijada e incrustada en parafina para análisis de la expresión génica, que comprende los pasos de:
(a) poliadenilación de dicho RNA; y
(b) conversión del RNA poliadenilado en cDNA por medio de transcriptasa inversa e iniciadores oligo dT-T7 que contienen un promotor de RNA-polimerasa T7; y
(c) sometimiento del DNA bicatenario obtenido en el paso (b) a transcripción in vitro con una RNA-polimerasa T7 para obtener RNA complementario (cRNA) amplificado.
Patentes similares o relacionadas:
 COMPOSICIONES Y MÉTODOS PARA LA REACCIÓN EN CADENA DE LA POLIMERASA DE TRANSCRIPTASA INVERSA (RT-PCR), del 30 de Marzo de 2011, de LIFE TECHNOLOGIES CORPORATION: Un procedimiento para amplificar una molécula de ácido nucleico, dicho procedimiento que comprende (a) mezclar un molde de ARN con una composición que […]
COMPOSICIONES Y MÉTODOS PARA LA REACCIÓN EN CADENA DE LA POLIMERASA DE TRANSCRIPTASA INVERSA (RT-PCR), del 30 de Marzo de 2011, de LIFE TECHNOLOGIES CORPORATION: Un procedimiento para amplificar una molécula de ácido nucleico, dicho procedimiento que comprende (a) mezclar un molde de ARN con una composición que […]
METODO DE PRODUCCION DE ADNC DE LONGITUD COMPLETA MEDIANTE LIGACION DE ADAPTADOR DE EXTREMO COHESIVO, del 10 de Mayo de 2010, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE (CNRS) INSTITUT NATIONAL DE LA RECHERCHE AGRONOMIQUE: Método de producción de ADNc de longitud completa que comprende una etapa a) de desfosforilación de los fragmentos de ácidos nucleicos no provistos de un protector en 5'', […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.