INTEGRACION ALEATORIA DE UN POLINUCLEOTIDO DESPUES DE LINEALIZACION IN VIVO.
Un procedimiento no terapéutico para integrar aleatoriamente un polinucleótido en un genoma de la célula huésped mediante preparación en la célula huésped de dicho polinucleótido lineal que tiene extremos 5'' y 3'' libres a partir de un vector,
en el que dicho procedimiento comprende:
a) Introducir en dicha célula huésped un vector que no tiene extremos 5'' y 3'' libres y que comprende dicho polinucleótido, comprendiendo dicho vector al menos un sitio de escisión que no se encuentra en el genoma de la célula huésped;
b) producir la escisión de dicho(s) sitio(s) en dicha célula huésped, creando o liberando así dicho polinucleótido en una forma lineal que tiene extremos 5'' y 3'' libres a partir de dicho vector en dicha célula huésped; y,
c) mantener la célula huésped en condiciones y durante un periodo de tiempo suficiente para producir la integración aleatoria de dicho polinucleótido liberado en dicho genoma de la célula huésped,
y en el que dicha célula huésped es una célula no humana
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP02/10224.
Solicitante: CELLECTIS.
Nacionalidad solicitante: Francia.
Dirección: 102, ROUTE DE NOISY,93230 ROMAINVILLE.
Inventor/es: CHOULIKA, ANDRE, JOLY,JEAN-STEPHANE, THERMES,VIOLETTE, RISTORATORE,FILOMENA.
Fecha de Publicación: .
Fecha Concesión Europea: 28 de Abril de 2010.
Clasificación Internacional de Patentes:
- C12N15/90 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Introducción estable de ADN extraño en el cromosoma.
Clasificación PCT:
- A01K67/027 NECESIDADES CORRIENTES DE LA VIDA. › A01 AGRICULTURA; SILVICULTURA; CRIA; CAZA; CAPTURA; PESCA. › A01K CRÍA DE ANIMALES; AVICULTURA; APICULTURA; PISCICULTURA; PESCA; ANIMALES PARA CRIA O REPRODUCCIÓN, NO PREVISTOS EN OTRO LUGAR; NUEVAS VARIEDADES DE ANIMALES. › A01K 67/00 Cría u obtención de animales, no prevista en otro lugar; Nuevas razas de animales. › Nuevas razas de vertebrados.
- C12N15/00 C12N […] › Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K).
- C12N15/55 C12N 15/00 […] › Hidrolasas (3).
- C12N15/64 C12N 15/00 […] › Métodos generales para la preparación del vector, para su introducción en la célula o para la selección del huésped que contiene el vector.
- C12N15/79 C12N 15/00 […] › Vectores o sistemas de expresión especialmente adaptados a huéspedes eucariotas.
- C12N15/90 C12N 15/00 […] › Introducción estable de ADN extraño en el cromosoma.
- C12N5/10 C12N […] › C12N 5/00 Células no diferenciadas humanas, animales o vegetales, p. ej. líneas celulares; Tejidos; Su cultivo o conservación; Medios de cultivo para este fin (reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00). › Células modificadas por introducción de material genético extraño, p. ej. células transformadas por virus.
Clasificación antigua:
- A01K67/027 A01K 67/00 […] › Nuevas razas de vertebrados.
- C12N15/00 C12N […] › Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K).
- C12N15/55 C12N 15/00 […] › Hidrolasas (3).
- C12N15/64 C12N 15/00 […] › Métodos generales para la preparación del vector, para su introducción en la célula o para la selección del huésped que contiene el vector.
- C12N15/79 C12N 15/00 […] › Vectores o sistemas de expresión especialmente adaptados a huéspedes eucariotas.
- C12N15/90 C12N 15/00 […] › Introducción estable de ADN extraño en el cromosoma.
- C12N5/10 C12N 5/00 […] › Células modificadas por introducción de material genético extraño, p. ej. células transformadas por virus.
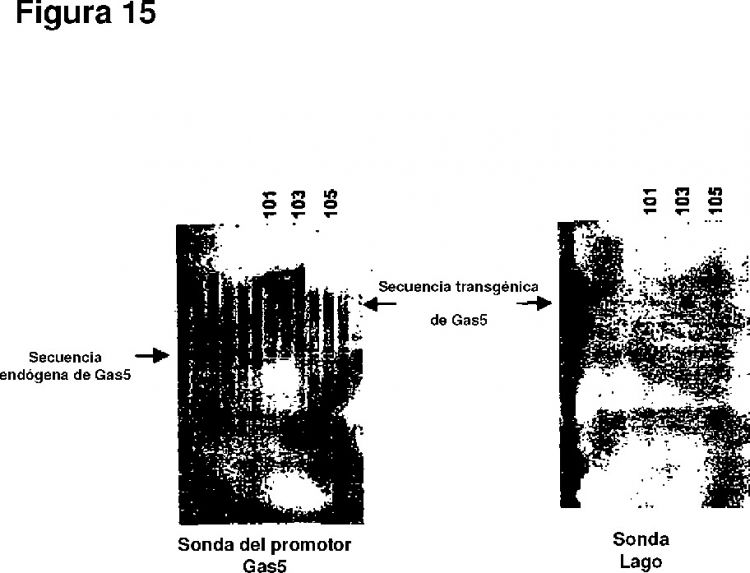
Fragmento de la descripción:
Integración aleatoria de un polinucleótido después de linealización in vivo.
Antecedentes
La invención se refiere a un procedimiento de liberación in vivo de un fragmento de ADN lineal de un vector con el fin de integrar este fragmento en el genoma celular. La invención se refiere además al uso de este procedimiento y a los resultados de este uso.
El primer gran avance de la genética inversa hace 20 años fue la transgénesis. La transgénesis es la técnica que permite introducir una secuencia de ADN exógeno en una célula huésped. Por ejemplo, la microinyección de ADN en un huevo fecundado por mamífero conduce, en un cierto número de casos, a la integración de la molécula de ADN microinyectado en el genoma del huevo fecundado. La transgénesis implica que un fragmento de ADN extraño se introduzca en el genoma de un organismo multicelular y se transmita a la progenie. Por tanto, el ADN extraño debe estar presente en una forma estable en el embrión en una fase precoz de desarrollo con el fin de transmitirse a la progenie.
La transfección de ADN en células de mamífero por medio de precipitación de fosfato de calcio precipitado también puede conducir, en un cierto número de casos, a la integración del ADN exógeno en el genoma de la célula huésped que se llama transfección estable. El ADN exógeno puede introducirse en células bajo dos formas, bien lineal o circular. Cuando el ADN se introduce en forma lineal, el fragmento lineal se prepara in vitro antes de su introducción en la célula huésped, generalmente por escisión del fragmento deseado con enzima de restricción de, por ejemplo, un plásmido. En lo que se refiere al ADN en forma circular, el ADN introducido es generalmente un plásmido superenrollado.
Todas las células tienen sistemas de mantenimiento y reparación de su ADN. Una de las señales particulares de estrés que activa la intervención de estos sistemas es la generación de extremos libres de ADN en la célula. La célula tiene entonces dos soluciones para resolver este tipo de problema:
Como consecuencia, los trabajos que tienen como objetivo integrar el ADN exógeno mediante la introducción de ADN bicatenario desnudo en forma circular o lineal se encuentran con tres tipos de problemas principales, además de un cierto número de problemas colaterales.
La transgénesis es más que nunca una herramienta esencial para los biólogos. El estudio de enfermedades humanas se basa en gran parte en el uso de modelos animales. Están disponibles una gran cantidad de informaciones de secuencias génicas para la industria farmacéutica. Estas informaciones proporcionarían nuevas dianas para fármacos. Sin embargo, la función de la mayoría de los genes y las proteínas relacionadas en un organismo sigue siendo desconocida y la eficiencia de la validación de dianas no parece haber cambiado significativamente.
Los estudios genéticos y la genómica estructural han mostrado que las rutas bioquímicas y la fisiología están sumamente conservadas en todo el mundo animal. Por tanto, pueden usarse modelos animales para investigar procedimientos relevantes para enfermedades humanas. El modelo animal puede usarse para identificar y validar eficazmente dianas de cribado óptimas. La selección de dianas de cribado puede mejorarse y conducirá a menos fallos y a un procedimiento de desarrollo de fármacos más eficaz.
El ratón es un modelo de mamífero muy conocido que se usa abundantemente. El procedimiento más ampliamente usado para la producción de animales transgénicos es la microinyección de ADN en los pronúcleos de embriones fecundados. Este procedimiento es bastante eficiente para la producción de ratones transgénicos pero es mucho menos eficaz para la producción de mamíferos transgénicos grandes tales como vacas y ovejas. Además, los animales transgénicos del procedimiento de transgénesis disponible son frecuentemente un mosaico para el transgén dando como resultado la falta de transmisión del transgén a la progenie. Algunos animales presentan una alta resistencia a la transgénesis tales como peces o aves. Entre ellos, el problema de transgénesis de peces se detalla más a continuación.
Los peces de pecera han alcanzado una alta popularidad como modelos de vertebrados en la biología y la genética del desarrollo. De hecho, el pez cebra (Danio rerio) es un sistema de modelo popular para estudios de desarrollo de vertebrados porque ofrece la oportunidad de combinar análisis de genética clásica con un embrión fácilmente accesible y manipulable. Los estudios genéticos de los peces cebra se benefician del tiempo de generación de 2-3 meses, la capacidad de las hembras de poner rutinariamente cientos de huevos y el pequeño tamaño de los adultos. Los estudios embriológicos se benefician de los grandes embriones transparentes.
Sin embargo, la utilidad de peces de pecera está todavía limitada por la falta de algunas herramientas metodológicas, sobre todo una tecnología simple y eficaz para la transgénesis, que se ha convertido en una técnica importante en la investigación fundamental y tiene varias aplicaciones en agronomía y biomedicina. En particular, los intentos por establecer células madre embriónicas (ES) en peces como vectores celulares para la transgénesis ha sido hasta la fecha poco satisfactorios.
Por tanto, el procedimiento de elección para generar peces transgénicos sigue siendo la inyección...
Reivindicaciones:
1. Un procedimiento no terapéutico para integrar aleatoriamente un polinucleótido en un genoma de la célula huésped mediante preparación en la célula huésped de dicho polinucleótido lineal que tiene extremos 5' y 3' libres a partir de un vector, en el que dicho procedimiento comprende:
y en el que dicha célula huésped es una célula no humana.
2. Un procedimiento in vitro para integrar aleatoriamente un polinucleótido en un genoma de la célula huésped mediante preparación en la célula huésped de dicho polinucleótido lineal que tiene extremos 5' y 3' libres a partir de un vector, en el que dicho procedimiento comprende:
y en el que dicha célula huésped es una célula somática no embriónica humana.
3. Procedimiento según la reivindicación 1 ó 2, en el que dicho procedimiento comprende además, antes de la etapa (b), una etapa adicional de introducir en dicha célula huésped un agente de escisión o un vector que comprende un ácido nucleico que codifica dicho agente de escisión.
4. Procedimiento según la reivindicación 3, en el que dicho procedimiento comprende además, antes de la etapa (b), una etapa adicional de introducir en dicha célula huésped un agente de escisión.
5. Procedimiento según la reivindicación 1-4, en el que dicho polinucleótido está flanqueado por al menos un sitio de escisión.
6. Procedimiento según la reivindicación 5, en el que dicho polinucleótido está flanqueado por dos sitios de escisión.
7. Procedimiento según la reivindicación 1-6, en el que dicho sitio de escisión es un sitio de endonucleasa y dicho agente de escisión es la endonucleasa correspondiente.
8. Procedimiento según la reivindicación 7, en el que dicha endonucleasa tiene un sitio de reconocimiento de al menos 12 nucleótidos.
9. Procedimiento según la reivindicación 8, en el que dicha endonucleasa es una meganucleasa.
10. Procedimiento según la reivindicación 9, en el que dicha meganucleasa se selecciona del grupo que está constituido por I-Ceu I, I-Cre I, I-Chu I, I-Csm I, I-Dmo I, I-Pan I, I-Sce I, I-Sce II, I-Sce III, I-Sce IV, F-Sce I, F-Sce II, PI-Aae I, PI-Ape-I, PI-Ceu I, PI-Cir I, PI-Ctr I, PI-Dra I, PI-Mav I, PI-MfI I, PI-Mgo I, PI-Mja I, PI-Mka I, PI-Mle I, PI-Mtu I, PI-MtuH I, PI-Pab III, PI-Pfu I, PI-Pho I, PI-Pko I, PI-Psp I, PI-Rma I, PI-Sce I, PI-Ssp I, PI-Tfu I, PI-Tfu II, PI-TIi I, PI-TIi II, PI-Tsp I, PI-Tsp II, PI-Bsp I, PI-Mch I, PI-Mfa I, PI-Mga I, PI-Mga II, PI-Min I, PI-Mma I, PI-Msh I, PI-Msm II, PI-Mth I, PI-Tag I, PI-Thy II, I-Ncr I, I-Ncr II, I-Pan II, I-Tev I, I-Ppo I, I-Dir I, I-Hmu I, I-Hmu II, I-Tev II, I-Tev III, F-Sce I, F-Sce II (HO), F-Suv I, F-Tev I y F-Tev II.
11. Procedimiento según la reivindicación 10, en el que dicha meganucleasa se selecciona del grupo que está constituido por I-Ceu I, I-Cre I, I-Sce I.
12. Procedimiento según la reivindicación 8, en el que dicha endonucleasa es sintética.
13. Procedimiento según la reivindicación 1-12, en el que dicho polinucleótido no puede someterse a recombinación homóloga con el genoma de la célula huésped.
14. Procedimiento según la reivindicación 13, en el que las secuencias de 5' y 3' de dicho polinucleótido no tienen homología con una secuencia genómica de la célula huésped.
15. Procedimiento según la reivindicación 1-14, en el que dicho vector es un plásmido.
16. Procedimiento según una cualquiera de las reivindicaciones 1 y 3-15, en el que dicha célula huésped no humana se selecciona del grupo que está constituido por una célula madre, una célula somática, un gameto, un blastómero y un huevo.
17. Procedimiento según la reivindicación 16, en el que dicha célula huésped no humana es una célula madre.
18. Procedimiento según la reivindicación 16, en el que dicha célula huésped no humana es una célula somática.
19. Procedimiento según la reivindicación 16, en el que dicha célula huésped no humana es un huevo.
20. Procedimiento según la reivindicación 1-19, en el que dicha secuencia de polinucleótidos es una secuencia que codifica un polipéptido o un antisentido, una secuencia reguladora o una secuencia de reconocimiento para una molécula.
21. Procedimiento según la reivindicación 17, en el que dicha célula huésped no humana proviene de pez, ave, mamíferos no humanos, insecto, anfibio o reptil.
22. Procedimiento según la reivindicación 21, en el que dicha célula huésped proviene de medaka, pez cebra, pez espinoso, Astyanax, ratones, pollos, Xenopus, ovejas, vaca, conejo.
23. Procedimiento según una cualquiera de las reivindicaciones 1 y 3-22, en el que el procedimiento comprende además la etapa de generar una célula de mamífero o pez, un animal mamífero no humano o pez transgénicos o una planta transgénica para producir proteína, biomolécula, biomaterial o vacuna.
24. Un procedimiento para producir un animal transgénico no humano, en el que células madre embriónicas se transfectan mediante el procedimiento según la reivindicación 1 y 3-22, las células se inyectan en embriones en una fase en la que pueden integrar las células transfectadas, los embriones se reimplantan luego en una madre sustituta y los individuos quiméricos obtenidos al final de la gestación, y en los que se observa la colonización por células madre embriónicas de la línea germinal, se aparean para obtener animales transgénicos.
25. Un procedimiento para producir un animal transgénico no humano en el que el huevo fecundado se transfecta mediante el procedimiento según la reivindicación 1 y 3-22, los huevos se reimplantan en una madre sustituta y los individuos transgénicos se obtienen al final de la gestación.
26. Un procedimiento para producir un animal transgénico no humano en el que el huevo fecundado se transfecta mediante el procedimiento según la reivindicación 1 y 3-22, los huevos se incuban en condiciones apropiadas para desarrollar dicho animal transgénico.
27. Una composición para transgénesis que comprende:
28. Composición según la reivindicación 27, en la que dicho polinucleótido está flanqueado por al menos un sitio de escisión.
29. Composición según la reivindicación 28, en la que dicho polinucleótido está flanqueado por dos sitios de escisión.
30. Composición según la reivindicación 26-29, en la que dicho sitio de escisión es un sitio de endonucleasa y dicho agente de escisión es la endonucleasa correspondiente.
31. Composición según la reivindicación 30, en la que dicha endonucleasa tiene un sitio de reconocimiento de al menos 12 nucleótidos.
32. Composición según la reivindicación 31, en la que dicha endonucleasa es una meganucleasa.
33. Composición según la reivindicación 32, en la que dicha meganucleasa se selecciona del grupo que está constituido por I-Ceu I, I-Cre I, I-Chu I, I-Csm I. I-Dmo I, I-Pan I, I-Sce I, I-Sce II, I-Sce III. I-Sce IV, F-Sce I, F-Sce II, PI-Aae I, PI-Ape I, PI-Ceu I, PI-Cir I, PI-Ctr I, PI-Dra I, PI-Mav I, PI-Mfl I, PI-Mgo I, PI-Mja I, PI-Mka I, PI-Mle I, PI-Mtu I, PI-MtuH I, PI-Pab III, PI-Pfu I, PI-Pho I, PI-Pko I, PI-Psp I, PI-Rma I, PI-Sce I, PI-Ssp I, PI-Tfu I, PI-Tfu II, PI-Tli I, PI-Tli II, PI-Tsp I, PI-Tsp II, PI-Bsp I, PI-Mch I, PI-Mfa I, PI-Mga I, PI-Mga II, PI-Min I, PI-Mma I, PI-Msh I, PI-Msm II, PI-Mth I, PI-Tag I, PI-Thy II, I-Ncr I, I-Ncr II, I-Pan II, I-Tev I, I-Ppo I, I-Dir I, I-Hmu I, I-Hmu II, I-Tev II, I-Tev III, F-Sce I, F-Sce II (HO), F-Suv I, F-Tev I y F-Tev II.
34. Composición según la reivindicación 33, en la que dicha meganucleasa se selecciona del grupo que está constituido por I-Ceu I, I-Cre I, I-Sce I.
35. Composición según la reivindicación 32, en la que dicha endonucleasa es sintética.
36. Composición según la reivindicación 27-35, en la que dicho vector es un plásmido.
37. Composición según la reivindicación 27-36, en la que dicho vector que comprende dicha secuencia de polinucleótidos que va a linealizarse comprende además dicho ácido nucleico que codifica dicho agente de escisión.
38. Composición según la reivindicación 27-37, en la que dicho polinucleótido no puede someterse a recombinación homóloga con el genoma de la célula huésped.
39. Composición según la reivindicación 38, en la que las secuencias de 5' y 3' de dicho polinucleótido no tienen homología con una secuencia genómica de la célula huésped.
Patentes similares o relacionadas:
 Animales no humanos que tienen un locus de cadena ligera lambda de inmunoglobulina modificado por ingeniería, del 29 de Julio de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor cuyo genoma de la línea germinal comprende un locus de cadena ligera λ de inmunoglobulina endógeno que comprende:
(a) uno o más segmentos […]
Animales no humanos que tienen un locus de cadena ligera lambda de inmunoglobulina modificado por ingeniería, del 29 de Julio de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor cuyo genoma de la línea germinal comprende un locus de cadena ligera λ de inmunoglobulina endógeno que comprende:
(a) uno o más segmentos […]
Métodos y composiciones para el tratamiento de enfermedades por almacenamiento lisosomal, del 27 de Mayo de 2020, de Sangamo Therapeutics, Inc: Uno o más transgenes y una o más nucleasas con dedos de zinc (ZFN) para su uso en un método de tratamiento de una enfermedad por almacenamiento lisosomal, el método […]
Terapias de aumento génico de la degeneración retiniana causada por mutaciones en el gen PRPF31, del 6 de Mayo de 2020, de MASSACHUSETTS EYE & EAR INFIRMARY: Un vector de virus adeno-asociado de tipo 2 (AAV2) que comprende una secuencia que codifica PRPF31 humano, operativamente enlazado a un promotor […]
Proteínas que tienen actividad nucleasa, proteínas de fusión y usos de estas, del 18 de Marzo de 2020, de HELMHOLTZ ZENTRUM MUNCHEN DEUTSCHES FORSCHUNGSZENTRUM FUR GESUNDHEIT UND UMWELT (GMBH): Una molécula de ácido nucleico que codifica (I) un polipéptido que tiene la actividad de una endonucleasa, que es (a) una molécula de ácido nucleico que […]
Animales no humanos que tienen una interrupción en un locus C9ORF72, del 19 de Febrero de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor que comprende en su genoma una deleción de la porción codificante del exón 2 hasta la porción codificante del exón 11 de un locus C9orf72 endógeno, […]
Métodos y composiciones para modificar un locus objetivo, del 12 de Febrero de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para la modificación en serie de un locus objetivo en una célula, que comprende: (a) proporcionar la célula que comprende el locus objetivo, en donde el locus objetivo […]
Modelo de cerdo para la diabetes, del 29 de Enero de 2020, de AARHUS UNIVERSITET: Un cerdo transgénico que comprende un gen de polipéptido amiloide de los islotes (IAPP) mutado humano o parte del mismo, y que muestra al menos […]
Procedimientos de modificación de células hospedadoras, del 22 de Enero de 2020, de GLAXOSMITHKLINE BIOLOGICALS S.A.: Una célula hospedadora procariota aislada, en la que i) un gen heterólogo que codifica una oligosacaril transferasa y ii) ADN de al menos 8 kb que codifica […]