INSERCION MEJORADA DIRECCIONADA DE DNA EN LAS PLANTAS.
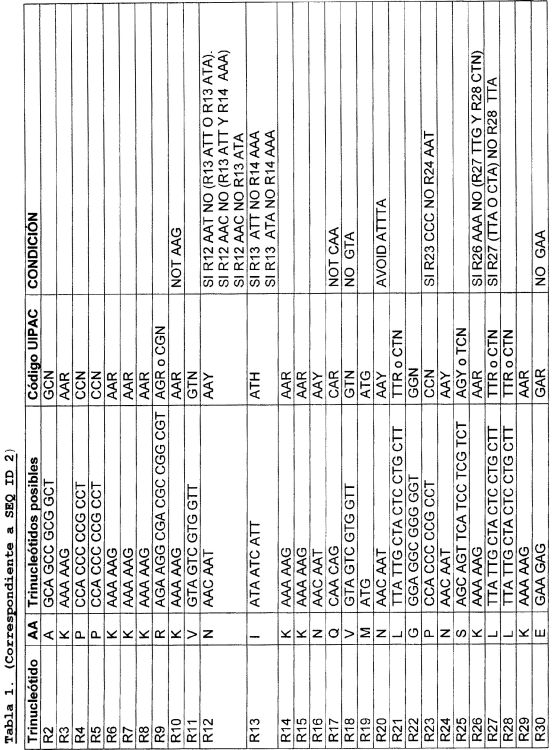
Un fragmento de DNA aislado que comprende una secuencia de nucleótidos que codifica una endonucleasa I-SceI en donde dicha secuencia de nucleótidos comprende la secuencia de nucleótidos de SEQ ID No.
4 o cualquier secuencia de nucleótidos obtenida por reemplazamiento de 1 a 20 nucleótidos de SEQ ID No. 4 por cualquiera de las alternativas proporcionadas en la secuencia de nucleótidos de SEQ ID No. 3, de tal manera que el contenido de GC de dicha secuencia de nucleótidos es aproximadamente 50 a aproximadamente 60%, con la condición de que #a) si la secuencia de nucleótidos desde la posición 121 a la posición 123 es GAG, entonces la secuencia de nucleótidos desde la posición 124 a la 126 no es CAA; #b) si la secuencia de nucleótidos desde la posición 253 a la posición 255 es GAC, entonces la secuencia de nucleótidos desde la posición 256 a 258 no es CAA; #c) si la secuencia de nucleótidos desde la posición 277 a la posición 279 es CAT entonces la secuencia de nucleótidos desde la posición 280 a 282 no es AAA; #d) si la secuencia de nucleótidos desde la posición 340 a la posición 342 es AAG entonces la secuencia de nucleótidos desde la posición 343 a la posición 345 no es CAT; #e) si la secuencia de nucleótidos desde la posición 490 a la posición 492 es TCA entonces la secuencia de nucleótidos desde la posición 493 a la posición 495 no es ACC; #f) si la secuencia de nucleótidos desde la posición 499 a la posición 501 es AAA entonces la secuencia de nucleótidos desde la posición 502 a 504 no es TCA o AGC; #g) si la secuencia de nucleótidos desde la posición 517 a la posición 519 es ACC, entonces la secuencia de nucleótidos desde la posición 520 a la posición 522 no es CAA simultáneamente al hecho de que la secuencia de nucleótidos desde la posición 523 a 525 sea TCN; #h) si la secuencia de nucleótidos desde la posición 661 a la posición 663 es ATT, entonces la secuencia de nucleótidos desde la posición 664 a la posición 666 no es AAA; #i) los codones de la secuencia de nucleótidos desde la posición 7 a la posición 15 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de siete nucleótidos contiguos del grupo de G o C; #j) los codones de la secuencia de nucleótidos desde la posición 61 a la posición 69 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de siete nucleótidos contiguos del grupo de G o C; #k) los codones de la secuencia de nucleótidos desde la posición 130 a la posición 138 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos no comprende un tramo de siete nucleótidos contiguos del grupo de G o C; #l) los codones de la secuencia de nucleótidos desde la posición 268 a la posición 279 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de siete nucleótidos contiguos del grupo de G o C; #m) los codones de la secuencia de nucleótidos desde la posición 322 a la posición 333 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de siete nucleótidos contiguos del grupo de G o C; #n) los codones de la secuencia de nucleótidos desde la posición 460 a la posición 468 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de siete nucleótidos contiguos del grupo de G o C; #o) los codones de la secuencia de nucleótidos desde la posición 13 a la posición 27 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #p) los codones de la secuencia de nucleótidos desde la posición 37 a la posición 48 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #q) los codones de la secuencia de nucleótidos desde la posición 184 a la posición 192 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #r) los codones de la secuencia de nucleótidos desde la posición 214 a la posición 219 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #s) los codones de la secuencia de nucleótidos desde la posición 277 a la posición 285 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #t) los codones de la secuencia de nucleótidos desde la posición 388 a la posición 396 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #u) los codones de la secuencia de nucleótidos desde la posición 466 a la posición 474 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #v) los codones de la secuencia de nucleótidos desde la posición 484 a la posición 489 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #w) los codones de la secuencia de nucleótidos desde la posición 571 a la posición 576 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #x) los codones de la secuencia de nucleótidos desde la posición 598 a la posición 603 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #y) los codones de la secuencia de nucleótidos desde la posición 604 a la posición 609 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #z) los codones de la secuencia de nucleótidos desde la posición 613 a la posición 621 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #aa) los codones de la secuencia de nucleótidos desde la posición 646 a la posición 651 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; #bb) los codones de la secuencia de nucleótidos desde la posición 661 a la posición 666 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T; y #cc) los codones de la secuencia de nucleótidos desde la posición 706 a la posición 714 se seleccionan de acuerdo con las opciones proporcionadas de tal manera que la secuencia de nucleótidos resultante no comprende un tramo de cinco nucleótidos contiguos del grupo de A o T.
Tipo: Resumen de patente/invención.
Solicitante: BAYER BIOSCIENCE N.V..
Nacionalidad solicitante: Bélgica.
Dirección: TECHNOLOGIEPARK 38,9052 GENT.
Inventor/es: D\'HALLUIN, KATHLEEN, VANDERSTRAETEN,CHANTAL, RUITER,RENE.
Fecha de Publicación: .
Fecha Concesión Europea: 31 de Diciembre de 2008.
Clasificación PCT:
- C12N15/82 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › para células vegetales.
- C12N9/22 C12N […] › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › Ribonucleasas.
Patentes similares o relacionadas:
Composiciones para la supresión de la formación de inhibidores contra el factor VIII en pacientes con hemofilia A., del 22 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Una composición que comprende material vegetal liofilizado que comprende al menos un fragmento de FVIII conjugado con la subunidad B de la toxina […]
Plantas con rendimiento incrementado y método de obtención de dichas plantas, del 15 de Julio de 2020, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE (CNRS): Método para mejorar por lo menos un carácter fenotípico seleccionado de entre el rendimiento en semillas, la velocidad de germinación o el crecimiento […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Moléculas de ácido nucleico de Nucampholin para controlar plagas de insectos coleópteros, del 17 de Junio de 2020, de FRAUNHOFER-GESELLSCHAFT ZUR FORDERUNG DER ANGEWANDTEN FORSCHUNG E.V.: Una molécula de ácido nucleico aislada que comprende un polinucleótido que codifica una molécula de ácido ribonucleico de horquilla (hpARN) con una estructura […]
Polinucleótidos aislados y métodos y plantas que usan los mismos para regular la acidez de las plantas, del 10 de Junio de 2020, de The State of Israel, Ministry of Agriculture and Rural Development, Agricultural Research Organization, (A.R.O.), Volcani Cent: Una célula de planta o una planta que comprende una construcción de ácido nucleico que comprende una secuencia de ácido nucleico que codifica un polipéptido […]
Genes de Chromobacterium subtsugae, del 27 de Mayo de 2020, de Marrone Bio Innovations, Inc: Una célula vegetal que comprende un polipéptido que tiene la secuencia de aminoácidos expuesta en las SEQ ID NO: 1, 2 o 3.
Moléculas polinucleotídicas para la regulación génica en plantas, del 6 de Mayo de 2020, de MONSANTO TECHNOLOGY, LLC: Un procedimiento para regular la expresión de un gen diana endógeno en plantas en crecimiento, que comprende: aplicar por vía tópica sobre […]
Producción de partículas similares al virus de la gripe en plantas, del 6 de Mayo de 2020, de MEDICAGO INC.: Un ácido nucleico que comprende una región reguladora activa en una planta y un potenciador de la expresión activo en una planta, la región […]