Recombinación de ADN específica de secuencia en células eucariotas.
Procedimiento in vitro o ex vivo para expresar al menos un gen de interés que codifica para uno o más producto(s)/polipéptido(s) terapéutico(s) deseado(s) en una célula eucariota,
en el que la célula eucariota no es un ovocito humano ni una célula madre embrionaria humana, que comprende
a) introducir de un primer ADN que comprende una secuencia attB de SEQ ID n.º: 13, una attP de SEQ ID n.º 14, una attL de SEQ ID n.º 15 o una attR de SEQ ID n.º 16 o un derivado de la misma en el interior de una célula;
b) seleccionar subclones candidatos en un biorreactor a pequeña escala que imita un proceso de producción de alimentación por lotes basado en parámetros tales como una alta viabilidad a alta densidad celular, el metabolismo y la capacidad de permitir una alta expresión y un rendimiento reproducible durante la producción,
c) introducir un segundo ADN que comprende una secuencia attB de SEQ ID n.º: 13, attP de SEQ ID n.º: 14, attL de SEQ ID n.º: 15 o attR de SEQ ID n.º: 16 o un derivado de la misma, y al menos un gen de interés que codifica para una proteína terapéutica en la célula del paso b);
d) poner en contacto dicha célula con una integrasa Int de bacteriófago lambda;
e) realizar la recombinación específica de secuencia por una integrasa Int de bacteriófago lambda, en donde el segundo ADN se integra dentro del primer ADN;
f) cultivar dicha célula en condiciones en las que se expresa(n) el(los) gen(es) de interés, en las que dicha célula se cultiva en medio libre de suero y en cultivo en suspensión; y
g) aislar dicho producto(s)/polipéptido(s) terapéutico(s) deseado(s) de la célula anfitriona o del medio de cultivo celular, en donde dicho derivado se define como una secuencia attB, attP, attL y attR que tiene una, dos, tres, cuatro, cinco, seis o siete sustituciones en la región de superposición y/o en la región de núcleo, o al menos un sitio de unión Int a núcleo de attB, attP, attL o attR, o al menos un sitio de unión Int a núcleo de attP, attL o attR más una o más copias de los sitios de unión a brazo para Int, o al menos un sitio de unión Int a núcleo de attP, attL o attR más una o más copias de los sitios de unión de factor IHF, FIS o XIS, o combinaciones de los mismos.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2003/013414.
Solicitante: BOEHRINGER INGELHEIM PHARMA GMBH & CO.KG.
Nacionalidad solicitante: Alemania.
Dirección: BINGER STRASSE 173 55216 INGELHEIM AM RHEIN ALEMANIA.
Inventor/es: ENENKEL,BARBARA, DROGE,PETER.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/90 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Introducción estable de ADN extraño en el cromosoma.
PDF original: ES-2507665_T3.pdf
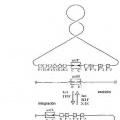


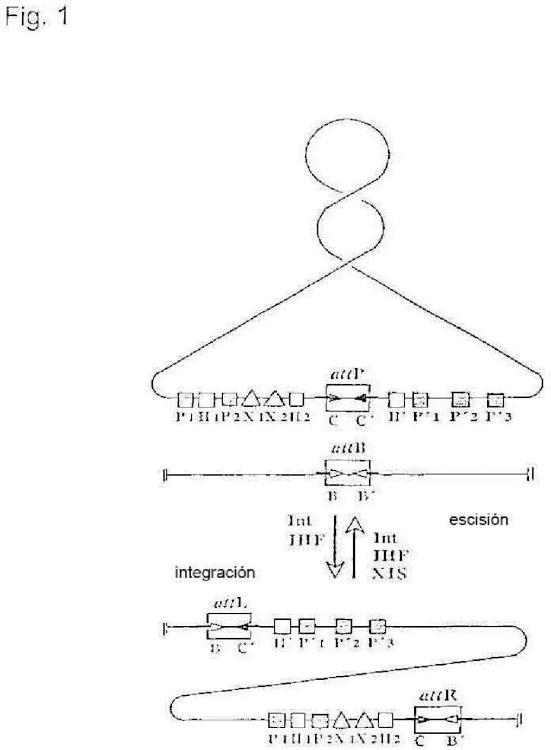
Fragmento de la descripción:
Recombinación de ADN específica de secuencia en células eucariotas
[1] La presente invención se refiere a un procedimiento in vitro o ex vivo para expresar al menos un gen de interés que codifica para uno o más producto(s)/polipéptido(s) deseado(s) en una célula eucariota, que comprende la introducción en una célula de un primer ADN que comprende una secuencia nucleotídica que contiene al menos una secuencia de recombinación, la introducción en el interior de una célula de un segundo ADN que comprende una secuencia nucleotídica que contiene al menos una secuencia de recombinación adicional y la realización de la 1 recombinación específica de secuencia por una integrasa Int del bacteriófago lambda.
[2] La manipulación controlada de genomas eucarióticos y la expresión de proteínas recombinantes a partir de vectores episómicos son procedimientos importantes para analizar las funciones de genes específicos en organismos vivos. Además, tales manipulaciones tienen importancia en los procedimientos de terapia genética en 15 medicina. En este contexto, son de importancia particular la generación de animales transgénicos, el cambio de genes o los segmentos de gen (denominado «direccionamiento genético») y la integración dirigida de ADN exógeno en el genoma de eucariotas superiores. Recientemente, estas tecnologías han podido mejorarse por medio de caracterización y aplicación de sistemas de recombinación específica de secuencia.
[3] Además, la integración específica de secuencia de casetes (secuencias cortas) de expresión, que codifican y expresan un polipéptido/producto deseado en el genoma de células anfitrionas pertinentes biotecnológicas, también adquiere más significación para la producción de sustancias biofarmacéuticas. El nivel de expresión para un polipéptido deseado en una estirpe celular transformada estable depende del sitio de integración. Mediante integración específica de secuencia, es posible utilizar preferentemente una alta actividad de transcripción. 25 El procedimiento convencional para generar estirpes celulares de producción que expresan un polipéptido/producto deseado se basa en la integración aleatoria del vector de expresión recombinante en el genoma de la célula anfitriona. Las variaciones en el nivel de expresión de los genes integrados de interés en estirpes celulares transformadas estables se atribuye principalmente a diferencias en las ubicaciones cromosómicas y el número de copias. La integración aleatoria en la proximidad de la heterocromatina tiene como resultado niveles variables de 3 expresión del transgén. Se cree que las ubicaciones del cromosoma que promueve la expresión de uno o varios de
los genes integrados de interés son regiones transcripcionalmente activas de eucromatina. Esta condición aleatoria
de la integración provoca una gran diversidad en la robustez, productividad y calidad de las células recombinantes, lo que obliga a realizar procesos elaborados de detección y separación sistemáticos (cribado) para identificar y aislar un clon celular adecuado que exprese el polipéptido deseado en gran cantidad. Además, la heterogeneidad también 35 significa que para cada clon debe desarrollarse un procedimiento de producción optimizado, lo que vuelve el
desarrollo de una estirpe celular de producción adecuada un proceso que consume tiempo, que requiere trabajo y
que es costoso.
[4] Las ADN recombinasas específicas de secuencia conservadoras se han dividido en dos familias. Los 4 miembros de la primera familia, la denominada familia de «¡ntegrasas», catalizan la separación y la unión nuevamente de cadenas de ADN entre dos secuencias nucleotídicas definidas, las cuales se denominarán en lo sucesivo secuencias de recombinación. Las secuencias de recombinación pueden estar en dos moléculas de ADN diferentes o sobre una molécula de ADN, lo que se traduce en recombinación intermolecular e intramolecular, respectivamente. Para recombinación intramolecular, el resultado de la reacción depende de la orientación 45 respectiva de las secuencias de recombinación entre sí. En el caso de una orientación invertida, es decir opuesta, de las secuencias de recombinación, se produce inversión de los segmentos de ADN que se encuentran entre las secuencias de recombinación. En el caso de secuencias repetidas directas, es decir, repeticiones en tándem de las secuencias de recombinación sobre un ADN sustrato, se produce la supresión. En el caso de la recombinación intermolecular, es decir, ambas secuencias de recombinación se localizan en dos moléculas de ADN diferentes, se 5 puede producir la fusión de las dos moléculas de ADN. Aunque los miembros de la familia de las integrasas habitualmente catalizan la recombinación tanto intramolecular como intermolecular, las recombinasas de la segunda familia de las denominadas «invertasas/resolvasas» son únicamente capaces de catalizar la recombinación intramolecular.
[5] Actualmente, las recombinasas que se utilizan para la manipulación de genomas eucarióticos pertenecen a la familia de las integrasas. Tales recombinasas son las recombinasas Cre del bacteriófago P1 y la recombinasa Flp de levadura (Müller, U. (1999) Mech. Develop., 82, pp. 3). Las secuencias de recombinación a las cuales se une la recombinasa Cre se denominan loxP. LoxP es una secuencia nucleotídica de 34 pares de base de longitud que consiste en dos secuencias nucleotídicas invertidas de 13 pares de bases de longitud y un separador
de 8 pares de bases de longitud que se encuentra entre las secuencias Invertidas (Hoess, R. y cois. (1985) J. Mol. Biol., 181, pp. 351). Las secuencias de unión denominadas FRT para Flp se construyen de manera similar. No obstante, difieren de loxP (Kilby, J. y cois. (1993) Trends Gene!, 9, pp. 413). Por lo tanto, las secuencias de recombinación no se pueden sustituir entre sí, es decir, Cre no es capaz de recombinar secuencias FRT y FLP no es 5 capaz de recombinar secuencias loxP. Ambos sistemas de recombinación son activos sobre distancias grandes, es decir, el segmento de ADN que se va a invertir o suprimir y flanqueado por dos secuencias loxP o FRT puede ser de varias 1 pares de base de longitud.
[6] Por ejemplo, una recombinación específica de tejido en un sistema de ratón, un desplazamiento
cromosómico en plantas y animales y una inducción controlada de la expresión del gen se obtuvieron con los dos
sistemas; revísese el artículo de Müller, U. (1999) Mech. Develop., 82, pp. 3. La ADN polimerasa p se suprimió en tejidos particulares en ratones de esta manera; Gu, H, y cois. (1994) Science, 265, pp. 13. Un ejemplo adicional es la activación específica del oncogén del ADN del virus de tumor SV4 en los cristalinos de ratón, que conduce a la formación de tumor exclusivamente en esos tejidos. Asimismo, se ha utilizado la estrategia Cre-loxP en relación con 15 promotores inducibles. Por ejemplo, la expresión de la recombinasa se reguló con un promotor inducible por interferón, lo que condujo a la supresión de un gen específico en el hígado y no (o únicamente en un bajo grado) en otros tejidos; Kühn, R. y cois. (1995) Science, 269, pp.1427.
[7] Flasta ahora se han utilizado tres miembros de la familia invertasa/resolvasa para la manipulación de
genomas eucarióticos. Un mutante del bacteriófago Mu invertasa Gin puede catalizar la inversión de un fragmento
de ADN en protoplastos vegetales sin cofactores. No obstante, se ha descubierto que este mutante es
hiperrecombinogénico, es decir, cataliza las separaciones de la cadena de ADN también en otras secuencias diferentes a las secuencias de recombinación natural. Esto lleva a sucesos de recombinación parcialmente mortales no deseados en los genomas de protoplasto de planta. La recombinasa p de Streptococcus pyogenes cataliza la 25 recombinación en cultivos de célula de ratón entre dos secuencias de recombinación como secuencias repetidas directas, lo que conduce a la escisión del segmento. No obstante, simultáneamente con la supresión, también se ha detectado la inversión, lo que hace que el uso controlado del sistema para la manipulación de genomas eucarióticos resulte inadecuado. Se ha demostrado que los mutantes de la resolvasa y5 de £. coli son activos en secuencias de recombinación genómicas episómicas e introducidas artificialmente, pero la eficacia de esta última reacción aún es 3 bastante baja.
[8] La manipulación de genomas eucarióticos con la recombinasa Cre y Flp, respectivamente, muestra desventajas significativas. En caso de supresión, es decir, la recombinación de dos secuencias de recombinación loxP o FRT repetidas en tándem en un genoma, existe una pérdida irreversible del segmento de ADN que se
... [Seguir leyendo]
Reivindicaciones:
de interés que codifica para uno o más en el que la célula eucariota no es un
ID n.°: 13, una affP de SEQ ID n.° 14, una affL de SEQ ID n.° 15 o una affR de SEQ ID n.° 16 o un derivado de la misma en el interior de una célula;
b) seleccionar subclones candidatos en un biorreactor a pequeña escala que imita un proceso de producción de alimentación por lotes basado en parámetros tales como una alta viabilidad a alta densidad celular, el metabolismo y la capacidad de permitir una alta expresión y un rendimiento reproducible durante la producción,
c) introducir un segundo ADN que comprende una secuencia affB de SEQ ID n.°: 13, affP de SEQ ID n.°: 14, affL de 15 SEQ ID n.°: 15 o affR de SEQ ID n.°: 16 o un derivado de la misma, y al menos un gen de interés que codifica para
una proteína terapéutica en la célula del paso b);
d) poner en contacto dicha célula con una integrasa Int de bacteriófago lambda\
e) realizar la recombinación específica de secuencia por una integrasa Int de bacteriófago lambda, en donde el segundo ADN se integra dentro del primer ADN;
f) cultivar dicha célula en condiciones en las que se expresa(n) el(los) gen(es) de interés, en las que dicha célula se cultiva en medio libre de suero y en cultivo en suspensión; y
g) aislar dicho producto(s)/polipéptido(s) terapéutico(s) deseado(s) de la célula anfitriona o del medio de cultivo celular, en donde dicho derivado se define como una secuencia affB, affP, affL y affR que tiene una, dos, tres, cuatro, cinco, seis o siete sustituciones en la región de superposición y/o en la región de núcleo, o al menos un sitio de unión Int a núcleo de affB, affP, affL o affR, o al menos un sitio de unión Int a núcleo de affP, affL o affR más una
o más copias de los sitios de unión a brazo para Int, o al menos un sitio de unión Int a núcleo de affP, affL o affR más una o más copias de los sitios de unión de factor IHF, FIS o XIS, o combinaciones de los mismos.
2. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 1, en el que dicha primera secuencia de ADN comprende una secuencia affB o un derivado de la misma, dicha segunda secuencia comprende
una secuencia affB, affL o affR o un derivado de la misma, o en el que si dicha primera secuencia de ADN comprende una secuencia affP o un derivado de la misma, dicha segunda secuencia comprende una secuencia affP, affL o affR o un derivado de la misma, o en el que si dicha primera secuencia de ADN comprende una secuencia affL o un derivado de la misma, dicha segunda secuencia comprende una secuencia affB, affP o affL o un derivado de la misma, o en el que si dicha primera secuencia de ADN comprende una secuencia affR o un derivado 4 de la misma, dicha segunda secuencia comprende una secuencia affB, affP o affR o un derivado de la misma, y en el que dicho derivado se define como una secuencia affB, affP, affL y affR que tiene una, dos, tres, cuatro, cinco, seis o siete sustituciones en la región de superposición y/o en la región de núcleo, o al menos un sitio de unión Int a núcleo de affB, affP, affL o affR, o al menos un sitio de unión Int a núcleo de affP, affL o affR más una o más copias de los sitios de unión a brazo para Int, o al menos un sitio de unión Int a núcleo de affP, affL o affR más una o más 45 copias de los sitios de unión de factor IHF, FIS o XIS, o combinaciones de los mismos.
3. Un procedimiento in vitro o ex vivo de acuerdo con las reivindicaciones 1 ó 2, en el que el primer ADN se ha integrado en el genoma, un minicromosoma artificial o un elemento episómico de la célula anfitriona, antes de que se introduzca dicho segundo ADN en dicha célula.
4. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 3, en el que el primer ADN se ha integrado en el genoma de la célula anfitriona.
5. Un procedimiento in vitro o ex vivo de acuerdo con una cualquiera de las reivindicaciones anteriores, 55 en el que dicha recombinación específica de secuencia se realiza por Int y uno o más cofactores seleccionados
entre XIS, FIS y/o IHF.
6. Un procedimiento in vitro o ex vivo e acuerdo con una cualquiera de las reivindicaciones anteriores, en el que la recombinación específica de secuencia se realiza por una Int modificada, que tiene al menos el 9 % de la
1. Procedimiento in vitro o ex vivo para expresar al menos un gen
producto(s)/polipéptido(s) terapéutico(s) deseado(s) en una célula eucariota, 5 ovocito humano ni una célula madre embrionaria humana, que comprende
a) introducir de un primer ADN que comprende una secuencia affB de SEQ
actividad de la Int natural.
7. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 6, en el que la Int modificada es Int-h o lnt-h/218.
8. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 6 ó 7, en el que Int, Int-h o lnt- h/218, XIS, FIS y/o IHF se añaden a la célula en forma purificada o se coexpresan por dicha célula anfitriona, en donde se realiza la recombinación específica de secuencia.
9. Un procedimiento in vitro o ex vivo de acuerdo con una cualquiera de las reivindicaciones anteriores,
en el que adlclonalmente se introducen en la célula una tercera o una tercera y cuarta secuencias de ADN que comprenden un gen para Int, o un gen para Int y uno o más genes cofactores seleccionados entre el gen para XIS, el gen para FIS y/o el gen para IHF, respectivamente.
1. Un procedimiento in vitro o ex vivo de acuerdo con una cualquiera de las reivindicaciones anteriores,
en el que no se requiere XIS, FIS ni IHF cuando se realiza la recombinación específica de secuencia por una Int modificada, que tiene al menos el 9 % de la actividad de la Int natural.
11. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 1, en el que la Int modificada es 2 Int-h o lnt-h/218.
12. Un procedimiento in vitro o ex vivo de acuerdo con una cualquiera de las reivindicaciones anteriores, en el que la primera y/o la segunda secuencia de recombinación comprende además un ácido nucleico que codifica para un polipéptido terapéutico de interés.
13. Un procedimiento in vitro o ex vivo de acuerdo con una cualquiera de las reivindicaciones anteriores, en el que dicho polipéptido terapéutico de Interés es un anticuerpo, hormona o factor de crecimiento.
14. Un procedimiento in vitro o ex vivo de acuerdo con una cualquiera de las reivindicaciones anteriores, 3 en el que la célula anfitriona es una célula de mamífero.
15. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 14, en el que la célula de mamífero es una célula de roedor.
16. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 15, en el que la célula de
mamífero es una célula de ratón o de hámster.
17. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 16, en el que la célula de hámster es una célula BHK o CHO y la célula de ratón es una célula de mieloma murino.
18. Un procedimiento in vitro o ex vivo de acuerdo con la reivindicación 17, en el que la célula de mieloma murino es una célula NS o Sp2/.
Patentes similares o relacionadas:
 Animales no humanos que tienen un locus de cadena ligera lambda de inmunoglobulina modificado por ingeniería, del 29 de Julio de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor cuyo genoma de la línea germinal comprende un locus de cadena ligera λ de inmunoglobulina endógeno que comprende:
(a) uno o más segmentos […]
Animales no humanos que tienen un locus de cadena ligera lambda de inmunoglobulina modificado por ingeniería, del 29 de Julio de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor cuyo genoma de la línea germinal comprende un locus de cadena ligera λ de inmunoglobulina endógeno que comprende:
(a) uno o más segmentos […]
Métodos y composiciones para el tratamiento de enfermedades por almacenamiento lisosomal, del 27 de Mayo de 2020, de Sangamo Therapeutics, Inc: Uno o más transgenes y una o más nucleasas con dedos de zinc (ZFN) para su uso en un método de tratamiento de una enfermedad por almacenamiento lisosomal, el método […]
Terapias de aumento génico de la degeneración retiniana causada por mutaciones en el gen PRPF31, del 6 de Mayo de 2020, de MASSACHUSETTS EYE & EAR INFIRMARY: Un vector de virus adeno-asociado de tipo 2 (AAV2) que comprende una secuencia que codifica PRPF31 humano, operativamente enlazado a un promotor […]
Proteínas que tienen actividad nucleasa, proteínas de fusión y usos de estas, del 18 de Marzo de 2020, de HELMHOLTZ ZENTRUM MUNCHEN DEUTSCHES FORSCHUNGSZENTRUM FUR GESUNDHEIT UND UMWELT (GMBH): Una molécula de ácido nucleico que codifica (I) un polipéptido que tiene la actividad de una endonucleasa, que es (a) una molécula de ácido nucleico que […]
Animales no humanos que tienen una interrupción en un locus C9ORF72, del 19 de Febrero de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor que comprende en su genoma una deleción de la porción codificante del exón 2 hasta la porción codificante del exón 11 de un locus C9orf72 endógeno, […]
Métodos y composiciones para modificar un locus objetivo, del 12 de Febrero de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para la modificación en serie de un locus objetivo en una célula, que comprende: (a) proporcionar la célula que comprende el locus objetivo, en donde el locus objetivo […]
Modelo de cerdo para la diabetes, del 29 de Enero de 2020, de AARHUS UNIVERSITET: Un cerdo transgénico que comprende un gen de polipéptido amiloide de los islotes (IAPP) mutado humano o parte del mismo, y que muestra al menos […]
Procedimientos de modificación de células hospedadoras, del 22 de Enero de 2020, de GLAXOSMITHKLINE BIOLOGICALS S.A.: Una célula hospedadora procariota aislada, en la que i) un gen heterólogo que codifica una oligosacaril transferasa y ii) ADN de al menos 8 kb que codifica […]