Genotipificación de loci de HLA.
Un método de genotipificación en un locus de HLA utilizando un sistema de PCR múltiple que tiene una mezclamaestra de PCR uniforme y aplicando un perfil de termociclación uniforme,
cuyo método consiste en las siguientesetapas:
i) establecer una pluralidad de al menos 25 recipientes de reacción PCR con una solución acuosa cadauno que contiene un par de cebadores de PCR para amplificar una región control de ADN, un par decebadores de PCR específicos de alelos de HLA diferentes para amplificar diferentes alelos de HLA en ellocus y una mezcla maestra de PCR uniforme que tiene un colorante fluorescente capaz de detectar ADNde doble hebra;
ii) añadir una muestra biológica que contiene ADN codificante de HLA y ADN codificante control;
iii) amplificar el ADN codificante de HLA y el ADN codificante control en los recipientes de reacciónutilizando un perfil de termociclación uniforme en la pluralidad de recipientes de reacción para obtenersoluciones que contienen ADN amplificado que tiene un amplicón control y amplicones de HLA, donde latemperatura de fusión del amplicón control es al menos 10°C inferior a la temperatura de fusión de losamplicones de HLA; y
iv) determinar el tipo de HLA por el perfil de fusión del ADN en las soluciones.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2006/013462.
Solicitante: Linkage Biosciences, Inc.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 890 Dubuque Avenue South San Francisco, CA 94080 ESTADOS UNIDOS DE AMERICA.
Inventor/es: ANTOVICH,ZACHARY.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07H21/02 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con ribosilo como radical sacárido.
- C07H21/04 C07H 21/00 […] › con desoxirribosilo como radical sacárido.
- C12Q1/68 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2394097_T3.pdf
Fragmento de la descripción:
Genotipificación de loci de HLA
Campo de la invención Métodos para detectar secuencias de nucleótidos y genotipificar loci complejos.
Antecedentes de la invención El complejo mayor de histocompatibilidad (MHC) de humanos es un grupo de genes localizados en el sexto cromosoma. Los genes del Antígeno Leucocitario Humano (HLA) se localizan en este complejo y codifican proteínas que presentan antígeno de la superficie celular. Estas proteínas tienen un importante papel en la capacidad del sistema inmune para reconocer "lo propio" frente a "lo ajeno" y son un factor de gran importancia que influye en la inmunidad, la autoinmunidad, la enfermedad genética y el trasplante de tejidos. Las proteínas HLA se dividen en tres clases: Clase I (HLA A, B, C, etc.) , Clase II (HLA DR, DQ, DP, etc.) y Clase III, que incluyen otros componentes, tales como proteínas del complemento y citoquinas. Los genes de las Clases I y II son altamente polimórficos, lo que da como resultado una tremenda diversidad genética entre la población humana.
La tipificación del HLA es un procedimiento mediante el cual se identifica una proteína o genotipo HLA de un individuo. Habitualmente se realiza la tipificación del HLA antes del trasplante de órganos sólidos. La compatibilidad del HLA de un paciente y un donante puede reducir el rechazo inmunológico y mejorar los resultados de supervivencia (Takemoto et al., Twelve years’ experience with national sharing of HLA-matched cadaveric kidneys for transplantation, The New England Journal of Medicine, 12 de Oct. de 2000, 343 (15) : 1078-84) . También se usa la tipificación del HLA en una amplia variedad de otras aplicaciones, incluyendo las pruebas de paternidad, el análisis forense, las pruebas de enfermedades genéticas y la investigación biomédica general.
Históricamente, la tipificación del HLA era realizada utilizando técnicas serológicas; sin embargo, estas técnicas no pueden diferenciar entre muchos de los alelos que se sabe existen en la población. Los métodos de genotipificación basados en ADN más recientes son más informativos y están encontrando una amplia aceptación. Existen tres métodos comunes de genotipificación del HLA: (1) Secuenciación directa del ADN, que requiere una etapa de amplificación por PCR y procedimientos caros de secuenciación; este método es utilizado para descubrir nuevos alelos y cuando se requiere una tipificación de alta resolución. (2) PCR con Sonda Oligonucleotídica Específica de Secuencia (SSOP-PCR) , que requiere una etapa de amplificación por PCR, una etapa de inmovilización, una etapa de detección con sonda y una etapa de análisis. (3) PCR con Cebado Específico de Secuencia (SSP-PCR) , que requiere una etapa de amplificación por PCR, una etapa de electroforesis en gel y una etapa de análisis. Downing et al., 2004, 8 (3) : 301-312, describen la tipificación de HLA de cinco loci de voluntarios donantes de células madre hematopoyéticas utilizando SSP-PCR. Tiemann et al., 2002, 47: 131-134, describen la tipificación rápida del ADN del alelo HLA-B27 por PCR en tiempo real utilizando tecnología LightCycler. Slateva et al., 2002, Tissue Antigens, 59 (2) , suplemento 13, describe la fluorotipificación de HLA-DRB basada en curvas de fusión mediadas por SYBR Green.
Los tres métodos consumen tiempo y son realizados por tecnólogos médicos altamente entrenados. La secuenciación directa del HLA es innecesaria y demasiado cara para la mayoría de las necesidades de tipificación. La SSOP-PCR requiere mucha mano de obra a menos que se emplee maquinaria cara para automatizar los procedimientos. La SSP-PCR también requiere mucha mano de obra, pero puede ser el método más simple de llevar a cabo. Con la SSP-PCR, se utiliza el poder de discriminación de la PCR para amplificar alelos o grupos de alelos específicos. Se diseñan cebadores oligonucleotídicos para solaparse con regiones polimórficas. Los cebadores que se hibridan por completo con estas regiones tienen una temperatura de fusión (Tf) mayor que los pares mal emparejados y facilitan la amplificación. Los cebadores mal emparejados no consiguen amplificar. La genotipificación de un locus de HLA mediante SSP-PCR requiere múltiples reacciones PCR, suficientes para definir cada alelo o grupo de alelos. Por ejemplo, el gen HLA-A solo tiene 262 alelos y aproximadamente 25 familias de alelos, según definen Schreuder, G. et al., The HLA Dictionar y 2004: a summar y of HLA-A, - B, -C, -DRB 1/3/4/5 and -DQBI alleles and their association with serologically defined HLA-A, -B, -Q, -DR and -DQ antigens, Tissue Antigens 2005, 65: 1-55. Véase también Krausa y Browning, A comprehensive PCR-SSP typing system for identification of HLA-A locus alleles, Tissue Antigens, 1996, 47: 237-144.
La tecnología SSP-PCR es comúnmente utilizada para realizar la genotipificación de HLA de cadáveres donantes de órganos. Frecuentemente, este trabajo se realiza tarde por la noche, cuando, lamentablemente, llegan muchas víctimas de accidentes a los hospitales o a los depósitos de cadáveres. Se recogen los órganos donados y se guardan en hielo a la espera de trasplante. Mientras tanto, se envían muestras de sangre de los cadáveres donantes a los laboratorios de diagnóstico para realizar la tipificación del HLA. El tiempo de isquemia de los órganos es inversamente proporcional al éxito en el trasplante, por lo que resulta crítico un tiempo rápido de entrega de la tipificación del HLA. Cuanto antes se pueda trasplantar el órgano, mejor. La tipificación del HLA es con frecuencia la variable limitante del tiempo en este proceso. El análisis posterior a la ejecución después de la SSP-PCR es un proceso largo que requiere electroforesis en gel, documentación de datos y evaluación de datos. Existe, por lo tanto, una necesidad en la técnica de una versión mejorada de la genotipificación por SSP-PCR, mediante la cual se puedan obtener resultados más rápidamente, con menos etapas y de un modo más automatizado.
Utilizando la invención aquí descrita, la identificación del HLA se produce en una sola etapa. Se añaden muestras de ensayo de ADN a recipientes de reacción llenados previamente y se cargan en un ciclador/lector térmico. No se necesitan pipeteado, manipulación, lectura o análisis adicionales. Sólo cargar la muestra, proceder e informar.
Resumen de la invención La invención describe un nuevo método de genotipificación en un locus de HLA utilizando un sistema de PCR múltiple que tiene una mezcla maestra de PCR uniforme y aplicando un perfil de termociclación uniforme, cuyo método consiste en las siguientes etapas: (i) establecer una pluralidad de al menos 25 recipientes de reacción PCR, cada uno con una solución acuosa que contiene un par de cebadores de PCR control para amplificar una región control de ADN, un par de cebadores de PCR específicos para alelos de HLA diferentes para amplificar diferentes alelos de HLA en el locus y una mezcla maestra de PCR uniforme que tiene un colorante fluorescente capaz de unirse selectivamente a ADN de doble hebra; (ii) añadir una muestra biológica que contiene ADN codificante de HLA y ADN codificante control; (iii) amplificar el ADN codificante de HLA y el ADN codificante control en los recipientes de reacción utilizando un perfil de termociclación uniforme en la pluralidad de recipientes de reacción para obtener soluciones que contienen ADN amplificado que tiene un amplicón control y amplicón de HLA y amplicones de HLA, donde la temperatura de fusión del amplicón control es al menos 10°C inferior a la temperatura de fusión de los amplicones de HLA; y (iv) determinar el tipo de HLA por el perfil de fusión de ADN en las soluciones. La invención también describe un sistema de genotipificación en un locus de HLA utilizando un método de la invención, donde el sistema comprende:
i) un termociclador que consta de una pluralidad de al menos 25 recipientes de reacción PCR, donde cada recipiente de reacción contiene un par de cebadores de PCR capaces de amplificar una región control de ADN, un par diferente de cebadores de PCR específicos para alelos de HLA para amplificar diferentes alelos de HLA en el locus de HLA y una mezcla maestra de PCR uniforme que tiene un colorante fluorescente capaz de detectar ADN de doble hebra, donde la temperatura de fusión del amplicón control amplificado por el par de cebadores control es al menos 10°C inferior a la temperatura de fusión del amplicón de HLA amplificado por el par de cebadores específicos de alelos; y ii) un ordenador conectado de forma operativa a los recipientes de reacción, donde el ordenador es capaz de determinar el genotipo de HLA comparando los perfiles de fusión de las soluciones con los de soluciones estándar.
Breve descripción de los dibujos... [Seguir leyendo]
Reivindicaciones:
1. Un método de genotipificación en un locus de HLA utilizando un sistema de PCR múltiple que tiene una mezcla maestra de PCR uniforme y aplicando un perfil de termociclación uniforme, cuyo método consiste en las siguientes etapas:
i) establecer una pluralidad de al menos 25 recipientes de reacción PCR con una solución acuosa cada uno que contiene un par de cebadores de PCR para amplificar una región control de ADN, un par de cebadores de PCR específicos de alelos de HLA diferentes para amplificar diferentes alelos de HLA en el locus y una mezcla maestra de PCR uniforme que tiene un colorante fluorescente capaz de detectar ADN de doble hebra; ii) añadir una muestra biológica que contiene ADN codificante de HLA y ADN codificante control; iii) amplificar el ADN codificante de HLA y el ADN codificante control en los recipientes de reacción utilizando un perfil de termociclación uniforme en la pluralidad de recipientes de reacción para obtener soluciones que contienen ADN amplificado que tiene un amplicón control y amplicones de HLA, donde la temperatura de fusión del amplicón control es al menos 10°C inferior a la temperatura de fusión de los amplicones de HLA; y iv) determinar el tipo de HLA por el perfil de fusión del ADN en las soluciones.
2. Un método de la reivindicación 1, donde el marcaje fluorescente se une al surco menor del ADN de doble hebra amplificado.
3. Un método de la reivindicación 1, donde el amplicón control tiene una Tf por debajo de 75°C.
4. Un método de la reivindicación 1, donde el número de recipientes de reacción es suficiente para identificar suficientes polimorfismos como para definir una familia de alelos o un solo alelo para un locus de HLA dado.
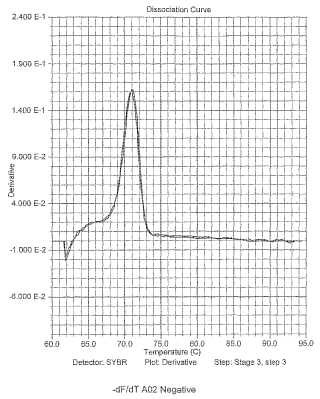
5. Un método de la reivindicación 1, donde la etapa determinante incluye el cálculo de la primera derivada del perfil de fusión fluorescente de las soluciones.
6. Un método de la reivindicación 1, donde el método comprende la etapa de utilización de un ordenador para calcular la primera derivada a partir de los datos sin procesar y de aporte de resultados gráficos.
7. Un método de la reivindicación 1, donde la muestra biológica procede de tejido cadavérico.
8. Un método de la reivindicación 1, donde el número de recipientes de reacción de la PCR es mayor de 35.
9. Un método de la reivindicación 1, donde el locus es seleccionado entre el grupo consistente en HLA A, HLA B, HLA C, HLA DR, HLA DP y HLA DQ.
10. Un sistema de genotipificación en un locus de HLA utilizando un método de la reivindicación 1, donde el sistema comprende:
i) un termociclador consistente en una pluralidad de al menos 25 recipientes de reacción de PCR, donde cada recipiente de reacción contiene un par de cebadores de PCR capaz de amplificar una región control de ADN, un par de cebadores de PCR específicos de alelos de HLA para amplificar diferentes alelos de HLA en el locus de HLA y una mezcla maestra de PCR uniforme que tiene un colorante fluorescente capaz de detectar ADN de doble hebra, donde la temperatura de fusión del amplicón control amplificado por el par de cebadores control es al menos 10°C inferior a la temperatura de fusión del amplicón de HLA amplificado por el par de cebadores específicos de alelos; y ii) un ordenador operativamente conectado a los recipientes de reacción, donde el ordenador es capaz de determinar el genotipo de HLA comparando los perfiles de fusión de las soluciones con los de soluciones estándar.
11. Un sistema de la reivindicación 10, donde el colorante fluorescente se une al surco menor del ADN de doble hebra amplificado.
12. Un sistema de la reivindicación 10, donde el ordenador determina el genotipo de HLA calculando la primera derivada del perfil de fusión fluorescente de las soluciones.
13. Un sistema de la reivindicación 10, donde el amplicón control tiene una Tf por debajo de 75°C.
14. Un sistema de la reivindicación 10, donde el número de recipientes de reacción es suficiente para identificar
suficientes polimorfismos como para definir una familia de alelos o un solo alelo para un locus de HLA dado.
15. Un sistema de la reivindicación 10, donde el ordenador calcula la primera derivada de los perfiles de fusión y
proporciona resultados gráficos. 5
16. Un sistema de la reivindicación 10, donde el número de recipientes de reacción de la PCR es mayor de 35.
17. Un sistema de la reivindicación 10, donde el locus es seleccionado entre el grupo consistente en HLA A, HLA B,
HLA C, HLA DR, HLA DP y HLA DQ. 10
18. Un sistema de la reivindicación 10, donde el ordenador proporciona un perfil de termociclación uniforme en la pluralidad de recipientes de reacción.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]