Detección no invasiva del cáncer de vejiga mediante hibridación fluorescente in situ de Aurora A.
Un método para la detección de cáncer de vejiga en un paciente que comprende:
aislar células de la vejigade la orina del paciente, hibridar las células de la vejiga con una sonda de ADN marcada, en donde lasonda reconoce al menos una porción del gen de la quinasa Aurora A, para obtener una muestra de célulasde la vejiga hibridadas con la sonda de ADN marcada; y contar el número de células de la vejiga presentesen la muestra, el número de copias del gen de la quinasa Aurora A en cada célula de la vejiga de lamuestra y el número de células de la vejiga en la muestra que tienen al menos un primer número umbral decopias del gen de la quinasa Aurora A, comprendiendo además, si un porcentaje mayor o igual a un primerporcentaje umbral de las células de la vejiga tiene al menos el primer número umbral de copias del gen dela quinasa Aurora A, suponer que el paciente tiene cáncer de vejiga, en donde el primer número umbral decopias del gen de la quinasa Aurora A es tres y el primer porcentaje umbral es 15%.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2008/013632.
Solicitante: BOARD OF REGENTS THE UNIVERSITY OF TEXAS SYSTEM.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 201 WEST 7TH STREET AUSTIN, TX 78701 ESTADOS UNIDOS DE AMERICA.
Inventor/es: GROSSMAN,H. BARTON, CZERNIAK,BOGDAN, SUBRATA,SEN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2407630_T3.pdf
Fragmento de la descripción:
Detección no invasiva del cáncer de vejiga mediante hibridación fluorescente in situ de Aurora A
Antecedentes de la invención Esta invención se hizo con el respaldo del gobierno de los Estados Unidos bajo la subvención número U01 CA 85078 concedida por el National Cancer Institute. El gobierno de los Estados Unidos tiene determinados derechos en la invención.
La presente invención se refiere, en líneas generales, al campo de la detección del cáncer. Más concretamente, se refiere a la detección del cáncer de vejiga.
Resumen de la invención La presente invención, según se define en la reivindicación 1, se refiere a un método para la detección de cáncer de vejiga en un paciente que consiste en aislar células de la vejiga de la orina del paciente, hibridar las células de la vejiga con una sonda de ADN marcada, en donde la sonda reconoce al menos una porción del gen de la quinasa Aurora A, para obtener una muestra de células de la vejiga hibridadas con la sonda de ADN marcada; y contar el número de células de la vejiga presentes en la muestra , el número de copias del gen de la quinasa Aurora A en cada célula de la vejiga de la muestra y el número de células de la vejiga presentes en la muestra que tienen al menos un primer número umbral de copias del gen de la quinasa Aurora A, consistiendo, además, en que si los números superiores o iguales a un primer porcentaje umbral de las células de la vejiga tienen el primer número umbral de copias del gen de la quinasa Aurora A, suponer que el paciente tiene cáncer de vejiga, en donde el primer número umbral de copias del gen de la quinasa Aurora A es tres y el primer porcentaje umbral es de un 15%.
Breve descripción de los dibujos Los dibujos siguientes forman parte de la presente especificación y se incluyen para demostrar aún más algunos aspectos de la presente invención. La invención puede comprenderse mejor haciendo referencia a uno o más de estos dibujos en combinación con la descripción detallada de las realizaciones específicas aquí presentadas.
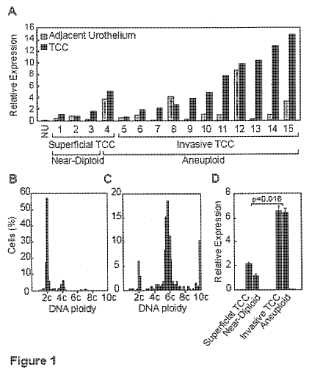
Figura 1. Expresión de la quinasa Aurora A en carcinomas de células transicionales (TCC) y su relación con la ploidía del ADN. A, Niveles de quinasa Aurora A en muestras pareadas de urotelio y TCC adyacentes revelados por una RT-PCR cuantitativa. B y C. Histogramas de ADN generados mediante análisis de imágenes de núcleos tumorales en los que puede verse un contenido de ADN casi diploide (2c) y una aneuploidía pronunciada (6c) , respectivamente. D, Niveles medios de expresión de la quinasa Aurora A en TCCs de grado bajo (grado 1-2) , superficial (Ta-T1a) y grado alto (grado 3) , invasivo (T1b y superior) además de TCCs casi diploides y aneuploides.
Figura 2. Expresión de la quinasa Aurora A y número de copias cromosómicas en líneas cancerosas de la vejiga. A, Expresión de la quinasa Aurora A en líneas celulares cancerosas de la vejiga mediante una RT-PCR cuantitativa en comparación con células uroteliales cultivadas (NU204) . B, Número de copias cromosómicas revelado por una FISH cuantitativa utilizando sondas centroméricas a los cromosomas 3, 7 y 17. C, Valores medios de expresión de la quinasa Aurora A en tres grupos de líneas celulares cancerosas de la vejiga. D, Número medio de copias cromosómicas en líneas celulares cancerosas revelado por una FISH con sondas centroméricas a los cromosomas 3, 7 y 17 en 3 grupos de líneas celulares cancerosas de la vejiga mostradas en C.
Figura 3. Expresión ectópica de la proteína de fusión quinasa Aurora A-GFP en células uroteliales SV40 transfectadas con un vector adenoviral que contiene un inserto de quinasa Aurora A de tipo salvaje utilizando un anticuerpo anti-GFP. A, Análisis Western blot en el que se muestra la expresión de la proteína de fusión quinasa Aurora A-GFP (panel superior) . Análisis de inmunofluorescencia que muestra la expresión ectópica de la proteína de fusión quinasa Aurora A-GFP con DAPI como contracolorante nuclear (panel inferior) . Imágenes de inmunofluorescencia de células infectadas con un vector adenoviral sin un inserto de quinasa Aurora A (a, b) . Imagen obtenida con un filtro rojo que revela dos centrosomas (a) . Imagen obtenida con un filtro verde que revela una expresión no ectópica de una proteína de fusión quinasa Aurora A-GFP (b) . Imágenes de inmunofluorescencia de células infectadas con un vector adenoviral que contiene un inserto de quinasa Aurora A (c, d) . Imagen obtenida con un filtro rojo que muestra una multiplicación de centrosomas revelada por un anticuerpo anti-tubilina-y (rojo) (c) . Imagen obtenida con un filtro verde que muestra la expresión de una proteína de fusión quinasa Aurora A-GFP expresada ectópicamente en centrosomas (d) . B, Evaluación cuantitativa del número de copias de centrosomas y cromosómicas inducida por una quinasa Aurora A expresada ectópicamente. C, Análisis FACS por fluorescencia doble de una proteína de quinasa Aurora A-GFP expresada ectópicamente (fluorescencia verde) en relación con el contenido de ADN revelado con un contracolorante de PI (fluorescencia roja) . Diagrama de dispersión de fluorescencia doble tras la infección con un vector adenoviral que contiene un inserto de quinasa Aurora A de tipo salvaje (panel derecho) . Diagrama de dispersión de control de células infectadas con un vector adenoviral vacío (panel derecho) . Nótese el aumento de células aneuploides en las zonas trazadas del diagrama de dispersión designadas como A y B (panel derecho) en comparación con el control (panel izquierdo) . D, Evaluación cuantitativa de las células aneuploides revelada por el análisis FACS por fluorescencia doble mostrada C y formación decolonias en agar blando. Control/Ad: células infectadas con un vector adenoviral sin un inserto de quinasa Aurora A, GFP Ad/quinasa Aurora A: células transfectadas con un vector adenoviral que contiene una quinasa Aurora A de tipo salvaje, MOI: multiplicidad de infección.
Figura 4. Detección de células cancerosas en orina evacuada con una sonda de FISH de la quinasa Aurora A. A, una FISH por fluorescencia doble en la que se demuestra un número de copias del gen de la quinasa Aurora A (rojo) y un brazo corto de una sonda específica al cromosoma (verde) . Número diploide de copias de quinasa Aurora A en células uroteliales normales (a) . Bajo nivel de amplificación del gen de la quinasa Aurora A (3 copias) en un TCC de grado bajo (grado 1-2) (b) . Alto nivel de amplificación del gen de la quinasa Aurora A (>4 copias) en un TCC invasivo de grado alto (grado 3) (c) . Polisomía del cromosoma 20 en un TCC invasivo de grado alto (grado 3) que contiene múltiples copias de quinasa Aurora A y sondas del cromosoma 20p (d) . B, Análisis FISH cuantitativo del número de copias del gen de la quinasa Aurora A en especímenes de orina evacuada de 23 pacientes con TCC de la vejiga (set de formación) . Se muestran los porcentajes de células que muestran unos niveles de amplificación bajos (3-4 copias) y altos (> 4 copias) de quinasa Aurora A en los pacientes individuales. C, curva ROC para una prueba de FISH de la quinasa Aurora A en un set de formación que consiste en 23 muestras de orina de pacientes con cáncer de vejiga y siete muestras de orina de controles de pacientes sanos no afectados (n=30) . D, curva ROC para una prueba de FISH de la quinasa Aurora A en un grupo de análisis que consiste en 51 muestras de orina de pacientes con cáncer de vejiga y 30 muestras de orina de controles sanos no afectados (n=81) . E, puntuación de la quinasa Aurora A por grado histológico.
Figura 5. Análisis de FISH cuantitativa del número de copias del gen de la quinasa Aurora A en muestras de orina evacuada de 74 pacientes con TCC de la vejiga y 37 controles de pacientes sanos (sets de formación y de prueba combinados) . Se muestran los porcentajes de células que muestran unos niveles de amplificación bajos (3-4 copias) y altos (> 4 copias) de quinasa Aurora A en pacientes individuales.
Figura 6. Un mapa de la sonda de AURKA 20ql3.
Descripción detallada de la invención Las células malignas que manifiestan una inestabilidad cromosómica en forma de aneuploidía, a menudo presentan centrosomas supernumerarios y husos mitóticos multipolares que sugieren que las anormalidades en los orgánulos que controlan la segregación de cromosomas durante la mitosis desempeñan una función en el desarrollo de la aneuploidía.1-3
La quinasa Aurora A, también conocida como STK15 o BTAK, es un miembro de la familia de las serina-treonina quinasas que incluye aurora quinasa de la Drosophila y quinasa IPL1 de Saccharomyces cervisiae.4-6 Estas quinasas han sido identificadas como reguladores críticos de la segregación cromosómica normal y de las funciones de los centrosomas... [Seguir leyendo]
Reivindicaciones:
1. Un método para la detección de cáncer de vejiga en un paciente que comprende: aislar células de la vejiga de la orina del paciente, hibridar las células de la vejiga con una sonda de ADN marcada, en donde la sonda reconoce al menos una porción del gen de la quinasa Aurora A, para obtener una muestra de células de la vejiga hibridadas con la sonda de ADN marcada; y contar el número de células de la vejiga presentes en la muestra, el número de copias del gen de la quinasa Aurora A en cada célula de la vejiga de la muestra y el número de células de la vejiga en la muestra que tienen al menos un primer número umbral de copias del gen de la quinasa Aurora A, comprendiendo además, si un porcentaje mayor o igual a un primer porcentaje umbral de las células de la vejiga tiene al menos el primer número umbral de copias del gen de la quinasa Aurora A, suponer que el paciente tiene cáncer de vejiga, en donde el primer número umbral de copias del gen de la quinasa Aurora A es tres y el primer porcentaje umbral es 15%.
2. El método de la reivindicación 1, en donde el recuento comprende además contar el número de células de la vejiga que tiene al menos un segundo número umbral de copias del gen de la quinasa Aurora A, en donde el segundo número umbral de copias es mayor que el primer número umbral de copias.
3. El método de la reivindicación 2, en donde el segundo número umbral de copias del gen de la quinasa Aurora A es cinco.
4. El método de la reivindicación 1, que comprende además, si un porcentaje mayor que o igual a un segundo porcentaje umbral de las células de la vejiga tiene al menos el primer número umbral de copias del gen de la quinasa Aurora A, en donde el segundo porcentaje umbral es mayor que el primer porcentaje umbral,
suponer que el paciente tiene un cáncer de vejiga agresivo.
5. El método de la reivindicación 4, en donde el segundo porcentaje umbral es de un 20%.
6. El método de la reivindicación 1, en donde el gen de la quinasa Aurora A comprende una secuencia de ADN seleccionada del grupo que consiste en el SEQ ID NO: 1 y SEQ ID NO:2.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]