Sistema bihíbrido de levaduras/bacterias y procedimientos para su utilización.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2005/031141.
Solicitante: FOX CHASE CANCER CENTER.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 333 COTTMAN AVENUE PHILADELPHIA, PA 19111-2497 ESTADOS UNIDOS DE AMERICA.
Inventor/es: SEREBRIISKII,Ilya, GOLEMIS,Erica A, JOUNG,Keith J.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07H21/04 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con desoxirribosilo como radical sacárido.
- C12N15/63 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Introducción de material genético extraño utilizando vectores; Vectores; Utilización de huéspedes para ello; Regulación de la expresión.
PDF original: ES-2381384_T3.pdf
Fragmento de la descripción:
Sistema bihíbrido de levaduras/bacterias y procedimientos para su utilización
CAMPO DE LA INVENCIÓN
Esta invención está relacionada con el campo de la biología molecular. Más específicamente, la invención proporciona nuevas composiciones y procedimientos para facilitar el aislamiento y la caracterización de nuevas interacciones proteína-proteína involucradas en la regulación del crecimiento celular y el metabolismo.
CAMPO DE INVENCIÓN
A lo largo de esta solicitud se citan varias publicaciones y documentos de patente para definir mejor el estado de la técnica a la que pertenece esta invención. Cada una de las siguientes citas se incorpora en el presente documento por referencia.
Los sistemas bihíbridos de levaduras (Chien y col. 1991; Fields y Song 1989; Gyuris y col. 1993; Vojtek y col. 1993) son herramientas habituales utilizadas para identificar nuevas interacciones proteína-proteína y para realizar análisis de estructura-función sobre interacciones proteína-proteína previamente definidas. Dichos sistemas son efectivos con una parte sustancial de proteínas eucariotas y han desempeñado un papel importante en análisis proteómicos de alto rendimiento dirigidos a establecer grupos de proteínas que interaccionan (por ejemplo, Giot y col. 2003; lto y col. 2000; Li y col. 2004; Uetz y col., 2000) . Con el fin de incrementar la versatilidad de un enfoque bihíbrido para identificar y analizar interacciones de proteínas en aplicaciones de alto rendimiento, uno de los enfoques ha sido trasladar los componentes básicos del sistema bihíbrido de levaduras a un organismo hospedador bacteriano (Dove y col. 1997; Joung y col. 2000) . Hasta la fecha, no se ha comparado directamente la eficacia relativa de la detección de las interacciones entre proteínas en bacterias y levaduras. No obstante, existen una serie de razones para anticipar que es posible que se observen diferencias. Puesto que las levaduras son eucariotas, es más probable que las proteínas eucariotas utilizadas como "cebos" en selecciones de bihíbridos se plieguen y se modifiquen post-traduccionalmente de manera apropiada en levaduras que en bacterias, incrementando así sus oportunidades de identificar pares fisiológicos. No obstante, ciertas proteínas pueden ser problemáticas como cebos en sistemas bihíbridos de levaduras; por ejemplo, proteínas que normalmente se excluyen del núcleo en eucariotas, que se pueden secuestrar mediante la interacción con un par abundante evolutivamente conservado en levaduras, o que estimulan la trascripción en levaduras (es decir, que "se autoactivan") . Se espera que todos estos posibles problemas sean menos problemáticos en el sistema bihíbrido de bacterias. Para maximizar las oportunidades de obtener todos los interactores relevantes para una proteína de interés, sería deseable tener la capacidad de someter a ensayo rápidamente un cebo dado tanto en entornos de levaduras como de bacterias.
RESUMEN DE LA INVENCIÓN
Según la presente invención, se proporcionan plásmidos y cepas adecuados para su uso tanto en sistemas de interacción entre proteínas de levaduras como de bacterias. Se describen una nueva serie de vectores en los que un solo plásmido que contiene un promotor modificado dirige la expresión eficiente de una proteína cebo en levaduras o en bacterias, permitiendo así el estudio en paralelo en ambos organismos. Además, se proporcionan cepas de soporte informadoras de levaduras y bacterias optimizadas.
Así, en un aspecto de la invención, se proporciona un ácido nucleico aislado que comprende una secuencia promotora procedente del nucleótido en posición 4 al nucleótido en posición 545 de la Figura 1C que dirige la expresión de una secuencia codificante unida de manera operable tanto en levaduras como en bacterias. También se proporcionan plásmidos que comprenden este promotor seleccionado del grupo constituido por (SEQ ID NO: 2) (pGLS20) , (SEQ ID NO: 3) (pGLS22) y pGLS23 en el que pGLS23 es idéntico a pGLS22 excepto por un cambio en la posición 3267 de gaattC a gaattA y pBR-AMP-aLPL (SEQ ID NO: 5) . También se describen células hospedadoras que comprenden estos plásmidos. Dichas células hospedadoras son preferentemente células de E. coli y S. cerevisiae.
En el presente documento también se describen nuevas cepas de E. coli. Se proporcionan E. coli KJI 567 y E. coli AG58A (RP28) . También se describen nuevas cepas de levadura, por ejemplo, S. cerevisiae PRT50 y S. cerevisiae PRT475.
En una forma de realización preferida de la invención, se proporciona un procedimiento para comparar las interacciones de unión entre una primera proteína y una segunda proteína tanto en entornos de organismos bacterianos como de levaduras utilizando una construcción que funciona en ambos organismos, que comprende:
a) el suministro de células hospedadoras de levaduras y bacterianas, comprendiendo cada una, i) un gen informador unido de manera operable a una secuencia de ADN que comprende un sitio de unión a proteínas;
ii) un primer gen de fusión que expresa una primera proteína de fusión, dicha primera proteína de fusión comprendiendo dicha primera proteína unida covalentemente a un resto de unión que es capaz de unirse específicamente a dicho sitio de unión a proteínas que está dirigido por un elemento promotor según la reivindicación 1 y iii) un segundo gen de fusión que expresa una segunda proteína de fusión, dicha segunda proteína de fusión comprendiendo dicha segunda proteína unida covalentemente al resto de activación génico;
b) permitir que dichas primera y segunda proteínas interaccionen; y c) medir la expresión de dicho gen informador como medida de dicha interacción entre dichas primera y segunda proteínas tanto en E. coli como en levaduras.
En la presente invención también se proporcionan kits para poner en práctica el procedimiento descrito anteriormente. Un kit de ejemplo comprende:
a) un plásmido seleccionado del grupo constituido por la SEQ ID NO: 2 (pGLS20) , SEQ ID NO: 3 (pGLS22) y pGLS23 en el que pGLS23 es idéntico a pGLS22 excepto por un cambio en la posición 3667 de gaattC a gaattA;
b) al menos uno de pAC-AW-aLPL, y/o pBR-AMP-aLPL;
c) una cepa de E. coli seleccionada del grupo constituido por E. coli KJI 567 o E. coli AG58A (RP28) ; y d) una cepa de levaduras seleccionada del grupo constituido por PRT50, y PRT475.
BREVE DESCRIPCIÓN DE LOS DIBUJOS
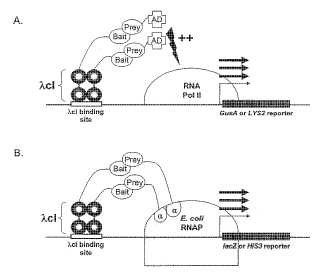
Figura 1. Esquema de los sistemas bihíbridos de levaduras y bacterias. A. En el sistema bihíbrido de levaduras mostrado, una proteína híbrida dimérica A cI-cebo interacciona con un dominio de activación (AD) -proteína híbrida presa estimulando así la transcripción a partir de un promotor adyacente que dirige la expresión de un gen informador GusA cuantitativo o LYS2 seleccionable. B. En el sistema bihíbrido bacteriano mostrado, una proteína híbrida dimérica A cI-cebo interacciona con una subunidad a de la ARN polimerasa de E. coli (ARNP) -proteína híbrida presa, enganchando así la ARNP a un promotor adyacente que dirige la expresión de un gen informador lacZ cuantitativo o HIS3 seleccionable. Nótese que ambos sistemas utilizan una proteína híbrida A cI-cebo procedente de un único plásmido eficaz en cualquiera de los organismos. C-E. Secuencia en pGLS20 (SEQ ID NO: 2) . F-H. Secuencia en pGLS22 (SEQ ID NO: 3) . I-J. Secuencia en pBR_AMP alphaLPL (SEQ ID NO: 5) .
Figura 2. Expresión del cebo a partir de un plásmido de expresión combinada en bacterias/levaduras. A. Los plásmidos pGLS20 y pGLS23 utilizan un promotor TEFI/uvLac combinado para expresar cebos A cI fusionados en levaduras o bacterias. Los plásmidos se seleccionan en levaduras mediante selección para resistencia a G418 (pGLS20) o complementación HISS (pGLS23) , y en bacterias mediante selección por resistencia a kanamicina (pGLS20) o por resistencia a cloranfenicol (pGLS23) . La expresión relativa de cebos cl a partir de estos plásmidos, frente a la descripta previamente para los vectores pGBS10 (bihíbrido en levaduras, (Serebriiskii y col. 2002) ) o pBT (bihíbrido en bacterias, Stratagene) se muestra en bacterias (panel central) . B. Para demostrar los niveles relativos de cebo se confirmó la concentración de proteína total igual mediante tinción de Coomassie de un gel PAGE cargado con cantidades equivalentes de lisado celular para bacterias que expresan cada plásmido (no mostrados) . A continuación, se cargaron volúmenes iguales de diluciones de 1:40 (para pGLS20) o 1:100 (para pGT) de extractos en tampón... [Seguir leyendo]
Reivindicaciones:
1. Un ácido nucleico aislado que comprende una secuencia promotora procedente del nucleótido en posición 4 al nucleótido en posición 545 de la Figura 1C que dirige la expresión de una secuencia codificante unida de manera operable tanto en levaduras como en bacterias.
2. Un plásmido que comprende el promotor de la reivindicación 1 seleccionado del grupo constituido por la SEQ ID NO: 2 (pGLS20) , SEQ ID NO: 3 (pGLS22) y pGLS23 en el que pGLS23 es idéntico a pGLS22 excepto por un cambio en la posición 3267 de gaattC a gaattA.
3. Una célula hospedadora que expresa un plásmido de la reivindicación 2.
4. La célula hospedadora de la reivindicación 3, seleccionada del grupo constituido por células eucariotas y células bacterianas.
5. La célula hospedadora de la reivindicación 4 seleccionada del grupo constituido por S. cerevisiae PRT50, y cepas diploides que resultan del apareamiento de las cepas SKY191 y PRT50 con las cepas homólogas adecuadas.
6. La célula hospedadora de la reivindicación 4, que es E. coli KJ1567 o E. coli AG58A (RP28) .
7. Un procedimiento para comparar las interacciones de unión entre una primera proteína y una segunda proteína tanto en entornos de organismos bacterianos como de levaduras utilizando una construcción que funciona en ambos organismos, que comprende:
a) el suministro de células hospedadoras de levaduras y bacterianas, comprendiendo cada una, i) un gen informador unido de manera operable a una secuencia de ADN que comprende un sitio de unión a proteínas; ii) un primer gen de fusión que expresa una primera proteína de fusión, dicha primera proteína de fusión comprendiendo dicha primera proteína unida covalentemente a un resto de unión que es capaz de unirse específicamente a dicho sitio de unión a proteínas que está dirigido por una secuencia promotora entre el nucleótido en posición 4 y el nucleótido en posición 545 de la Figura 1C, y iii) un segundo gen de fusión que expresa una segunda proteína de fusión, dicha segunda proteína de fusión comprendiendo dicha segunda proteína unida covalentemente al resto de activación génico;
b) permitir que dichas primera y segunda proteínas interaccionen; y c) medir la expresión de dicho gen informador como medida de dicha interacción entre dichas primera y segunda proteínas tanto en E. coli como en levaduras.
8. Un sistema bihíbrido unificado de levaduras y bacterias para la puesta en práctica del procedimiento de la reivindicación 7, que comprende:
a) un plásmido seleccionado del grupo constituido por la SEQ ID NO: 2 (pGLS20) , SEQ ID NO: 3 (pGLS22) y pGLS23 en el que pGLS23 es idéntico a pGLS22 excepto por un cambio en la posición 3267 de gaattC a gaattA;
b) al menos un plásmido presa seleccionado del grupo constituido por pBR-AMP-aLPL (SEQ ID NO: 5) y pAC-AMPaLPL;
c) una cepa de E. coli seleccionada del grupo constituido por E. coli KJ1567 o E. coli AG58A (RP28) ; y d) una cepa de levaduras seleccionada del grupo constituido por PRT50, SKY191 y PRT475.
Patentes similares o relacionadas:
 Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Anticuerpo biespecífico o mezcla de anticuerpos con cadenas ligeras comunes, del 15 de Julio de 2020, de Jiangsu Alphamab Biopharmaceuticals Co., Ltd: Anticuerpo biespecífico o parte de unión a antígeno del mismo, en el que el anticuerpo biespecífico o la parte de unión a antígeno del mismo tiene una cadena […]
Vacunas y diagnóstico de torque teno virus porcino, del 18 de Junio de 2020, de VIRGINIA TECH INTELLECTUAL PROPERTIES, INC.: Composición inmunogénica que comprende una proteína según SEQ ID NO. 16.
Producción de FDCA catalizada por deshidrogenasa, del 17 de Junio de 2020, de PURAC BIOCHEM B.V.: Proceso para oxidar ácido 5-hidroximetil-2-furancarboxílico (HMFCA) a ácido 5-formil-2-furoico (FFA), donde el proceso comprende la etapa de incubar una […]
Vectores de AAV dirigidos a oligodendrocitos, del 10 de Junio de 2020, de THE UNIVERSITY OF NORTH CAROLINA AT CHAPEL HILL: Un ácido nucleico que codifica una cápside de AAV, comprendiendo el ácido nucleico una secuencia codificante de la cápside de AAV que es al menos el 96 % idéntica […]
Ácido nucleico que comprende o codifica para un tallo-bucle de histona y una secuencia poli(A) o una señal de poliadenilación para aumentar la expresión de un antígeno patogénico codificado, del 3 de Junio de 2020, de CureVac AG: Secuencia de ácido nucleico que comprende o codifica en la dirección 5' → 3' para: i) • una región codificante, que codifica para al menos […]
Agrupación de genes de biosíntesis de carrimicina, del 27 de Mayo de 2020, de Shenyang Fuyang Pharmaceutical Technology Co., Ltd: Agrupación de genes de biosíntesis de carrimicina, que consiste en 44 genes que comprende: 1) cinco genes de policétido sintasa, incluyendo los residuos […]
Microorganismo con productividad de l-lisina aumentada y procedimiento para producir l-lisina utilizando el mismo, del 27 de Mayo de 2020, de CJ CHEILJEDANG CORPORATION: Una subunidad beta prima (subunidad-β') mutante de la ARN polimerasa, en la que la subunidad beta prima (subunidad-β') mutante de la ARN polimerasa tiene […]