Detección de ácidos nucleicos con ribonucleótidos marcados con etiquetas.
Un método para detectar un ácido nucleico diana, el método comprende:
(a) poner en contacto el ácido nucleico diana con un polinucleótido, un grupo de nucleótidos y un nucleótido que incorpora un componente biocatalítico, en el que;
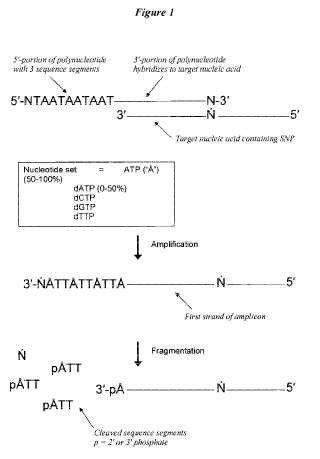
(i) el polinucleótido comprende una porción 5’ y una porción 3’, la porción 5’ comprende al menos uno o al menos dos segmentos de secuencias contiguos, en el que cada segmento de secuencia comprende al menos tres bases de nucleótido, y la base de nucleótido del extremo 5’ de cada segmento de secuencia es único dentro de un segmento de secuencia y es el mismo en cada segmento de secuencia; y la porción 3’ comprende un segmento de secuencia que es sustancialmente complementario y suficiente para hibridar con el ácido nucleico diana y extenderse bajo condiciones de amplificación;
(ii) el grupo de nucleótidos comprende al menos dos bases de nucleótido en forma de desoxiribonucleótidos (dNTP), y la mayoría de al menos una base de nucleótido en forma de un ribonucleótido (rNTP), en el que la base del ribonucleótido es complementaria a la base de nucleótido 5’ única de cada segmento de secuencia del polinucleótido; y
(iii) el nucleótido que incorpora un componente biocatalítico comprende un desoxiribonucleótido y un ribonucleótido que incorpora actividades;
(b) amplificar el ácido nucleico diana bajo condiciones de amplificación en presencia de uno o más pares de cebadores para producir un amplicón que comprende un segmento de secuencia 3’ complementaria a la porción 5’ del polinucleótido, en el que el segmento de secuencia 3’ comprende uno o más segmentos de secuencias contiguos, en el que la base de nucleótido del extremo 3’ de cada segmento de secuencia es un nucleósido monofosfato (rNMP) que posee la misma base que el rNTP en el grupo de nucleótidos, en el que al menos uno de los cebadores de dicha pareja de cebadores comprende el polinucleótido de acuerdo con (i) de la reivindicación 1;
(c) escindir el amplicón 3’ de cada NMP en fragmentos, en el que la escisión libera los segmentos de secuencias como fragmentos individuales ; y
(d) detectar los fragmentos de amplicón, en el que los fragmentos de los segmentos de secuencias detectados indican la amplificación de la secuencia de ácido nucleico diana, detectando por lo tanto la secuencia de ácido nucleico diana.
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E09005451.
Solicitante: F. HOFFMANN-LA ROCHE AG.
Nacionalidad solicitante: Suiza.
Dirección: GRENZACHERSTRASSE 124 4070 BASEL SUIZA.
Inventor/es: GUT, IVO, GLYNNE, GELFAND, DAVID, H., BAUER,Keith A, MAUGER,FLORENCE.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2392783_T3.pdf
Fragmento de la descripción:
Detección de ácidos nucleicos con ribonucleótidos marcados con etiquetas
Campo de la invención
La presente invención está relacionada con el campo de la detección de ácidos nucleicos. En particular, la presente invención proporciona polinucleótidos que poseen múltiples segmentos de secuencia contiguos, en el que la base de nucleótido del extremo 5’ de cada segmento de secuencia es único dentro del segmento de secuencia y es el mismo en cada segmento de secuencia, y los métodos para su utilización en la detección de ácidos nucleicos diana.
Antecedentes de la invención
Se conocen muchos métodos para la detección de ácidos nucleicos diana (por ejemplo, genotipaje de SNP) . Los ensayos homogéneos actualmente disponibles para el genotipaje de SNP incluye los ensayos de cebador TASMAN®, AMPLIFLUOR®, unión de colorantes, PCR cinética selectiva de alelo, y SCORPION®. Estos ensayos proporcionan uno, o como máximo dos (si se utilizan cuatro tinciones fluorescentes diferentes) SNP por pocillo de reacción (por ejemplo la PCR cinética de unión a colorantes requiere dos pocillos por cada SNP) . Los métodos disponibles para el genotipaje de SNP están en el rango de aquellos que permiten el genotipaje de un único SNP en un pocillo de reacción hasta los métodos que permiten el genotipaje de varios miles de SNP en un único pocillo (por ejemplo, ensayo GOLDEN GATE®, Illumina) . Los presentes procedimientos de ensayo de genotipaje no se pueden multiplexar fácilmente debido a que requieren de una tinción diferente por cada tipo de alelo, y por lo tanto está limitado en su potencial para mejorar. El ensayo GOLDEN GATE® requiere de dispositivos de análisis complejos como un lector de fibra óptica. Los analizadores para la lectura de genotipaje de SNP están en el rango de lectores de placas (opcionalmente, con una máquina de PCR incluida) , hasta secuenciadores (por ejemplo, secuenciadores capilares) , lectores de matrices y espectrómetros de masas. Además, aunque los espectrómetros de masas se han utilizado para el genotipaje, el procedimiento actualmente disponible no permite el verdadero multiplexado.
Breve resumen de la invención
La presente invención proporciona polinucleótidos para la eficiente y simultánea detección altamente sensible de uno o más ácidos nucleicos diana. Los polinucleótidos están diseñados con una porción 5' que comprende uno, dos o más segmentos de secuencias contiguos, de tal manera que la masa de un segmento de secuencia, o complemento de la misma, identifica un ácido nucleico diana o un nucleótido diana dentro de un ácido nucleico diana. En algunas realizaciones, el segmento de secuencia son de la misma masa. Los segmentos de secuencias de igual masa puede, pero no necesita tener la misma secuencia. El segmento de secuencia se escinde del complemento del polinucleótido y su masa se mide o detecta para identificar la presencia, ausencia o nivel de presencia de un ácido nucleico diana.
En consecuencia, la presente Invención está relacionada con un polinucleótido que comprende uno, dos o más segmentos de secuencias. En algunas realizaciones, el polinucleótido comprende una porción 5’ y una porción 3', en la que
a) la porción 5 ' comprende al menos uno o dos segmentos de secuencias contiguos, en el que cada segmento de secuencia comprende al menos tres bases de nucleótido, es de igual masa, y el extremo 5 'de la base de nucleótido de cada segmento de secuencia es único dentro de un segmento de secuencia y es el mismo en cada segmento de secuencia; y
b) la porción 3' comprende al menos 5 nucleótidos, en el que el polinucleótido tiene menos de aproximadamente 100 bases de nucleótido de longitud. En algunas realizaciones, el polinucleótido es menor de aproximadamente 75, 50 o 25 nucleótidos de longitud.
En realizaciones preferibles, cada segmento de secuencia posee la misma secuencia de nucleótido.
En otras realizaciones preferibles, la porción 5’ comprende al menos 3, 4, 5, 6, 7 o 8 segmentos de secuencias contiguos. En algunas realizaciones preferibles, cada segmento de secuencia comprende al menos 3, 4, 5, 6, 7 o 8 nucleótidos.
En también realizaciones preferibles, el extremo 3’ comprende una porción de bloqueo extraíble que previene sustancialmente la extensión del polinucleótido. En realizaciones especialmente preferibles, el extremo 3’ comprende una porción terminadora 2’.
La invención proporciona métodos para detectar un ácido nucleico diana. En algunas realizaciones, los métodos comprenden:
(a) poner en contacto el ácido nucleico diana con un polinucleótido, un grupo de nucleótidos y un nucleótido que incorpora un componente biocatalítico, en el que;
(i) el polinucleótido comprende una porción 5’ y una porción 3’, la porción 5’ comprende al menos uno o al menos dos segmentos de secuencias contiguos, en el que cada segmento de secuencia comprende al menos tres bases de nucleótido, y la base de nucleótido del extremo 5’ de cada segmento de secuencia es único dentro de un segmento de secuencia y es el mismo en cada segmento de secuencia; y la porción 3’ comprende un segmento de secuencia que es sustancialmente complementario y suficiente para hibridar con el ácido nucleico diana y extenderse bajo condiciones de amplificación;
(ii) el grupo de nucleótidos comprende al menos dos bases de nucleótido en forma de desoxiribonucleótidos (dNTP) , y la mayoría de al menos una base de nucleótido en forma de un ribonucleótido (rNTP) , en el que la base del ribonucleótido es complementaria a la base de nucleótido 5’ única de cada segmento de secuencia del polinucleótido; y
(iii) el nucleótido que incorpora un componente biocatalítico comprende un desoxiribonucleótido y un ribonucleótido que incorporan actividades;
(b) amplificar el ácido nucleico diana bajo condiciones de amplificación en presencia de uno o más pares de cebadores para producir un amplicón que comprende un segmento de secuencia 3’ (es decir una porción 3’) complementaria a la porción 5’ del polinucleótido, en el que el segmento de secuencia 3’ comprende al menos uno o al menos dos segmentos de secuencias, en el que la base de nucleótido del extremo 3’ de cada segmento de secuencia es un nucleósido monofosfato (NMP) que posee la misma base que el NTP en el grupo de nucleótidos;
(c) escindir el amplicón 3’ de cada NMP en fragmentos, en la que la escisión libera el segmento de secuencia como fragmentos individuales; y
(d) detectar los fragmentos de amplicón, en el que los fragmentos del segmento de secuencia detectados indican la amplificación de la secuencia de ácido nucleico diana, detectando por lo tanto la secuencia de ácido nucleico diana.
En realizaciones preferibles, el segmento de secuencia de la porción 5’ es de la misma masa.
En otras realizaciones preferibles, al menos el 80% de al menos una base de nucleótido está en forma de un ribonucleótido (rNTP) .
En otras realizaciones preferibles, la escisión se realiza sometiendo al amplicón a una solución alcalina.
En realizaciones preferibles, la solución alcalina comprende al menos una de las siguientes: NaOH, KOH, RbOH, Mg (OH) 2, Ca (OH) 2, o NH4OH. En más realizaciones preferibles, la solución alcalina comprende al menos una de las siguientes: NaOH, KOH o NH4OH.
En otras realizaciones preferibles, los métodos comprenden además el paso de poner en contacto la secuencia del ácido nucleico diana con una pareja de polinucleótidos, en el que cada polinucleótido en la pareja de polinucleótidos comprende el segmento de secuencia de la porción 5’ de la misma masa.
En otras realizaciones preferibles, los métodos comprenden además el paso de poner en contacto el ácido nucleico diana con al menos un polinucleótido, en el que la detección de los fragmentos del segmento de secuencia del polinucleótido, o su complementario, indica la presencia de un alelo de un nucleótido polimórfico en la secuencia de ácido nucleico diana.
En otras realizaciones preferibles, los métodos comprenden además el paso de poner en contacto el ácido nucleico diana con al menos dos polinucleótidos diferentes, en el que los... [Seguir leyendo]
Reivindicaciones:
1. Un método para detectar un ácido nucleico diana, el método comprende:
(a) poner en contacto el ácido nucleico diana con un polinucleótido, un grupo de nucleótidos y un nucleótido que incorpora un componente biocatalítico, en el que;
(i) el polinucleótido comprende una porción 5’ y una porción 3’, la porción 5’ comprende al menos uno o al menos dos segmentos de secuencias contiguos, en el que cada segmento de secuencia comprende al menos tres bases de nucleótido, y la base de nucleótido del extremo 5’ de cada segmento de secuencia es único dentro de un segmento de secuencia y es el mismo en cada segmento de secuencia; y la porción 3’ comprende un segmento de secuencia que es sustancialmente complementario y suficiente para hibridar con el ácido nucleico diana y extenderse bajo condiciones de amplificación;
(ii) el grupo de nucleótidos comprende al menos dos bases de nucleótido en forma de desoxiribonucleótidos (dNTP) , y la mayoría de al menos una base de nucleótido en forma de un ribonucleótido (rNTP) , en el que la base del ribonucleótido es complementaria a la base de nucleótido 5’ única de cada segmento de secuencia del polinucleótido; y
(iii) el nucleótido que incorpora un componente biocatalítico comprende un desoxiribonucleótido y un ribonucleótido que incorpora actividades;
(b) amplificar el ácido nucleico diana bajo condiciones de amplificación en presencia de uno o más pares de cebadores para producir un amplicón que comprende un segmento de secuencia 3’ complementaria a la porción 5’ del polinucleótido, en el que el segmento de secuencia 3’ comprende uno o más segmentos de secuencias contiguos, en el que la base de nucleótido del extremo 3’ de cada segmento de secuencia es un nucleósido monofosfato (rNMP) que posee la misma base que el rNTP en el grupo de nucleótidos, en el que al menos uno de los cebadores de dicha pareja de cebadores comprende el polinucleótido de acuerdo con (i) de la reivindicación 1;
(c) escindir el amplicón 3’ de cada NMP en fragmentos, en el que la escisión libera los segmentos de secuencias como fragmentos individuales ; y
(d) detectar los fragmentos de amplicón, en el que los fragmentos de los segmentos de secuencias detectados indican la amplificación de la secuencia de ácido nucleico diana, detectando por lo tanto la secuencia de ácido nucleico diana.
2. El método de la reivindicación 1, que comprende poner en contacto la secuencia del ácido nucleico diana con dos polinucleótidos de acuerdo con (i) de la reivindicación 1, en el que cada polinucleótido en la pareja de polinucleótidos comprenden segmentos de secuencias de la porción 5’ de la misma masa.
3. El método de la reivindicación 1, que comprende poner en contacto el ácido nucleico diana con al menos un polinucleótido de acuerdo con (i) de la reivindicación 1, en el que la detección de los fragmentos del segmento de secuencia del complemente del polinucleótido indica la presencia de un alelo de un nucleótido polimórfico en la secuencia del ácido nucleico diana.
4. El método de la reivindicación 1, que comprende poner en contacto el ácido nucleico diana con al menos dos polinucleótidos diferentes de acuerdo con (i) de la reivindicación 1, en el que la porción 3’ de los polinucleótidos difiere solo en la base de nucleótido del extremo 3’, en el que la base de nucleótido del extremo 3’ de cada polinucleótido corresponde con segmentos de secuencia de masa única, y la detección de los fragmentos del segmento de secuencia del complemente de al menos uno de los polinucleótidos indica la presencia de un alelo de un nucleótido polimórfico en la secuencia del ácido nucleico diana.
5. El método de la reivindicación 1, en el que un grupo de diferentes secuencias de ácido nucleico diana se pone en contacto con un grupo de polinucleótidos diferentes de acuerdo con (i) de la reivindicación 1, cada uno de los polinucleótidos diferentes comprende un segmento de secuencia de masa única, en el que los segmentos de secuencias de polinucleótidos son identificadores de uno del grupo de secuencias de ácido nucleico diana diferentes, mientras que la detección de los fragmentos del segmento de secuencia del complementario de al menos un de los polinucleótidos indica la presencia de al menos uno del grupo de secuencias de ácido nucleico diana diferentes.
6. El método de la reivindicación 1, en el que el polinucleótido de acuerdo con (i) de la reivindicación 1 comprende una porción de bloqueo que previene sustancialmente la extensión del polinucleótido, y en el que el método comprende además el paso de eliminar la porción de bloqueo del polinucleótido antes de amplificar.
FIGURA 1
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]