MÉTODO PARA LA AMPLIFICACIÓN DE ALELOS HLA DE CLASE I.
Método para tipar o subtipar uno o más alelos HLA-A en una muestra,
que comprende las siguientes etapas: (i) si es necesario, liberar, aislar y/o concentrar los ácidos nucleicos presentes en dicha muestra; (ii) amplificar de forma separada, específica del locus, el exón 2, exón 3 y exón 4 de los alelos HLA-A, haciendo uso de al menos tres conjuntos de cebadores, en los que - para la amplificación del exón 2, el cebador inverso se hibrida específicamente a una secuencia diana específica del locus en el intrón 2 de HLA-A, - para la amplificación del exón 3, el cebador directo se hibrida específicamente a una secuencia diana específica del locus en el intrón 2 de HLA-A, y/o el cebador inverso se hibrida específicamente a una secuencia diana específica del locus en el intrón 3 de HLA-A, y - para la amplificación del exón 4, el cebador directo se hibrida específicamente a una secuencia diana específica del locus en el intrón 3 de HLA-A; (iii) tipar el alelo HLA-A específico presente en dicha muestra
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2000/002998.
Solicitante: INNOGENETICS N.V..
Nacionalidad solicitante: Bélgica.
Dirección: INDUSTRIEPARK ZWIJNAARDE 7, BOX 4 9052 GHENT BELGICA.
Inventor/es: ROSSAU, RUDI, DE CANCK, ILSE, ROMBOUT,ANNELIES.
Fecha de Publicación: .
Fecha Solicitud PCT: 5 de Abril de 2000.
Fecha Concesión Europea: 25 de Agosto de 2010.
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
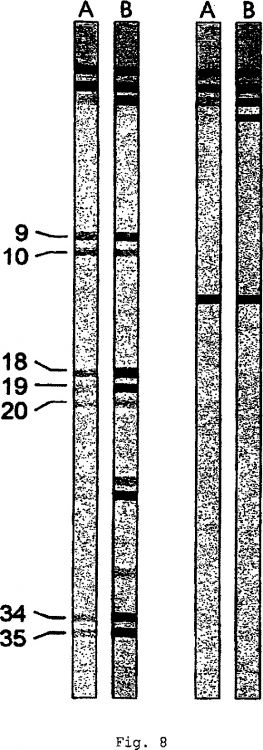
Fragmento de la descripción:
Método para la amplificación de alelos HLA de clase I.
Campo de la invención
La presente invención se refiere a un método para el tipado o subtipado de HLA-A. Más específicamente, la presente invención se refiere a un método para la amplificación separada, específica del locus, del exón 2, exón 3 y exón 4 de HLA-A.
Antecedentes de la invención
El complejo mayor de histocompatibilidad (MHC) humano está contenido en alrededor de 4 Mpb de ADN en el brazo corto del cromosoma 6 en 6p21.3 (Campbell y Trowsdale, 1993). El MHC humano se divide en las regiones de clase I, clase II y clase III. Los genes de clase I y clase II codifican moléculas de la superficie celular muy polimórficas que se unen y presentan antígenos procesados en forma de péptidos a linfocitos T, iniciando respuestas inmunitarias tanto celulares como humorales.
Las moléculas de clase I del MHC humano, HLA-A, -B, y -C, se encuentran en la mayoría de las células nucleadas. Son glucoproteínas de la superficie celular que se unen y presentan péptidos procesados derivados de proteínas endógenamente sintetizadas a células T CD8+. Estos heterodímeros consisten en una cadena α codificada por HLA asociada con un polipéptido monomórfico no codificado por MHC, β2-microglobulina (Townsend y Bodmer, 1989; Spencer y Parham, 1996). Las moléculas de clase II del MHC humano están codificadas en la región de HLA-D. Estas glucoproteínas de la superficie celular consisten en una cadena α y β codificada por HLA, asociada con heterodímeros sobre la superficie celular de células que presentan antígenos, tales como células B y macrófagos. Las moléculas de clase II sirven como receptores para péptidos procesados. Sin embargo, estos péptidos derivan predominantemente de proteínas membránicas y extracelulares, y se presentan a células T CD4+. La región de HLA-D contiene varios genes de clase II, y tiene tres subregiones principales: HLA-DR, -DQ, y -DP. Tanto las regiones HLA-DQ como -DP contienen un gen funcional para cada una de sus cadenas α y β. La subregión de HLA-DR contiene un gen funcional para la cadena α; el número de genes funcionales para la cadena β varía de uno a dos según el haplotipo (Andersson et al., 1987: Apple y Erlich, 1996).
Existe un amplio polimorfismo en la mayoría de los locus. A la vista de la importancia biológica y médica de estos antígenos, se requiere una técnica rápida y muy sensible para el tipado de HLA. Actualmente se usa una variedad de técnicas para detectar el polimorfismo de HLA, incluyendo el reconocimiento serológico, bioquímico, de células T y, más recientemente, métodos biológicos moleculares.
La serología sigue siendo el método de sostén para el tipado de HLA - especialmente para clase I - para muchos laboratorios de histocompatibilidad rutinarios. El microensayo de linfocitotoxicidad (Kissmeyer et al., 1969; Terasaki y McClelland, 1964) es el enfoque estándar: se mezclan células mononucleares de sangre periférica (clase I) viables o células B separadas (clase II) con antisueros (policlonales o monoclonales) de especificidad por HLA conocida.
La detección del polimorfismo se puede lograr observando la diferente composición de aminoácidos de las moléculas de HLA mediante técnicas bioquímicas tales como el enfoque isoeléctrico unidimensional (IEF; Yang, 1987). Este método se basa en sustituciones de aminoácidos que contribuyen a cambios en la carga de la molécula de HLA.
Otro método de tipado de HLA es la reacción linfocitaria mixta (MLR). Concurrente con las observaciones realizadas usando antisueros específicos de HLA, se observó que los linfocitos procedentes de dos fuentes no relacionadas, cuando se mezclan en cultivo, proliferan (Hirschom et al., 1963).
El análisis de las especificidades de HLA a partir de ADN proporcionó un nuevo enfoque para definir sus diferencias polimórficas. En lugar de fijarse en las diferencias en la molécula expresada, el polimorfismo se caracteriza a nivel nucleotídico.
Un desarrollo importante y poderoso en el campo de la biología molecular ha sido la reacción en cadena de la polimerasa (PCR; Mullis et al., 1986; Mullis y Faloona, 1987). En el tipado tisular, la PCR se usa para amplificar las regiones polimórficas de genes de HLA. Este producto de la PCR de HLA se puede analizar entonces en busca de sus diferencias polimórficas, para establecer el tipo de tejido. Se ha desarrollado un número de tales enfoques, incluyendo el análisis de heterodúplex de productos de la PCR (Clay et al., 1994), el análisis de polimorfismo conformacional monocatenario del producto de la PCR (PCR-SSCP; Yoshida et al., 1992), el tipado a base de secuencias (SBT; Santamaria et al., 1992 y 1993), el uso de cebadores específicos de secuencias en la reacción de PCR (PCR-SSP; Olerup y Zetterquist, 1991), el uso de PCR en combinación con el sondaje oligonucleotídico específico de secuencias (PCR-SSOP; Saiki et al., 1986) o el sondaje mediante transferencia de punto inversa (Saiki et al., 1989). Estos enfoques, usados de forma individual o en combinación, se han aplicado todos como métodos basados en ADN para el tipado tisular de las especificidades de HLA de clase I y clase II. Los métodos de tipado de ADN se deberían de preferir con respecto a los métodos serológicos con la condición de que esté disponible un método de tipado de ADN fácil, rápido y fiable. Algunas diferencias a nivel de subtipo, que son detectables mediante los métodos de ADN, pueden no ser detectadas mediante los métodos actuales de tipado serológico, aunque estas diferencias pueden provocar rechazo de haloinjertos (Fleischhauer et al., 1990).
El sistema de HLA es el sistema genético humano más polimórfico todavía conocido. Los genes de HLA clase I comparten una estructura similar (desde 5' a 3'): una región de flanqueo no traducida en 5', un primer exón (exón 1) que tiene una longitud de aproximadamente 73 pares de bases, un primer intrón (intrón 1) que tiene una longitud de aproximadamente 130 pares de bases, un segundo exón (exón 2), que tiene una longitud de aproximadamente 250 pares de bases, un segundo intrón (intrón 2), que tiene una longitud de aproximadamente 272 pares de bases, un tercer exón (exón 3), que tiene una longitud de aproximadamente 276 pares de bases, un tercer intrón (intrón 3), que tiene una longitud de aproximadamente 588 pares de bases, y un cuarto exón (exón 4), que tiene una longitud de aproximadamente 276 pares de bases. Las sustituciones polimórficas en los alelos HLA de clase I están mayoritariamente localizados tanto en el exón 2 como en el exón 3, que codifican la ranura de unión a péptidos de la molécula de clase I. Estos polimorfismos hacen una diferenciación entre alelos que se pueden lograr a través de una variedad de técnicas biológicas moleculares, tales como secuenciación o hibridación con sondas relevantes. En los kits de diagnóstico actuales, el exón 2 y el exón 3, se amplifican juntos, dando como resultado amplicones de alrededor de 1 kb, que consisten en al menos el exón 2, el intrón 2 y el exón 3. Existen cebadores específicos del locus para la amplificación de estos amplicones de 1 kb. Sin embargo, tales amplicones grandes son difíciles de amplificar y muestran una formación de estructura secundaria, dando como resultado la hibridación ineficaz de algunas sondas. Además, debido a la aparición de nuevos alelos HLA de clase I, ya no se pueden distinguir ciertas combinaciones alélicas mediante la detección de polimorfismos sólo en el exón 2 y en el exón 3, y se requiere un tipado adicional en el exón 4. Esto provoca la necesidad de la amplificación adicional del exón 4, dando como resultado un amplicón incluso más grande. Por lo tanto, sería deseable una amplificación separada del exón 2, exón 3 y exón 4, dando como resultado productos de amplificación que permiten un tipado más eficaz de alelos HLA de clase I. Sin embargo, puesto que los sitios de hibridación a cebadores específicos del locus son escasos y no se pueden encontrar en el exón 2, exón 3 o exón 4, la amplificación separada y específica del locus del exón 2, exón 3 y exón 4 de HLA-A, HLA-B o HLA-C no es tan evidente.
En los documentos in WO 99/07883, WO 97/23645, WO 98/26091, por ejemplo, se describen métodos y kits para el tipado de HLA-A a base de la amplificación de exones 2 y 3. En los documentos EP-A-0414469, WO 97/23650, WO 97/20197 se describen, por ejemplo, cebadores específicos de HLA-A.
Objetivos...
Reivindicaciones:
1. Método para tipar o subtipar uno o más alelos HLA-A en una muestra, que comprende las siguientes etapas:
2. Método según la reivindicación 1, caracterizado además porque la secuencia diana específica del locus está situada en la posición 67, 96, 109, 110, 118, 123, 131 ó 181 del intrón 2 de HLA-A (Fig. 1) y/o posición 32, 50, 62, 73, 83, 86, 118, 130, 150, 501, 525, 561 ó 571 del intrón 3 de HLA-A (Fig. 4).
3. Método según la reivindicación 2, caracterizado además porque dichas posiciones constituyen el extremo 3' del cebador que se usa para la amplificación del exón 2, exón 3 o exón 4.
4. Método según la reivindicación 3, caracterizado además porque el cebador se escoge de la siguiente lista:








5. Método según cualquiera de las reivindicaciones 1 a 4, caracterizado además porque la amplificación del exón 2 se lleva a cabo el siguiente cebador directo:
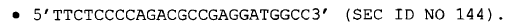
6. Método según cualquiera de las reivindicaciones 1 a 5, caracterizado además porque la amplificación del exón 4 se lleva a cabo con el siguiente cebador inverso:
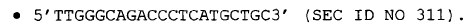
7. Método según cualquiera de las reivindicaciones 1 a 6, caracterizado además porque:
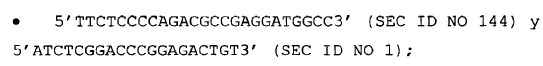


8. Método según cualquiera de las reivindicaciones 1 a 7, caracterizado además porque tanto el exón 2 como el exón 3 de HLA-A se amplifican mediante uso de una mezcla de cebadores múltiple que contiene al menos un par de cebadores para la amplificación del exón 2 y al menos un par de cebadores para la amplificación del exón 3.
9. Método según cualquiera de las reivindicaciones 1 a 8, caracterizado además porque los tres exones, exón 2, exón 3 y exón 4, de HLA-A se amplifican mediante uso de una mezcla de cebadores múltiple que contiene al menos un par de cebadores para la amplificación del exón 2, al menos un par de cebadores para la amplificación del exón 3 y al menos un par de cebadores para la amplificación del exón 4.
10. Método según cualquiera de las reivindicaciones 1 a 9, caracterizado además porque la etapa de tipado se lleva a cabo mediante hibridación con una o más sondas adecuadas.
11. Un kit de diagnóstico para tipar o subtipar uno o más alelos HLA-A en una muestra, que comprende los siguientes componentes:
12. Un kit de ensayo sondas en líneas para el tipado o subtipado de uno o más alelos HLA-A en una muestra, que comprende los siguientes componentes:
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]