USO DE HEMOLISINA CLYA PARA LA EXCRECION DE PROTEINAS DE FUSION.
Un método para expresar un gen en una célula bacteriana que comprende:
proporcionar un vector de expresión para una población de células bacterianas sin transformar, en el que el vector de expresión comprende un casete de expresión que comprende una secuencia que codifica una proteína de exportación fusionada genéticamente a una secuencia que codifica una proteína de interés; expresar el casete de expresión tal que una proteína de fusión proteína de exportación::proteína de interés es producida y exportada en el medio de cultivo, en el que la la secuencia que codifica una proteína de exportación es seleccionada del grupo que consiste en gen (clyA) citolisina A de Salmonella serovar entérica Typhi (S. Typhi), gen clyA (S. paratyphi) de Salmonella paratyphi o el gen hemolisina E (hlyE) de Eschericia coli (E. coli)
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2001/044185.
Solicitante: UNIVERSITY OF MARYLAND, BALTIMORE.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 620 WEST LEXINGTON STREET, 4TH FLOOR BALTIMORE, MARYLAND 21201 ESTADOS UNIDOS DE AMERICA.
Inventor/es: GALEN,JAMES,E.
Fecha de Publicación: .
Fecha Solicitud PCT: 23 de Noviembre de 2001.
Fecha Concesión Europea: 21 de Julio de 2010.
Clasificación Internacional de Patentes:
- C12N15/70 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Vectores o sistemas de expresión especialmente adaptados a E. coli.
- C12N15/74 C12N 15/00 […] › Vectores o sistemas de expresión especialmente adaptados a huéspedes procariotas distintos a E. coli, p. ej. Lactobacillus, Micromonospora.
Clasificación PCT:
- C12N15/62 C12N 15/00 […] › Secuencias de ADN que codifican proteínas de fusión.
Clasificación antigua:
- C12N15/62 C12N 15/00 […] › Secuencias de ADN que codifican proteínas de fusión.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
Fragmento de la descripción:
Apoyo gubernamental
El sistema de exportación de proteínas aquí definido fue desarrollado a través del apoyo de los proyectos 5 RO1 A129471, RO1 A140297, y Research Contract NO1 A145251 (M. M. Levine, investigador principal), del Instituto Nacional de Salud estadounidense (NIH). Aplicaciones relacionadas
Esta aplicación reivindica prioridad a la solicitud de patente provisional No. 60/252,51B, presentada el 22 de Noviembre del 2000. Antecedentes del invento
La descripción a continuación se refiere al uso de un sistema de exportación de proteínas. El sistema descrito proporciona métodos eficaces y composiciones útiles para la producción de proteínas recombinantes. Descripción de la técnica relacionada
Los sistemas de expresión de proteinas han usado durante mucho tiempo plásmidos de expresión o vectores de expresión de alto número de copia en un intento de aumentar los rendimientos de proteinas recombinantes de interés. Los plásmidos de expresión de alto número de copia y las proteínas de interés que codifican pueden ejercer un efecto negativo en la idoneidad de un hospedante que contiene un plásmido de expresión. La carga notable colocada en las células hospedantes procariotas que llevan plásmidos multicopia es el resultado acumulativo de una cascada metabólica desencadenada por dos procesos: 1) la replicación y el mantenimiento de los plásmidos de expresión y 2) transcripción y traducción de las diversas funciones codificadas por los plámidos incluyendo el gen de interés. Tales mecanismos podrían explicar la observación de que las bacterias que llevan plásmidos crecen más lento que las bacterias sin plásmidos. Esta carga puede también explicar la observación de que la tasa de crecimiento disminuye según el número de copia aumenta.
Según se expresa el gen de interés, la tasa de crecimiento de las células hospedantes recombinantes disminuye. La disminución de la tasa de crecimiento puede desencadenar la inducción de diversas proteasas celulares que pueden degradar la proteína producida de modo recombinante presente en el citoplasma de la célula hospedante. La tasa de recimiento reducida es por tanto la inevitable consecuencia de una carga metabólica, que a su vez el el resultado acumulativo de un número de perturbaciones fisiológicas. Debido a que esta reducción en la tasa de crecimiento crea una presión selectiva para la pérdida de los plásmidos residentes en ausencia de selección, la pérdida significativa de los plásmidos de expresión a partir de la célua hospedante que lleva un vector de expresión puede tener lugar tras la transformación de la célula hospedante.
Lás células hospedantes con tasa de crecmiento reducidas pueden despojarse de modo espontáneo de un plásmido de expresión para eliminar de la célula hospedante una carga metabólica innecesaria y permitir a las células hospedantes sin plásmido crecer más rápidamente que la población de células hospedantes que llevan plásmido. Tal cambio en la expresión de proteínas dentro de una población de células hospedantes se esperaría que redujera la producción proteica.
Por consiguiente, sería deseable preparar un sistema de expresión de proteínas que optimizaría la expresión de proteínas a partir del vector de expresión mientras que minimiza la carga metabólica en la célula hospedante generada por el vector de expresión. Sumario del invento
El material descrito se refiere al uso de una proteína de exportación para facilitar la exportación de una proteína de fusión fuera de la célula hospedante. Una realización descrita proporciona un método para expresar un gen en una célula bacteriana que comprende proporcionar un vector de expresión en una poblacicón de células hospedantes bacterianas, en la que el vector de expresión comprende un casete de expresión que comprende una secuencia que codifica una proteína de exporación genéticamente fusionada a una secuencia codificante para una proteína de interés, que expresa el casete de expresión tal que una proteína de fusión proteína de exportación–proteína de interés es producida y exportada o transportada al medio de cultivo, en el que la secuencia codificante para la proteína de exportación es seleccionada a partir del grupo que consiste en Salmonella enterica serovar Typhi (S. Typhi), el gen cytolisina A (clyA), gen de Salmonella paratyphi (S. paratyphi) clyA o el gen de hemolisina (hly E) de Escherichia coli (E. coli).
Otra realización descrita se refiere al uso de una población de células hospedantes bacterianas transformadas con un vecto de expresión que comprende un casete de expresión que comprende una secuencia codificante de proteína de exportación genéticamente fusionada a una secuencia que codifica una proteína de interés en la fabricación de un medicamento para obtener una respuesta inmune de un animal en el que la secuencia codificante de una proteína de exportación es seleccionada del grupo que consiste en el gen de citolisina A (clyA) de Salmonella enterica serovar Typhi (S. Thypi), el gen cly A de salmonella paratyphi (S. paratyphi)o el gen de hemolisina E (hly E) de Escherichia coli (E. coli).
Otra realización descrita se refiere a un sistema para expresar una proteína de interés que comprende: un vector de expresión que comprende un casete de expresión, en el que el casete de expersión comprende una secuencia codificante de la proteína de exportación genéticamente fusionada a una secuencia codificante de una proteína de interés, una célula hospedante transformada con el vector de expresión, y un medio de cultivo para la célula hospedante transformada, en el que el casete de expresión expresa una proteína de fusión proteína de expresión-proteína de interés, que es exportada hacia fuera de la célula hospedante transformada en el que la secuencia codificante de una proteína de exportación es seleccionada del grupo que consiste en el gen de citolisina A (clyA) de Salmonella enterica serovar Typhi (S. Thypi), el gen cly A de Salmonella paratyphi (S. paratyphi) o el gen de hemolisina E (hly E) de Escherichia coli (E. coli). Breve descripción de los dibujos
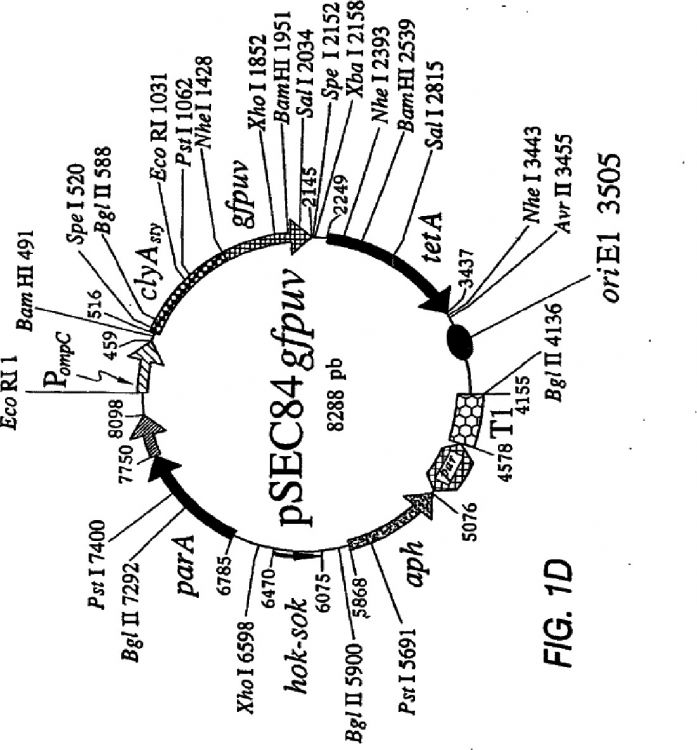
La Figura 1 proporciona ejemplos del vector de expresión de este invento. La Figura 1A ilustra el vector de expresión pSEC84. La Figura 1B ilustra el vector de expresión pSEC84bla . La figura 1C ilustra pSEC84sacB (SEQ. ID Nº: 18). La Figura 1D ilustra pSEC84gfpuv.
La Figura 2 ilustra la exportación de la proteína de fusión Cly-SacB que da como resultado la metabolisis de la sacarosa en medio de crecimiento sólido. Las cepas fueron hechas crecer en medio que contiene bien 8% de sacarosa (2A y 2B), 16% de sacarosa (2C y 2D), u 8% de sacarosa + 8% L-arabinosa (2E y 2F). Las Figuras 2A, 2C y 2E demuestran el crecimiento de CVD 908-htrA que expresa ClgA-SacB.
La Figura 3 ilustra el ceciminento de DVD 908-htrA que expresa bien ClgA (pSEC84) o ClgA-SacB (pSEC84sacB), hecho crecer en caldo de cultivo 2XLB50 suplementado con DHB y bien 10% de sacarosa ó 10% de glucosa.
La Figura 4 ilustra análisis de inmunotransferencia Western de fracciones celulares bacterianas de bien CVD 908-htrA (carriles 1-3) o CVD 908-htrA (pSEC84gfpuv) (carriles 4-8). Las fracciones celulares son cargadas como sigue: sobrenadantes, carriles 1 y 4; citoplásmicas, carriles 2 y 6; periplásmicas, carril 5; insoluble, carril 7: lisado celular total, carriles 3 y 8; y 50 ng GFPuv, carril 9. Las membranas con muestras idénticas fueron incubadas con anticuerpos específicos para GFPuv (panel A) o E. coli GroEL (panel B). Descrición detallada de la realización preferida
La descripción a continuación proporciona un sistema de exportación proteica para producir eficientemente proteínas recombinantes a partir de un hospedante bacteriano. En una realización preferida, el sistema de exportación proteica utiliza la maquinaria endógena de exportación proteica a la bacteria hospedante en la que el vector del sistema de exportación proteica es introducido.
El sistema de exportación de proteínas tiene un número de aplicaciones útiles. El sistema puede ser usado para producir de modo eficiente proteínas recombinantes de interés dentro de una célula hospedante bacteriana y exportar la proteína reocmbinante de interés de la célula hospedante bacteriana. Por ejemplo, el sistema descrito puede ser usado para producir de modo eficiente proteínas recombinantes de interés en un bioreactor.
El sistema de exportación de proteínas puede también...
Reivindicaciones:
Reinvindicaciones
1. Un método para expresar un gen en una célula bacteriana que comprende: proporcionar un vector de expresión para una población de células bacterianas
sin transformar, en el que el vector de expresión comprende un casete de expresión que comprende una secuencia que codifica una proteína de exportación fusionada genéticamente a una secuencia que codifica una proteína de interés;
expresar el casete de expresión tal que una proteína de fusión proteína de exportación::proteína de interés es producida y exportada en el medio de cultivo, en el que la la secuencia que codifica una proteína de exportación es seleccionada del grupo que consiste en gen (clyA) citolisina A de Salmonella serovar entérica Typhi (S. Typhi), gen clyA (S. paratyphi) de Salmonella paratyphi o el gen hemolisina E (hlyE) de Eschericia coli (E. coli).
2. El método de la reivindicación 1, en el que dicha secuencia que codifica la proteína de exportación es seleccionada del grupo que consiste en la secuencia de nucleótidos 516-1436 de la SEQ ID No. 1 que codifica la proteína clyA de S. Typhi y la secuencia que codifica la secuencia aminoácida de la SEQ ID No:2.
3. El método de la reivindicación 1 ó 2, en el que dicha célula bacteriana es una célula S. Typhi o una célula E. coli.
4. El método de cualquiera de las reivindicaciones 1 a 3, en el que la secuencia que codifica la proteína de exportación es la secuencia de nucleótidos 5161436 de la SEQ ID No. 1 que codifica la proteína clyA de S. Typhi.
5. El método de cualquiera de las reivindicaciones 1 a 4, en el que la secuencia que codifica la proteína de exportación es a) la secuencia de nucleótidos 516-1436 de la SEQ ID No. 1 que codifica la proteína clyA de S. Typhi o b) codifica la secuencia aminoácida de SEQ ID No: 2, con una o más mutaciones que dan como resultado sustituciones de aminoácidos seleccionados del grupo que consiste en sustituciones aminoácidos en las posiciones 180, 185, 187 y 193, que atenúan la actividad hemolítica de la proteína de exportación.
6. El método de cualquiera de las reivindicaciones 1 a 5, en el que la proteína de interés es un antígeno.
7. Un método como se reivindica en cualquiera de las reivindicaciones de 1 a 6, en el que dicha proteína de fusión exportada es aislada.
8. El uso de una población de células bacterianas transformadas con un vector de expresión que comprende un casete de expresión que comprende una secuencia que codifica la proteína de exportación fusionada genéticamente a una secuencia que codifica una proteína de interés en la fabricación de un medicamento para obtener una respuesta inmune de un hospedante, en el que la secuencia que codifica la proteína de exportación es seleccionada del grupo que consiste en gen citolisina A (clyA) de Salmonella serovar entérica Typhi (S. Typhi), gen clyA de Salmonella paratyphi (S. paratyphi) o el gen hemolisina E (hlyE) de (E. coli) Escherichia coli.
9. Un uso como se reivindica en la reivindicación 8, en el que dicha célula bacteriana, la secuencia que codifica la proteína de exportación y/o la proteína de interés es como se define en cualquiera de las reivindicaciones 2 a 6.
10. Un sistema para expresar una proteína de interés que comprende:
un vector de expresión que comprende un casete de expresión, en el que el casete de expresión comprende una secuencia que codifica la proteína de exportación fusionada genéticamente a una secuencia que codifica una proteína de interés;
una célula hospedante transformada con el vector de expresión; y
un entorno de cultivo para la célula hospedante transformada, en el que el casete de expresión cuando se expresa da como resultado una proteína de fusión proteína de exportación::proteína de interés, que es exportada fuera de la célula hospedante transformada, en el que la proteína de exportación que codifica la secuencia es seleccionada del grupo que consiste en gen citolisina A(clyA) de Salmonella serovar entérica Typhi (S. Typhi), gen clyA de Salmonella paratyphi (S. paratyphi) o el gen hemolisina E (hlyE) de Escherichia coli (E. coli).
11. Un sistema como se reivindica en la reivindicación 10, en el que dicha célula bacteriana, la secuencia que codifica la proteína de exportación y/o la proteína de interés es como se define en cualquiera de las reivindicaciones 2 a 6.
Patentes similares o relacionadas:
Sistema de expresión bacteriano cistrónico doble, del 25 de Marzo de 2020, de LUPIN LIMITED: Un procedimiento para la producción de un anticuerpo o un fragmento del mismo que comprende las etapas de: (i) transformar una célula hospedadora bacteriana […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 15 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de células bacterianas para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no replicativa (NRTP) para su introducción […]
Método de construcción de biblioteca de péptidos y vectores relacionados, del 1 de Enero de 2020, de Hunan Zonsen Peplib Biotech Co., Ltd: Método integrado de construcción de una biblioteca de péptidos completa que contiene todos los péptidos posibles para el tamaño específico, comprendiendo el método: (i) […]
 Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Transporte de proteínas basado en bacterias, del 14 de Agosto de 2019, de Universität Basel: Un vector que comprende, en la dirección 5' a 3': un promotor; una primera secuencia de ADN que codifica una señal de transporte procedente […]
Sistema de expresión y secreción, del 26 de Junio de 2019, de F. HOFFMANN-LA ROCHE AG: Una molécula de ácido nucleico que codifica un primer polipéptido y un segundo polipéptido, enlazada de forma funcional a una secuencia señal que codifica […]
 Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Síntesis microbiana de aldehídos y alcoholes correspondientes, del 17 de Junio de 2019, de Easel Biotechnologies, LLC: Un método para producir un alcohol, que comprende: cultivar una pluralidad de células microbianas en un medio de fermentación, en donde las […]