GRUPOS DE PROTEINAS REPETITIVAS QUE COMPRENDEN MODULOS REPETITIVOS.
Grupo de moléculas de ácidos nucleicos codificantes de un grupo de proteínas repetitivas,
comprendiendo cada proteína repetitiva un dominio repetitivo, que comprende un conjunto de módulos repetitivos consecutivos y un módulo de caperuza N-terminal y/o C-terminal que presenta una secuencia de aminoácidos diferente de cualquiera de dichos módulos repetitivos, en el que dichos módulos repetitivos presentan el mismo plegamiento y se apilan estrechamente para crear una estructura superhelicoidal que presenta un núcleo hidrofóbico común, en el que cada uno de dichos módulos repetitivos se deriva de una o más unidades repetitivas de una familia de proteínas repetitivas naturales, en el que dichas unidades repetitivas comprende residuos esqueléticos y residuos de interacción diana, en el que dichas proteínas repetitivas difieren en por lo menos una posición aminoácida de un residuo de interacción diana de los módulos repetitivos, y en el que dicha derivación de cada uno de dichos módulos repetitivos se lleva a cabo mediante un procedimiento que comprende las etapas siguientes:
(a) identificar dichas unidades repetitivas,
(b) determinar un motivo de secuencia repetitiva mediante análisis estructural y de secuencias de dichas unidades repetitivas, en el que dicho motivo de secuencia repetitiva comprende posiciones de residuo esquelético y posiciones de residuo de interacción diana, que corresponden a las posiciones de los residuos esqueléticos y los residuos de interacción diana en dichas unidades repetitivas, y
(c) construir el módulo repetitivo, de manera que comprende el motivo de secuencia repetitiva de (b)
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP01/10454.
Solicitante: UNIVERSITAT ZURICH.
Nacionalidad solicitante: Suiza.
Dirección: RAMISTRASSE 71,8006 ZURICH.
Inventor/es: PLUCKTHUN, ANDREAS, STUMPP,MICHAEL,TOBIAS, FORRER,PATRICK, BINZ,HANS,KASPAR.
Fecha de Publicación: .
Fecha Concesión Europea: 11 de Noviembre de 2009.
Clasificación Internacional de Patentes:
- C07K14/47 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › de mamíferos.
- C07K14/47A1A
- C12N15/10C3
Clasificación PCT:
- A61K38/16 NECESIDADES CORRIENTES DE LA VIDA. › A61 CIENCIAS MEDICAS O VETERINARIAS; HIGIENE. › A61K PREPARACIONES DE USO MEDICO, DENTAL O PARA EL ASEO (dispositivos o métodos especialmente concebidos para conferir a los productos farmacéuticos una forma física o de administración particular A61J 3/00; aspectos químicos o utilización de substancias químicas para, la desodorización del aire, la desinfección o la esterilización, vendas, apósitos, almohadillas absorbentes o de los artículos para su realización A61L; composiciones a base de jabón C11D). › A61K 38/00 Preparaciones medicinales que contienen péptidos (péptidos que contienen ciclos beta-lactama A61K 31/00; dipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina 2,5-dionas, A61K 31/00; péptidos basados en la ergolina A61K 31/48; que contienen compuestos macromoleculares que tienen unidades aminoácido repartidas estadísticamente A61K 31/74; preparaciones medicinales que contienen antígenos o anticuerpos A61K 39/00; preparaciones medicinales caracterizadas por los ingredientes no activos, p. ej. péptidos como soportes de fármacos, A61K 47/00). › Péptidos que tienen más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados.
- C07K14/435 C07K 14/00 […] › de animales; de humanos.
- C07K14/47 C07K 14/00 […] › de mamíferos.
- C12N15/10 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/63 C12N 15/00 […] › Introducción de material genético extraño utilizando vectores; Vectores; Utilización de huéspedes para ello; Regulación de la expresión.
Clasificación antigua:
- C07K14/00 C07K […] › Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados.

Fragmento de la descripción:
Grupos de proteínas repetitivas que comprenden módulos repetitivos.
La presente invención se refiere a grupos de proteínas repetitivas que comprenden módulos repetitivos que se derivan de una o más unidades repetitivas de una familia de proteínas repetitivas naturales, a grupos de moléculas de ácidos nucleicos codificantes de dichas proteínas repetitivas, a métodos para la construcción y aplicación de dichos grupos y a miembros individuales de dichos grupos.
Se citan varios documentos en la presente memoria. El contenido de la exposición de estos documentos se incorpora en la presente memoria como referencia.
Las interacciones proteína-proteína, o más generalmente, las interacciones proteína-ligando, desempeñan un papel importante en todos los organismos, y la compresión de las características clave del reconocimiento y la unión es un tema principal de la investigación bioquímica actual. Hasta el momento, los anticuerpos y cualquiera de los derivados que se han preparado se utilizan principalmente en este campo de investigación. Sin embargo, la tecnología de los anticuerpos adolece de desventajas bien conocidas. Por ejemplo, los anticuerpos difícilmente pueden aplicarse intracelularmente debido al ambiente reductor del citoplasma. De esta manera, existe una necesidad de moléculas ligantes de alta afinidad con características que superen la restricción de los anticuerpos. Este tipo de moléculas muy probablemente proporcionará nuevas soluciones en los campos de la medicina, biotecnología e investigación, en los que los ligantes intracelulares también crecerán en importancia en la genómica.
Se ha informado de diversos esfuerzos para construir nuevas proteínas ligantes (revisados en Nygren y Uhlen, 1997). La estrategia más prometedora aparentemente es una combinación de la generación limitada de una biblioteca y el cribado o selección para las propiedades deseadas. Habitualmente se reclutan andamiajes existentes para aleatorizar algunos residuos aminoácidos expuestos tras el análisis de la estructura cristalina. Sin embargo, a pesar de los avances conseguidos en términos de estabilidad y expresabilidad, las afinidades de las que se ha informado hasta el momento son considerablemente inferiores a las de los anticuerpos (Ku y Schultz, 1995). Una restricción podría ser la limitación a dianas para las que se conoce la estructura cristalina (Kirkham et al., 1999) o que son homólogas a la molécula diana original, de manera que hasta el momento no se ha identificado ningún andamiaje universal para la unión. Para incrementar la afinidad aparente de los ligantes tras el cribado, varios enfoques han utilizado la multimerización de ligantes individuales para aprovechar los efectos de avidez.
La patente WO nº 00/34784 describe proteínas que incluyen un dominio de fibronectina de tipo III que presenta por lo menos un bucle aleatorizado y la utilización de las mismas para desarrollar nuevas especies ligantes de compuesto.
La patente WO nº 96/06166 propone bibliotecas de secuencia de ADN que codifican motivos ligantes dedos de zinc para la expresión en una partícula que puede utilizarse para diseñar polipéptidos ligantes de dedo de zinc específicos para una secuencia diana particular.
Arndt (J. Mol. Biol. 295:627-639, 2000) informan de la generación de un péptido heterodimérico helicoidal enrollado que se seleccionó de un ensamblaje diseñado de biblioteca-versus-biblioteca derivado de secuencias de cremallera de leucinas.
De esta manera, el problema técnico subyacente a la presente invención es identificar nuevos enfoques para la construcción de grupos de proteínas de unión.
La solución a dicho problema técnico se consigue proporcionando las realizaciones caracterizadas en las reivindicaciones. De acuerdo con lo anterior, la presente invención permite construir grupos de proteínas repetitivas que comprenden módulos repetitivos. El enfoque técnico de la presente invención, es decir, derivar dichos módulos a partir de unidades repetitivas de proteínas repetitivas naturales, no ha sido proporcionado ni sugerido por la técnica anterior.
De esta manera, la presente invención se refiere a grupos de moléculas de ácidos nucleicos codificantes de grupos de proteínas repetitivas, comprendiendo cada proteína repetitiva un dominio repetitivo, que comprende un conjunto de módulos repetitivos consecutivos y un módulo de caperuza N-terminal y/o C-terminal que presenta una secuencia de aminoácidos diferente de cualquiera de dichos módulos repetitivos, en la que dichos módulos repetitivos presentan el mismo plegamiento y se apilan estrechamente para crear una estructura superhelicoidal que presenta un núcleo hidrofóbico común, en el que cada uno de dichos módulos repetitivos se deriva a partir de una o más unidades repetitivas de una familia de proteínas repetitivas naturales, en la que dichas unidades repetitivas comprenden residuos estructurales y residuos de interacción diana, en los que dichas proteínas repetitivas difieren en por lo menos una posición aminoácida de un residuo diana de interacción de los módulos repetitivos, y en los que dicha derivación de cada uno de dichos módulos repetitivos se lleva a cabo mediante un procedimiento que comprende las etapas siguientes:
En el contexto de la presente invención, el término grupo
se refiere a una población que comprende por lo menos dos entidades o miembros diferentes. Preferentemente, este grupo comprende por lo menos 105, más preferentemente más de 107, y todavía más preferentemente más de 109 miembros diferentes. También puede hacerse referencia a grupo
como biblioteca
o pluralidad
.
La expresión molécula de ácidos nucleicos
se refiere a una molécula polinucleótida, que es una molécula de ácido ribonucleico (ARN) o de ácido desoxirribonucleico (ADN), de una cadena o de doble cadena. Una molécula de ácidos nucleicos puede encontrarse presente en forma aislada, o estar comprendida de moléculas o vectores de ácidos nucleicos recombinantes.
La expresión proteína repetitiva
se refiere a un (poli)péptido/proteína que comprende uno o más dominios repetitivos (fig. 1). Preferentemente, cada una de dichas proteínas repetitivas comprende hasta cuatro dominios repetitivos. Más preferentemente, cada una de dichas proteínas repetitivas comprende hasta dos dominios repetitivos. Sin embargo, más preferentemente, cada una de las proteínas repetitivas comprende un dominio repetitivo. Además, dicha proteína repetitiva puede comprender dominios proteicos no repetitivos adicionales (figs. 2a y 2b), etiquetas (poli)péptido y/o secuencias (poli)peptídicas línker (fig. 1). La expresión etiqueta (poli)péptido
se refiere a una secuencia de aminoácidos unida a un (poli)péptido/proteína, en la que dicha secuencia de aminoácidos es utilizable para la purificación, detección o reconocimiento de dicho (poli)péptido/proteína, o en la que dicha secuencia de aminoácidos mejora el comportamiento fisicoquímico de dicho (poli)péptido/proteína, o en el que dicha secuencia de aminoácidos presenta una función efectora. Dichas etiquetas (poli)péptido pueden ser secuencias polipeptídicas pequeñas, por ejemplo Hisn (Hochuli et al., 1988; Lindner et al., 1992), myc, FLAG (Hopp et al., 1988; Knappik y Pluckthun, 1994) o Strep-tag (Schmidt y Skerra, 1993; Schmidt y Skerra, 1994; Schmidt et al., 1996). Estas etiquetas (poli)péptido son bien conocidas de la técnica y se encuentran totalmente disponibles para el experto en la materia. Otros dominios no repetitivos pueden ser grupos adicionales, tales como enzimas (por ejemplo enzimas como la fosfatasa alcalina), que permiten la detección de dichas proteínas repetitivas, o grupos que pueden utilizarse para el direccionamiento (tales como inmunoglobulinas o fragmentos...
Reivindicaciones:
1. Grupo de moléculas de ácidos nucleicos codificantes de un grupo de proteínas repetitivas, comprendiendo cada proteína repetitiva un dominio repetitivo, que comprende un conjunto de módulos repetitivos consecutivos y un módulo de caperuza N-terminal y/o C-terminal que presenta una secuencia de aminoácidos diferente de cualquiera de dichos módulos repetitivos, en el que dichos módulos repetitivos presentan el mismo plegamiento y se apilan estrechamente para crear una estructura superhelicoidal que presenta un núcleo hidrofóbico común, en el que cada uno de dichos módulos repetitivos se deriva de una o más unidades repetitivas de una familia de proteínas repetitivas naturales, en el que dichas unidades repetitivas comprende residuos esqueléticos y residuos de interacción diana, en el que dichas proteínas repetitivas difieren en por lo menos una posición aminoácida de un residuo de interacción diana de los módulos repetitivos, y en el que dicha derivación de cada uno de dichos módulos repetitivos se lleva a cabo mediante un procedimiento que comprende las etapas siguientes:
2. Grupo según la reivindicación 1, en el que dicho motivo repetitivo de la etapa (b) contiene por lo menos una posición aleatorizada correspondiente a una posición de un residuo de interacción diana de dichas unidades repetitivas.
3. Grupo según la reivindicación 1 ó 2, en el que dicho análisis de secuencia de la etapa (b) implica la provisión de una secuencia de consenso mediante la alineación de las secuencias de aminoácidos de dichas unidades repeti- tivas.
4. Grupo según cualquiera de las reivindicaciones 1 a 3, en el que cada uno de dichos módulos repetitivos presenta una secuencia de aminoácidos en la que por lo menos 70% de los residuos aminoácidos corresponden a:
(i) los residuos aminoácidos de consenso deducidos a partir de los residuos aminoácidos presentes en las posiciones correspondientes de por lo menos dos unidades repetitivas naturales, o
(ii) los residuos aminoácidos presentes en las posiciones correspondientes en una unidad repetitiva natural.
5. Grupo según cualquiera de las reivindicaciones 1 a 4, en el que dicho conjunto consiste de entre dos y aproximadamente 30 módulos repetitivos.
6. Grupo según cualquiera de las reivindicaciones 1 a 5, en el que dichos módulos repetitivos se encuentran conectados directamente.
7. Grupo según cualquiera de las reivindicaciones 1 a 6, en el que dichos módulos repetitivos se encuentran conectados mediante un línker (poli)péptido.
8. Grupo según cualquiera de las reivindicaciones 1 a 7, en el que dichas unidades repetitivas son repeticiones anquirina.
9. Grupo según la reivindicación 8, en el que cada uno de dichos módulos repetitivos comprende el motivo de secuencia repetitiva anquirina DxxGxTPLHLAaxxttx
se refiere a cualquier aminoácido,
se refiere a cualquier aminoácido o a una deleción, a
se refiere a un aminoácido con una cadena lateral apolar, y p
se refiere a un residuo con una cadena lateral polar.
10. Grupo según la reivindicación 8, en el que cada uno de dichos módulos repetitivos comprende el motivo de secuencia repetitiva anquirina DxxGxTPLHLAxxxGxxxVVxLLLxxGADVNAx, en el que x
se refiere a cualquier aminoácido.
11. Grupo según la reivindicación 8, en el que cada uno de dichos módulos repetitivos comprende el motivo de secuencia repetitiva anquirina DxxGxTPLHLAxxxGxxxIVxVLLxxGADVNAx, en el que x
se refiere a cualquier aminoácido.
12. Grupo según cualquiera de las reivindicaciones 9 a 11, en el que una o más de las posiciones denominadas x
se encuentra aleatorizada.
13. Grupo según la reivindicación 8, en el que cada uno de dichos módulos repetitivos comprende el motivo de secuencia repetitiva anquirina siguiente: D11G1TPLHLAA11GHLEIVEVLLK2GADVNA1, en la que 1 representa un residuo aminoácido seleccionado de entre el grupo siguiente: A, D, E, F, H, I, K, L, M, N, Q, R, S, T, V, W e Y, en el que 2 representa un residuo aminoácido seleccionado de entre el grupo siguiente: H, N e Y.
14. Grupo según cualquiera de las reivindicaciones 1 a 7, en el que dichas unidades repetitivas son repeticiones ricas en leucinas (LRR).
15. Grupo según la reivindicación 14, en el que cada uno de dichos módulos comprende el motivo de secuencia LRR siguiente: xLxxLxLxxN+xaxx+a++++a++ax
se refiere a cualquier aminoácido, a
se refiere a un aminoácido alifático y
se refiere a cualquier aminoácido o a una deleción.
16. Grupo según la reivindicación 14, en el que por lo menos uno de dichos módulos comprende el motivo de secuencia LRR siguiente: xLExLxLxxCxLTxxxCxxLxxaLxxxx, en el que x
se refiere a cualquier aminoácido y a
se refiere a un aminoácido alifático (LRR de tipo A).
17. Grupo según la reivindicación 14, en el que por lo menos uno de dichos módulos comprende el motivo de secuencia LRR siguiente: xLxELxLxxNxLGDxGaxxLxxxLxxPxx, en el que x
se refiere a cualquier aminoácido y a
se refiere a un aminoácido alifático (LRR de tipo B).
18. Grupo según cualquiera de las reivindicaciones 15 a 17, en el que una o más de las posiciones denominadas x
y/o
se ha aleatorizado.
19. Grupo según la reivindicación 16, en el que el residuo cisteína en la posición 10 en la secuencia de consenso LRR de tipo A se ha sustituido por un residuo aminoácido hidrofílico, y en el que el residuo cisteína en la posición 17 se ha sustituido por un residuo aminoácido hidrofóbico.
20. Grupo según cualquiera de las reivindicaciones 9 a 13 ó 15 a 19, en el que uno o más de los residuos aminoácidos en dichas secuencias de consenso se ha intercambiado por un residuo aminoácido presente en la posición correspondiente en una unidad repetitiva natural correspondiente.
21. Grupo según cualquiera de las reivindicaciones 1 a 20, en el que dicho conjunto consiste de un tipo de módulo repetitivo.
22. Grupo según cualquiera de las reivindicaciones 1 a 20, en el que dicho conjunto consiste de dos tipos diferentes de módulos repetitivos.
23. Grupo según la reivindicación 21, en el que dicho conjunto comprende dos tipos diferentes de módulos repetitivos consecutivos como parejas en dicho dominio repetitivo.
24. Grupo según la reivindicación 22 ó 23, en el que dichos dos tipos diferentes de módulos están basados en dichas LRR de tipo A y LRR de tipo B.
25. Grupo según cualquiera de las reivindicaciones 21 a 24, en el que las secuencias de aminoácidos de los módulos repetitivos comprendidos en dicho conjunto son idénticas para cada uno de dichos tipos, excepto para los residuos aleatorizados.
26. Grupo según la reivindicación 25, en el que las secuencias de ácidos nucleicos codificantes de las copias de cada uno de dichos tipos son idénticas excepto para los codones codificantes de residuos aminoácidos en posiciones que se aleatorizan.
27. Grupo según cualquiera de las reivindicaciones 1 a 26, en el que dichas moléculas de ácidos nucleicos comprenden secuencias de ácidos nucleicos idénticas de por lo menos 9 nucleótidos entre dichos módulos repetitivos.
28. Grupo según la reivindicación 23 ó 24, en el que dichas moléculas de ácidos nucleicos comprenden secuencias de ácidos nucleicos idénticas de por lo menos 9 nucleótidos entre dichas parejas.
29. Grupo según cualquiera de las reivindicaciones 1 a 27, en el que cada una de las secuencias de ácidos nucleicos entre dichos módulos, o dichas parejas, comprende una secuencia de reconocimiento de enzima de restricción.
30. Grupo según cualquiera de las reivindicaciones 1 a 28, en el que cada una de las secuencias de ácidos nucleicos entre dichos módulos, o dichas parejas, comprende una secuencia de ácidos nucleicos formada a partir de extremos cohesivos creados por dos enzimas de restricción compatibles.
31. Grupo según la reivindicación 27 ó 28, en el que dichas secuencias de ácidos nucleicos idénticas permiten un ensamblaje basado en PCR de dichas moléculas de ácidos nucleicos.
32. Grupo según la reivindicación 25, en el que dicho dominio repetitivo comprende una o más parejas de módulos basadas en dichas LRR de tipo A y de tipo B, en las que cada una de dichas parejas presenta la secuencia siguiente:
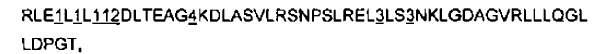
en la que 1 representa un residuo aminoácido seleccionado de entre el grupo siguiente: D, E, N, Q, S, R, K, W e Y,
en la que 2 representa un residuo aminoácido seleccionado de entre el grupo siguiente: N, S y T,
en la que 3 representa un residuo aminoácido seleccionado de entre el grupo siguiente: G, S, D, N, H y T, y
en la que 4 representa un residuo aminoácido seleccionado de entre el grupo siguiente: L, V y M.
33. Grupo según la reivindicación 32, en el que cada una de dichas parejas de módulos está codificada por la molécula de ácidos nucleicos siguiente:

en la que 111 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: D, E, N, Q, S, R, K, W e Y,
en la que 222 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: N, S y T,
en la que 333 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: G, S, D, N, H y T, y
en la que 444 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: L, V y M.
34. Grupo según la reivindicación 32, en el que uno o más de los residuos aminoácidos en por lo menos una de dichas parejas de módulos se ha intercambiado por un residuo aminoácido presente en la posición correspondiente en una LRR natural.
35. Grupo según la reivindicación 33, en el que uno o más de los codones de aminoácido en por lo menos una de dichas parejas de módulos se ha intercambiado por un codón codificante de un residuo aminoácido presente en la posición correspondiente en una LRR natural.
36. Grupo de moléculas de ácidos nucleicos recombinantes que comprende un grupo de moléculas de ácidos nucleicos según cualquier de las reivindicaciones 1 a 35.
37. Grupo de vectores que comprende un grupo de moléculas de ácidos nucleicos según cualquiera de las reivindicaciones 1 a 35, o un grupo de moléculas de ácidos nucleicos recombinantes según la reivindicación 36.
38. Grupo de células huésped que comprende un grupo de moléculas de ácidos nucleicos según cualquiera de las reivindicaciones 1 a 35, un grupo de moléculas de ácidos nucleicos recombinantes según la reivindicación 36, o un grupo de vectores según la reivindicación 37.
39. Grupo de proteínas repetitivas codificadas por un grupo de moléculas de ácidos nucleicos según cualquiera de las reivindicaciones 1 a 35, por un grupo de moléculas de ácidos nucleicos recombinantes según la reivindicación 36, por un grupo de vectores según la reivindicación 37, o producidas por un grupo de células huésped según la reivindicación 38.
40. Método para la construcción de un grupo de moléculas de ácidos nucleicos según cualquiera de las reivindicaciones 1 a 35, que comprende las etapas siguientes:
41. Método según la reivindicación 40, en el que dicho módulo o módulos repetitivos deducidos en la etapa (c) presenta una secuencia de aminoácidos, en la que por lo menos 70% de los residuos aminoácidos corresponden a:
42. Método para la producción de un grupo de proteínas repetitivas según la reivindicación 39, que comprende las etapas siguientes:
43. Método para la obtención de una proteína repetitiva que presenta una propiedad predeterminada, que comprende las etapas siguientes:
44. Método según la reivindicación 43, en el que dicha propiedad predeterminada es la unión a una diana.
45. Proteína repetitiva de un grupo según cualquiera de las reivindicaciones 1 a 35, en la que dicha proteína repetitiva presenta una propiedad predeterminada.
46. Molécula de ácidos nucleicos codificante de la proteína repetitiva según la reivindicación 45.
47. Vector que contiene la molécula de ácidos nucleicos según la reivindicación 46.
48. Composición farmacéutica que comprende la proteína repetitiva según la reivindicación 45 o la molécula de ácidos nucleicos según la reivindicación 46, y opcionalmente un portador y/o diluyente farmacéuticamente aceptable.
49. Molécula de ácidos nucleicos codificante de una pareja de módulos repetitivos para la construcción de un grupo según la reivindicación 32 ó 33, en la que dicha molécula de ácidos nucleicos es:

en la que 111 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: D, E, N, Q, S, R, K, W e Y,
en la que 222 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: N, S y T,
en la que 333 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: G, S, D, N, H y T, y
en la que 444 representa un codón codificante de un residuo aminoácido seleccionado de entre el grupo siguiente: L, V y M.
Patentes similares o relacionadas:
Polipéptidos de unión específica novedosos y usos de los mismos, del 15 de Julio de 2020, de Pieris Pharmaceuticals GmbH: Muteína de lipocalina lagrimal humana que tiene especificidad de unión para IL-17A, en la que la muteína se une a IL-17A con una KD de aproximadamente 1 nM o menos, en la que […]
Biomarcador de enfermedad autoinmunitaria, del 15 de Julio de 2020, de Tzartos, Socrates: Un método de diagnóstico o pronóstico de una enfermedad autoinmunitaria asociada con la formación de lesiones desmielinizadas del sistema nervioso central (SNC) […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Composiciones útiles en el tratamiento de la deficiencia de ornitina transcarbamilasa (OTC), del 8 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Un vector vírico recombinante que comprende una secuencia de ácido nucleico que codifica la proteína ornitina transcarbamilasa humana (hOTC) y secuencias […]
Procedimientos y composiciones para el tratamiento de una afección genética, del 24 de Junio de 2020, de Sangamo Therapeutics, Inc: Una célula precursora de glóbulos rojos genomanipulada caracterizada por una modificación genómica dentro del exón 2 o el exón 4 de BCL11A o dentro de BCL11A-XL […]
Reactivos SIRP-alfa de alta afinidad, del 24 de Junio de 2020, de THE BOARD OF TRUSTEES OF THE LELAND STANFORD JUNIOR UNIVERSITY: Un polipéptido SIRPα de alta afinidad que comprende al menos una y no más de 15 modificaciones de aminoácidos dentro del dominio d1 de una secuencia SIRPα de tipo […]
Inmunoterapia WT1 para enfermedad angiogénica intraocular, del 17 de Junio de 2020, de INTERNATIONAL INSTITUTE OF CANCER IMMUNOLOGY, INC.: Una composición farmacéutica que comprende un péptido WT1 o péptido WT1 variante para su uso en el tratamiento y/o prevención de una enfermedad […]
Estructuras artificiales de poliepítopos para uso en inmunoterapia, del 17 de Junio de 2020, de Invectys: Un vector de expresión de ADN o una mezcla de vectores de expresión de ADN que codifica al menos dos epítopos de CD4 de la transcriptasa inversa de la telomerasa […]