BIBLIOTECAS COMBINATORIAS DE DOMINIOS MONOMERICOS.
Un método para identificar un multímero que se una a una molécula diana,
comprendiendo los métodos, (a) proporcionar una biblioteca de polipéptidos, comprendiendo los polipéptidos diferentes dominios monoméricos, donde cada dominio monomérico tiene 30-100 aminoácidos, comprende al menos dos enlaces disulfuro, y es un dominio de clase A del receptor de LDL; (b) escrutar la biblioteca de polipéptidos en busca de la afinidad para una molécula diana; (c) identificar al menos un primer polipéptido que comprende un primer dominio monomérico que se une específicamente a una molécula diana; (d) conectar el primer dominio monomérico a una pluralidad de diferentes dominios monoméricos para formar una biblioteca de diferentes multímeros, comprendiendo los multímeros el primer dominio monomérico y uno de una pluralidad de diferentes dominios monoméricos; (e) escrutar la biblioteca de multímeros en busca de la capacidad para unirse a la molécula diana; y (f) identificar un multímero que se une específicamente a la molécula diana, donde el multímero comprende el primer dominio monomérico y un segundo dominio monomérico
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2002/013257.
Solicitante: AMGEN MOUNTAIN VIEW INC.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 1209 ORANGE STREET WILMINGTON DE 19801 ESTADOS UNIDOS DE AMERICA.
Inventor/es: STEMMER, WILLEM, P., C., KOLKMAN,JOOST,A.
Fecha de Publicación: .
Fecha Solicitud PCT: 26 de Abril de 2002.
Fecha Concesión Europea: 30 de Junio de 2010.
Clasificación PCT:
- C07K16/00 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › Inmunoglobulinas, p. ej. anticuerpos mono o policlonales.
- C12P21/06 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 21/00 Preparación de péptidos o de proteínas (proteína monocelular C12N 1/00). › preparados por hidrólisis de un enlace peptídico, p. ej. hidrolizados.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
Clasificación antigua:
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
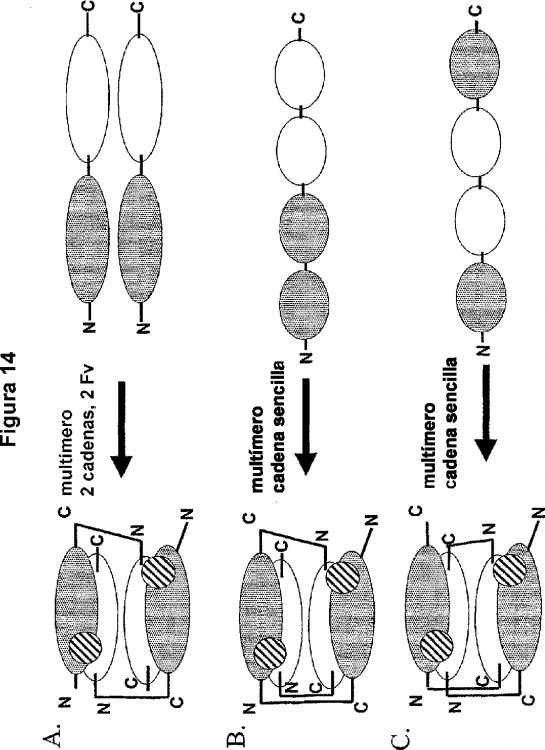
Fragmento de la descripción:
Bibliotecas combinatorias de dominios monoméricos.
Antecedentes de la invención
Los análisis de las secuencias de proteínas y de las estructuras tridimensionales han revelado que muchas proteínas están compuestas por un número discreto de dominios monoméricos. La mayoría de las proteínas con dominios monoméricos discretos es extracelular o constituye las porciones extracelulares de las proteínas unidas a la membrana.
Una importante característica de un dominio monomérico discreto es su capacidad para plegarse independientemente o con alguna ayuda limitada. La ayuda limitada puede incluir la ayuda de una o varias chaperoninas (p. ej., una proteína asociada con un receptor (RAP)). La presencia de uno o varios iones metálicos también ofrece ayuda limitada. La capacidad para plegarse independientemente evita el plegamiento erróneo del dominio cuando es insertado en el entorno de una nueva proteína. Esta característica ha permitido que los dominios monoméricos discretos sean evolutivamente móviles. Como resultado, los dominios discretos se han dispersado durante la evolución y ahora existen por otra parte en proteínas no relacionadas. Algunos dominios, incluyendo los dominios de tipo III de fibronectina y el dominio de tipo inmunoglobina, existen en numerosas proteínas, mientras otros dominios solamente se encuentran en un número limitado de proteínas.
Las proteínas que contienen estos dominios están involucradas en una variedad de procedimientos, tales como transportadores celulares, movimiento de colesterol, transducción de la señal y funciones de señalización que están implicadas en el desarrollo y la neurotransmisión. Véase Herz, Lipoprotein receptors: beacons to neurons?, (2001) Trends in Neurosciences 24(4):193-195; Goldstein y Brown, The Cholesterol Quartet, (2001) Science 292: 1310-1312. La función de un dominio monomérico discreto a menudo es específica pero también contribuye a la actividad global de la proteína o polipéptido. Por ejemplo, el dominio de clase A del receptor de LDL (también referido como módulo de clase A, una repetición de tipo complemento o un dominio A) está implicado en la unión al ligando mientras el dominio ácido gamma-carboxiglutámico (Gla) que se encuentra en las proteínas de coagulación dependientes de vitamina K está implicado en la unión de alta afinidad a las membranas de fosfolípidos. Otros dominios monoméricos discretos incluyen, p. ej., el dominio de tipo factor de crecimiento epidérmico (EGF) en el activador de plasminógeno de tipo tisular que media la unión a células del hígado y regula de ese modo el aclaramiento de esta enzima fibrinolítica de la circulación y la cola citoplásmica del receptor de LDL que está implicada en la endocitosis mediada por receptores.
Las proteínas individuales pueden poseer uno o más dominios monoméricos discretos. Estas proteínas a menudo se denominan proteínas mosaico. Por ejemplo, los miembros de la familia del receptor de LDL contienen cuatro dominios estructurales principales: las repeticiones del dominio A ricas en cisteína, las repeticiones de tipo precursor del factor de crecimiento epidérmico, un dominio transmembrana y un dominio citoplásmico. La familia del receptor de LDL incluye miembros que: 1) son receptores de la superficie celular; 2) reconocen ligandos extracelulares; y 3) los internalizan para su degradación por lisosomas. Véase Hussain et al., The Mammalian Low-Density Lipoprotein Receptor Family, (1999) Annu. Rev. Nutr. 19:141-72. Por ejemplo, algunos miembros incluyen receptores de lipoproteínas de muy baja densidad (VLDL-R), receptor 2 de apolipoproteína E, proteína relacionada con LDLR (LRP) y megalina. Los miembros de la familia tienen las siguientes características: 1) expresión en la superficie celular; 2) unión al ligando extracelular que consiste en repeticiones del dominio A; 3) requerimiento de calcio para la unión al ligando; 4) reconocimiento de la proteína asociada al receptor y la apolipoproteína (apo) E; 5) dominio de homología con el precursor del factor de crecimiento epidérmico (EGF) que contiene repeticiones YWTD; 6) región que abarca la membrana sencilla; y 7) endocitosis mediada por el receptor de diferentes ligandos. Véase Hussain, supra. Con todo, los miembros se unen a diversos ligandos estructuralmente distintos.
Resulta ventajoso desarrollar métodos para generar y optimizar las propiedades deseadas de estos dominios monoméricos discretos. Sin embargo, los dominios monoméricos discretos, si bien están estructuralmente conservados, no están conservados a nivel de nucleótidos o aminoácidos, excepto para ciertos aminoácidos, p. ej., los residuos de cisteína del dominio A. De este modo, los métodos de recombinación de nucleótidos existentes se quedan cortos para generar y optimizar las propiedades deseadas de estos dominios monoméricos discretos.
La presente invención está dirigida a estos y otros problemas.
Breve resumen de la invención
La presente invención proporciona un método para identificar un multímero que se une a una molécula diana, comprendiendo el método,
En algunas realizaciones, al menos tres dominios monoméricos se unen a la misma molécula diana.
Adicionalmente se describen en la presente memoria métodos que comprenden una etapa adicional de mutación de al menos un dominio monomérico, proporcionando de ese modo una genoteca que comprende dominios monoméricos mutados. La etapa de mutación puede comprender recombinar una pluralidad de fragmentos polinucleotídicos de al menos un polinucleótido que codifica un dominio monomérico. La etapa de mutación puede comprender una evolución dirigida. La etapa de mutación puede comprender una mutagénesis dirigida al sitio.
Adicionalmente se describen en la presente memoria métodos que comprenden: escrutar la biblioteca de dominios monoméricos en busca de afinidad hacia una segunda molécula diana; identificar un dominio monomérico que se une a una segunda molécula diana; conectar al menos un dominio monomérico con afinidad hacia la primera molécula diana con al menos un dominio monomérico con afinidad hacia una segunda molécula diana, formando de ese modo una biblioteca de multímeros; escrutar la biblioteca de multímeros en busca de la capacidad de unirse a la primera y segunda moléculas diana; e identificar un multímero que se une a la primera y segunda moléculas diana, identificando de ese modo un multímero que se une específicamente a una primera y una segunda moléculas diana.
Ciertos métodos descritos en la presente memoria comprenden adicionalmente: proporcionar una segunda biblioteca de dominios monoméricos; escrutar la segunda biblioteca de dominios monoméricos en busca de afinidad hacia al menos una segunda molécula diana; identificar un segundo dominio monomérico que se une a la segunda molécula diana; conectar los dominios monoméricos identificados que se unen a la primera molécula diana o la segunda molécula diana, formando de ese modo una biblioteca de multímeros; escrutar la biblioteca de multímeros en busca de la capacidad de unirse a la primera y segunda moléculas diana; e identificar un multímero que se une a la primera y segunda moléculas diana.
La molécula diana se puede seleccionar del grupo que consiste en un...
Reivindicaciones:
1. Un método para identificar un multímero que se una a una molécula diana, comprendiendo los métodos,
2. El método de la reivindicación 1, donde el multímero seleccionado comprende al menos tres dominios monoméricos.
3. El método de cualquiera de las reivindicaciones precedentes, donde la biblioteca de multímeros se expresa como una presentación en fagos, una presentación en ribosomas, o una presentación en la superficie celular.
4. El método de cualquiera de las reivindicaciones precedentes, donde los dominios monoméricos están conectados por un conector polipeptídico heterólogo.
5. Un método para identificar un multímero que se une a al menos una molécula diana, comprendiendo el método:
6. El método de la reivindicación 5, que comprende adicionalmente identificar los multímeros que se unen a la molécula diana que tienen una avidez por la molécula diana que es mayor que la avidez de un dominio monomérico sencillo por la molécula diana.
7. El método de la reivindicación 5 o la reivindicación 6, donde uno o más de los multímeros comprende un dominio monomérico que se une específicamente a una segunda molécula diana.
8. Una biblioteca de multímeros, donde
9. La biblioteca de la reivindicación 8, donde cada dominio monomérico de los multímeros es un dominio monomérico de origen no natural.
10. La biblioteca de la reivindicación 8, donde los dominios monoméricos están conectados por un conector polipeptídico.
11. El método o la biblioteca de cualquiera de las reivindicaciones anteriores, donde la biblioteca contiene al menos 100 miembros.
12. Un polipéptido que comprende al menos dos dominios monoméricos separados por un conector heterólogo, donde cada dominio monomérico tiene 30-100 aminoácidos y comprende al menos dos enlaces disulfuro, donde cada dominio monomérico se une específicamente a una molécula diana y donde los dominios monoméricos son dominios de clase A del receptor de LDL.
13. El polipéptido de la reivindicación 12, donde cada dominio monomérico es un dominio monomérico de proteína de origen no natural.
14. El polipéptido de la reivindicación 12, donde el polipéptido comprende un primer dominio monomérico que se une a una primera molécula diana y un segundo dominio monomérico que se une a una segunda molécula diana.
15. El polipéptido de la reivindicación 12, donde el polipéptido comprende dos dominios monoméricos, teniendo cada uno especificidad de unión por un sitio diferente en una primera molécula diana.
16. El polipéptido de la reivindicación 12, donde el polipéptido comprende al menos tres dominios monoméricos.
17. El polipéptido de la reivindicación 12, donde los dominios monoméricos están conectados por un segundo conector polipeptídico.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]