PROCEDIMIENTO PARA DETECTAR E IDENTIFICAR BONAMIA OSTREAE Y BONAMIA EXITIOSA EN UN ÚNICO ENSAYO DE REACCIÓN EN CADENA DE LA POLIMERASA.
La presente invención es un procedimiento para detectar e identificar Bonamia ostreae y Bonamia exitiosa en un único ensayo de reacción en cadena de la polimerasa.
El procedimiento comprende aislar el ADN presente en una muestra de tejido de ostra, realizar un ensayo de reacción en cadena de la polimerasa sobre el ADN extraído, utilizando una mezcla que comprende cebadores específicos para B. ostreae y B. exitiosa en la región ITS1 y un cebador común para ambas especies en el gen 18S ARNr, y detectar las secuencias nucleotídicas amplificadas. Asimismo, la invención también proporciona un kit para detectar e identificar Bonamia ostreae y Bonamia exitiosa, que comprende los cebadores mencionados anteriormente, fragmentos de ADN clonados de la región a amplificar para B. ostreae y B. exitiosa, desoxinucleótidos trifosfato, tampón de reacción y manual de instrucciones.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201131549.
Solicitante: CENTRO TECNOLOGICO DEL MAR - FUNDACION CETMAR.
Nacionalidad solicitante: España.
Inventor/es: ABOLLO RODRIGUEZ,Elvira, VILLALBA GARCIA,Antonio, RAMILO ALVAREZ,Andrea.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
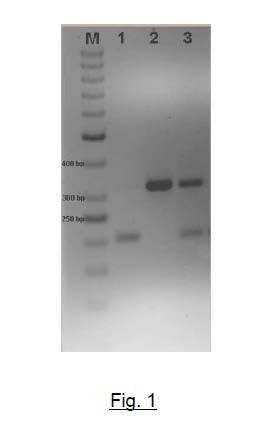
Fragmento de la descripción:
PROCEDIMIENTO PARA DETECTAR E IDENTIFICAR BONAMIA OSTREAE Y BONAMIA EXITIOSA EN UN ÚNICO ENSAYO DE REACCIÓN EN CADENA DE LA POLIMERASA
CAMPO DE LA INVENCIÓN
La presente invención se refiere a sondas de ADN procedentes de la región 18S-ITS1 del ADNr, para su utilización en la detección específica de Bonamia ostreae y Bonamia exitiosa en tejidos de ostra, mediante un procedimiento de reacción en cadena de la polimerasa multiplex. El procedimiento es de aplicación en el sector de la acuicultura para el control del estado de salud de los moluscos bivalvos, tanto en poblaciones naturales como cultivadas. Ambas especies patógenas son de declaración obligatoria, estando prohibidos los movimientos comerciales de animales enfermos a zonas legalmente reconocidas como libres de la enfermedad.
ANTECEDENTES DE LA INVENCIÓN
El género Bonamia agrupa actualmente cuatro especies de protozoos que infectan a varias especies de ostra en diferentes zonas del mundo. Dos de estas especies, B. ostreae y B. exitiosa, son altamente patógenas, siendo responsables de mortandades masivas de ostras tanto en el hemisferio Norte como Sur. Bonamia ostreae es una especie parásita que infecta a la ostra plana Ostrea edulis en Europa, los EE. UU., Canadá y Marruecos mientras que Bonamia exitiosa parasita a las especies de ostra Ostrea chilensis (=Tiostrea chilensis =Tiostrea lutaria) en Nueva Zelanda, Ostrea angasi en Australia y recientemente se ha descrito infectando a Ostrea edulis en Europa, aunque limitada en su extensión a Galicia (España) , algunos puntos del litoral atlántico y mediterráneo francés y a la costa italiana del mar Adriático.
La Organización Mundial de Salud Animal ha incluido ambas especies, B. ostreae y
B. exitiosa, en la lista de enfermedades que deben ser notificadas. La normativa europea recoge también que estas especies deben ser objeto de vigilancia, aunque B. exitiosa tiene consideración de enfermedad exótica pues en el momento de la redacción de la normativa no se había detectado esta especie en Europa.
Los métodos para la detección e identificación de Bonamia ostreae y B. exitiosa a partir de muestras de ostra se basan en métodos microscópicos (frotis, histopatología y microscopía electrónica) , métodos inmunológicos de detección antígeno-anticuerpo (Ag-Ac) y técnicas moleculares de reacción en cadena de la polimerasa (PCR) e hibridación in situ (ISH) . Sin embargo todos estos métodos adolecen de una serie de problemas: (1) los métodos citohistológicos y el método de detección Ag-Ac ofrece baja especificidad y baja sensibilidad en infecciones leves o latentes; (2) aunque el empleo de la microscopía electrónica ofrece buena especificidad su empleo en tareas rutinarias de detección y diagnóstico no es viable debido a la lentitud del proceso, la alta especialización profesional para efectuar un correcto diagnóstico y el propio coste de la técnica; y (3) las técnicas moleculares de PCR e ISH ofrecen una mayor sensibilidad que los métodos histocitológicos, pero carecen en su mayoría de especificidad.
Se han descrito diferentes procedimientos para la detección por PCR convencional de Bonamia ostreae. Estos procedimientos utilizan sondas que hibridan sobre el gen 18S del ARN ribosomal, el cual es un gen altamente conservado entre las especies de Bonamia por lo que las sondas no son específicas de especie. Un ejemplo es el procedimiento que utiliza los cebadores Bo-Boas en el que se detectan todos los haplosporidios englobados en la categoría de microcélulas, así como especies del género Haplosporidium (Cochennec, N. et al., 2000. Detection of Bonamia ostreae based on small subunit ribosomal probe. J. Invertebr. Pathol. 76, 26-32) . Otros dos ejemplos son el procedimiento que utiliza los cebadores CF-CR (Carnegie, R.B. et al., 2000. Development of a PCR assay for detection of the oyster pathogen Bonamia ostreae and support for its inclusion in the Haplosporidia. Dis. Aquat. Org. 42, 199-206) y el procedimiento que utiliza los cebadores BOG-F y BOG-R (Abollo,
E. et al., 2008. First detection of the protozoan parasite Bonamia exitiosa (Haplosporidia) infecting flat oyster Ostrea edulis grown in European waters. Aquaculture 274, 201-207) amplifican B. ostreae y B. exitiosa y otras especies. Ninguno de estos procedimientos de PCR es específico de especie, por lo que la identificación específica se debe realizar en un ensayo posterior mediante secuenciación o análisis de longitud de los polimorfismos de restricción (PCR-RFLPs) .
Empleando la misma región diana del ADN, el gen 18S del ARNr, también se ha desarrollado un ensayo de PCR cuantitativa descrito en Marty et al. (Marty, G. et al., 2006. Histopathology and a real-time PCR assay for detection of Bonamia ostreae in
Ostrea edulis cultured in western Canada. Aquaculture 261, 33-42) para la detección de las especies de Bonamia. Recientemente, también se ha desarrollado otro ensayo de PCR cuantitativa para Bonamia ostreae empleando como región diana en el diseño de los cebadores específicos el gen de la actina (Robert, M. et al., 2009. Molecular detection and quantification of the protozoan B. ostreae in the flat oyster Ostrea edulis. Molecular and Cellular Probes 2 (6) , 264-271) . Este último ensayo a diferencia de la invención propuesta no permite la detección simultánea y específica de ambas especies patógenas, Bonamia ostreae y B. exitiosa, en un único ensayo.
Para un diagnóstico seguro de los organismos patógenos utilizando la tecnología de las sondas de ADN, estas deben ser muy específicas (es decir no deben presentar reacciones de cruzamiento con los ácidos nucleicos de otros organismos) y muy sensibles (es decir deben detectar los niveles iniciales de infección por los patógenos) . A pesar de disponer de secuencias de dominio público en los bancos de genes no se conoce a priori a partir de qué genes del genoma pueden derivarse estas secuencias de sonda específicas. En la bibliografía no se presenta ninguna evidencia de que se puedan desarrollar sondas muy específicas en la región ITS1, pero sí se recogen los numerosos intentos fallidos de desarrollar sondas específicas a partir del gen 18S del ARNr (descritos en los dos párrafos anteriores) . La selección de sondas que resulten específicas para B. ostreae y B. exitiosa no resulta evidente para el experto en la materia a partir de la información disponible en el estado de la técnica. La presente invención proporciona sondas especie-específicas derivadas de la región espaciadora ITS1 y una sonda común derivada del gen 18S del ARNr para la identificación de B. ostreae y B. exitiosa.
El problema que plantea la técnica es la identificación simultánea y específica en un único ensayo de los patógenos Bonamia ostreae y Bonamia exitiosa, responsables de la bonamiosis en especies de ostra. La solución que propone la presente invención es un procedimiento de detección específica de Bonamia ostreae y Bonamia exitiosa en tejidos de ostra que comprende la realización de una PCR multiplex que utiliza cebadores específicos para B. ostreae y B. exitiosa en la región ITS1 y un cebador común para ambas especies en el gen 18S ARNr, identificados por las secuencias SEQ ID NO: 1, 2 y 3.
DESCRIPCIÓN DE LA INVENCIÓN
La presente invención es un procedimiento para detectar e identificar Bonamia ostreae y Bonamia exitiosa en un único ensayo de reacción en cadena de la polimerasa, que comprende: a) aislar el ADN presente en una muestra de tejido de ostra, b) realizar dicho ensayo de reacción en cadena de la polimerasa sobre el ADN extraído en a) utilizando una mezcla de cebadores que comprende cebadores específicos para B. ostreae y B. exitiosa en la región ITS1 y un cebador común para ambas especies en el gen 18S ARNr, identificados por las secuencias SEQ ID NO: 1, 2 y 3 y c) detectar las secuencias nucleotídicas amplificadas como resultado de dicha reacción en cadena de la polimerasa.
Una realización de la invención es el procedimiento de la invención, donde se utilizan cebadores que tienen una identidad del 95% respecto a los cebadores identificados por las secuencias SEQ ID NO: 1, 2 y 3. Una realización preferente es un procedimiento de la invención donde dicha identidad es del 85%.Y una realización más preferente es un procedimiento de la invención donde dicha identidad es del 75%.
En la presente solicitud, dicho porcentaje de identidad en una secuencia determinada se calcula teniendo en cuenta que un 95% de identidad significa que un 95% de residuos de la secuencia completa de los cebadores identificados por las secuencias SEQ ID NO: 1, 2 y 3 son idénticos a los residuos de la secuencia determinada.
La presente invención proporciona un nuevo método...
Reivindicaciones:
1. Un procedimiento para detectar e identificar Bonamia ostreae y Bonamia exitiosa en un único ensayo de reacción en cadena de la polimerasa, que comprende: a) aislar el ADN presente en una muestra de tejido de ostra, b) realizar dicho ensayo de reacción en cadena de la polimerasa sobre el ADN extraído en a) utilizando una mezcla de cebadores que comprende cebadores específicos para B. ostreae y B. exitiosa en la región ITS1 y un cebador común para ambas especies en el gen 18S ARNr, identificados por las secuencias SEQ ID NO: 1, 2 y 3 y c) detectar las secuencias nucleotídicas amplificadas como resultado de dicha reacción en cadena de la polimerasa.
2. Procedimiento según la reivindicación 1, donde se utilizan cebadores que tienen una identidad del 95% respecto a los cebadores identificados por las secuencias SEQ ID NO: 1, 2 y 3.
3. Procedimiento según la reivindicación 2, donde dicha identidad es del 85%.
4. Procedimiento según la reivindicación 2, donde dicha identidad es del 75%.
5. Un kit para detectar e identificar Bonamia ostreae y Bonamia exitiosa en un único ensayo de reacción en cadena de la polimerasa, que comprende: a) los cebadores específicos para B. ostreae y B. exitiosa en la región ITS1 y un cebador común para ambas especies en el gen 18S del ARNr, identificados por las secuencias SEQ ID NO: 1, 2 y 3, b) fragmentos de ADN clonados de la región a amplificar para B. ostreae y B. exitiosa, c) desoxinucleótidos trifosfato, d) tampón de reacción y e) manual de instrucciones que detalla el procedimiento a seguir.
6. Kit según la reivindicación 5, donde dichos desoxinucleótidos trifosfatos son dATP, dCTP, dGTP y dTTP a una concentración final de 200 μM.
7. Kit según una de las reivindicaciones 5 ó 6, donde dicho tampón de reacción es 50 mM de cloruro de potasio, 10 mM de Tris-HCl (pH 8.3 a temperatura ambiente) y 1, 5 mM de cloruro de magnesio.
Fig. 1
Fig. 2
Fig. 3
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]