Métodos de preparación de productos de expresión de proteínas de fusión modulares.
Vector que comprende cada uno de los siguientes elementos dispuestos secuencialmente para formar un marcode lectura abierto modular,
comprendiendo dicho vector:
a) un primer sitio de restricción que codifica para un primer grupo de aminoácidos, comprendiendo el primer grupode aminoácidos al menos dos aminoácidos,
b) un primer marco de lectura abierto que codifica para un polipéptido de interés, en el que la secuencia codificantedel polipéptido de interés está en marco con la secuencia codificante de al menos dos aminoácidos del primer sitiode restricción,
c) un segundo sitio de restricción que codifica para un segundo grupo de aminoácidos, comprendiendo el segundogrupo de aminoácidos al menos dos aminoácidos, en el que la secuencia codificante del segundo grupo deaminoácidos del segundo sitio de restricción está en marco con la secuencia codificante del polipéptido de interésdel primer marco de lectura abierto, y en el que la secuencia polinucleotídica del segundo sitio de restricción no es lamisma que la secuencia polinucleotídica del primer sitio de restricción,
d) una secuencia polinucleotídica espaciadora, codificando la secuencia polinucleotídica espaciadora para al menostres aminoácidos espaciadores, y en la que la secuencia codificante para la secuencia espaciadora está en marcocon la secuencia polinucleotídica del segundo sitio de restricción, y
e) un tercer sitio de restricción que codifica para un tercer grupo de aminoácidos, comprendiendo el tercer grupo deaminoácidos al menos dos aminoácidos, en el que la secuencia codificante del tercer grupo de aminoácidos deltercer sitio de restricción está en marco con la secuencia codificante de los al menos tres aminoácidos espaciadoresy en el que la secuencia polinucleotídica del tercer sitio de restricción no es la misma que la secuenciapolinucleotídica del primer sitio de restricción y el tercer sitio de restricción no es el mismo que la secuenciapolinucleotídica del segundo sitio de restricción;
en el que los elementos (a) a (e) están unidos en marco entre sí para formar el marco de lectura abierto modular,en el que el primer sitio de restricción es susceptible a una enzima de restricción que producirá una primeraproyección monocatenaria que comprende al menos 2 nucleótidos, en el que el segundo sitio de restricción essusceptible a una enzima de restricción que producirá una segunda proyección monocatenaria que comprende almenos 2 nucleótidos, y
en el que dichas proyecciones primera y segunda son complementarias entre sí,
en el que tras el apareamiento de las proyecciones primera y segunda, se destruyen el primer sitio de restricción y elsegundo sitio de restricción.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2006/060065.
Solicitante: INTREXON CORPORATION.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 1750 Kraft Drive, Suite 1400 Blacksburg, VA 24060 ESTADOS UNIDOS DE AMERICA.
Inventor/es: REED,THOMAS.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N1/21 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 1/00 Microorganismos, p.ej. protozoos; Composiciones que los contienen (preparaciones de uso médico que contienen material de protozoos, bacterias o virus A61K 35/66, de algas A61K 36/02, de hongos A61K 36/06; preparación de composiciones de uso médico que contienen antígenos o anticuerpos bacterianos, p. ej. vacunas bacterianas, A61K 39/00 ); Procesos de cultivo o conservación de microorganismos, o de composiciones que los contienen; Procesos de preparación o aislamiento de una composición que contiene un microorganismo; Sus medios de cultivo. › modificados por la introducción de material genético extraño.
- C12N15/74 C12N […] › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Vectores o sistemas de expresión especialmente adaptados a huéspedes procariotas distintos a E. coli, p. ej. Lactobacillus, Micromonospora.
PDF original: ES-2436277_T3.pdf
Fragmento de la descripción:
Métodos de preparación de productos de expresión de proteínas de fusión modulares
Campo de la invención La invención se refiere a métodos de biología molecular y a productos para realizar los métodos. Más específicamente, la invención se refiere a métodos de clonación de ácidos nucleicos que codifican para proteínas modulares, en los que pueden añadirse secuencialmente módulos en ubicaciones predeterminadas.
Esta solicitud tiene contenido relacionado con las solicitudes n.os US 2004/0185556, PCT/US2004/013517, WO 2005/040336 y US 20040572011 P (documento WO2005/116231) .
Antecedentes y técnica anterior
Se conocen en la técnica tecnologías de ADN recombinante y métodos de biología molecular de clonación de ácidos nucleicos. Tales métodos incluyen la manipulación de ácidos nucleicos usando endonucleasas de restricción, ligasas, nucleótido/nucleósido cinasas y fosfatasas, polimerasas y otras herramientas de biología molecular para generar ácidos nucleicos recombinantes deseados. Se han escrito varios manuales de laboratorio como libros de referencia para investigadores de biología molecular. Los ejemplos de estos manuales de referencia incluyen Sambrook, J., E. F. Fritsch y T. Maniatis, 1989. Molecular Cloning: A Laborator y Manual, 2ª. ed. Cold Spring Harbor, Nueva York: Cold Spring Harbor Laborator y Press; y Joseph Sambrook y David Russell, 2001. Molecular Cloning: A Laborator y Manual, 3ª ed. Cold Spring Harbor, Nueva York: Cold Spring Harbor Laborator y Press.
Un problema en la técnica es la falta de metodología para diseñar por ingeniería genética productos de expresión de proteínas quiméricas en los que puedan insertarse fácilmente elementos modulares en posiciones deseadas dentro de una quimera. En general, aunque se han preparado satisfactoriamente proteínas híbridas o quiméricas, el objetivo de la síntesis ha sido hacia productos terminales finales, sin la incorporación de un mecanismo predeterminado para añadir más módulos. Un aspecto de la invención aborda este problema contemplando la necesidad futura de quimeras y variaciones de las mismas y haciendo posible construirlas sin comenzar desde cero para cada una. Una realización de la invención se refiere en general a métodos de preparación de productos de expresión de proteínas de fusión. Otra realización de la invención se refiere a métodos de preparación de productos de expresión de proteínas de fusión con módulos diseñados previamente por ingeniería genética. Otro aspecto de la invención se refiere a módulos o moléculas de bloques de construcción, diseñándose previamente los módulos para que puedan insertarse en un casete de expresión de proteínas quiméricas. La presente invención también contempla bibliotecas de módulos diseñados por ingeniería genética que pueden utilizarse en los métodos dados a conocer. En una realización, se preparan módulos con sitios de restricción predeterminados.
Descripción detallada de secuencias polipeptídicas y polinucleotídicas La SEQ ID NO: 1 es un ejemplo de un módulo de ácido nucleico. El módulo tiene la estructura 5’ - 3’ de: 5’-sitio de restricción predeterminado-marco de lectura abierto (ORF) de polipéptido de interés-sitio de restricción predeterminado-fragmento de relleno-sitio de restricción predeterminado-3’. Los sitios de restricción predeterminados y el fragmento de relleno también codifican para aminoácidos en mamíferos y están en marco con 45 el polipéptido de interés, por tanto todo el módulo es un marco de lectura abierto compuesto. La SEQ ID NO: 1 se representa en la figura 11.
La SEQ ID NO: 2 es el polipéptido codificado por la SEQ ID NO: 1.
Breve descripción de los dibujos La figura 1 muestra un método para construir proteínas de fusión en una dirección de extremo N-terminal a extremo C-terminal, utilizando dos reactivos de partida de ADN circular.
La figura 2 muestra un método para construir proteínas de fusión en una dirección de extremo C-terminal a extremo N-terminal, utilizando dos reactivos de partida de ADN circular.
La figura 3 muestra un método para construir proteínas de fusión en una dirección de extremo N-terminal a extremo C-terminal, utilizando un reactivo de partida de ADN circular y un reactivo de partida de ADN lineal.
La figura 4 muestra un método para construir proteínas de fusión en una dirección de extremo N-terminal a extremo C-terminal, utilizando dos reactivos de partida de ADN lineal.
La figura 5 muestra un método recursivo para construir proteínas de fusión en una dirección de extremo N-terminal a 65 extremo C-terminal.
Las figuras 6A-6C muestran ejemplos de polipéptidos quiméricos en los que se derivan módulos de partes de exones.
Las figuras 7A-7C muestran ejemplos de polipéptidos quiméricos en los que un módulo contiene una señal de 5 localización.
Las figuras 8A-8C muestran ejemplos de proteínas de fusión en los que un módulo contiene una etiqueta de epítopo.
Las figuras 9A-9C muestran ejemplos de proteínas de fusión en los que los módulos contienen diferentes dominios 10 funcionales.
La figura 10 muestra un casete de expresión de ejemplo que contiene una secuencia que codifica para proteína quimérica preparado según los métodos de la invención.
La figura 11 muestra un módulo de ejemplo que contiene un marco de lectura abierto de interés (ORF) flanqueado por sitios de restricción predeterminados y un fragmento de relleno.
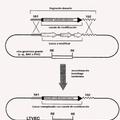
La figura 12 muestra un ejemplo de una biblioteca de módulos útiles en la invención. Los miembros de la biblioteca tienen sitios de restricción predeterminados idénticos indicados como Sitio I, Sitio II y Sitio III. Los marcos de lectura 20 abiertos no están dibujados a escala, y por tanto pueden variar en longitud. En una realización, los miembros de la biblioteca están contenidos dentro del ADN de vector. En una realización, el vector es un plásmido circular.
La figura 13 muestra un método de preparación de proteínas de fusión mediante síntesis combinatoria dinámica. Las abreviaturas en esta figura son tal como sigue: N4 representa NgoM IV; X1 representa Xma I; C1 representa Cla I; 25 dir significa directo; inv significa inverso; V representa sitios NgoM IV y Xma I vestigiales.
Las figuras 14A-14D muestran ejemplos de vectores que contienen constructos génicos de polipéptidos quiméricos, modulares.
Breve descripción de la invención La invención se refiere a métodos de preparación de productos de expresión de proteínas quiméricas modulares y a composiciones utilizadas en los métodos. En particular, la invención se refiere a la clonación secuencial, direccional de polinucleótidos que codifican para módulos polipeptídicos. Cada elemento clonable contiene un marco de lectura abierto flanqueado por sitios de restricción predeterminados. Los métodos incluyen el uso de elementos clonables y vectores que contienen estos elementos como materiales de partida para técnicas de ADN recombinante. Una ventaja de la invención es que permite que se preparen muchas variaciones de proteínas de fusión rápida y fácilmente sin necesidad de diseñar y evaluar cada etapa de clonación posterior.
La invención es un vector que comprende cada uno de los siguientes elementos dispuestos secuencialmente para formar un marco de lectura abierto modular, comprendiendo dicho vector:
a) un primer sitio de restricción que codifica para un primer grupo de aminoácidos, comprendiendo el primer grupo de aminoácidos al menos dos aminoácidos,
b) un primer marco de lectura abierto que codifica para un polipéptido de interés, en el que la secuencia codificante del polipéptido de interés está en marco con la secuencia codificante de al menos dos aminoácidos del primer sitio de restricción,
c) un segundo sitio de restricción que codifica para un segundo grupo de aminoácidos, comprendiendo el segundo grupo de aminoácidos al menos dos aminoácidos, en el que la secuencia codificante del segundo grupo de aminoácidos del segundo sitio de restricción está en marco con la secuencia codificante del polipéptido de interés del primer marco de lectura abierto, y en el que la secuencia polinucleotídica del segundo sitio de restricción no es la misma que la secuencia polinucleotídica del primer sitio de restricción,
d) una secuencia polinucleotídica espaciadora, codificando la secuencia polinucleotídica espaciadora para al menos tres aminoácidos espaciadores, y en el que la secuencia codificante para la secuencia espaciadora está en marco con la secuencia polinucleotídica del segundo sitio de restricción, y
e) un tercer... [Seguir leyendo]
Reivindicaciones:
1. Vector que comprende cada uno de los siguientes elementos dispuestos secuencialmente para formar un marco de lectura abierto modular, comprendiendo dicho vector:
a) un primer sitio de restricción que codifica para un primer grupo de aminoácidos, comprendiendo el primer grupo de aminoácidos al menos dos aminoácidos,
b) un primer marco de lectura abierto que codifica para un polipéptido de interés, en el que la secuencia codificante del polipéptido de interés está en marco con la secuencia codificante de al menos dos aminoácidos del primer sitio de restricción,
c) un segundo sitio de restricción que codifica para un segundo grupo de aminoácidos, comprendiendo el segundo grupo de aminoácidos al menos dos aminoácidos, en el que la secuencia codificante del segundo grupo de aminoácidos del segundo sitio de restricción está en marco con la secuencia codificante del polipéptido de interés del primer marco de lectura abierto, y en el que la secuencia polinucleotídica del segundo sitio de restricción no es la misma que la secuencia polinucleotídica del primer sitio de restricción,
d) una secuencia polinucleotídica espaciadora, codificando la secuencia polinucleotídica espaciadora para al menos tres aminoácidos espaciadores, y en la que la secuencia codificante para la secuencia espaciadora está en marco con la secuencia polinucleotídica del segundo sitio de restricción, y
e) un tercer sitio de restricción que codifica para un tercer grupo de aminoácidos, comprendiendo el tercer grupo de aminoácidos al menos dos aminoácidos, en el que la secuencia codificante del tercer grupo de aminoácidos del
tercer sitio de restricción está en marco con la secuencia codificante de los al menos tres aminoácidos espaciadores y en el que la secuencia polinucleotídica del tercer sitio de restricción no es la misma que la secuencia polinucleotídica del primer sitio de restricción y el tercer sitio de restricción no es el mismo que la secuencia polinucleotídica del segundo sitio de restricción;
en el que los elementos (a) a (e) están unidos en marco entre sí para formar el marco de lectura abierto modular,
en el que el primer sitio de restricción es susceptible a una enzima de restricción que producirá una primera proyección monocatenaria que comprende al menos 2 nucleótidos, en el que el segundo sitio de restricción es susceptible a una enzima de restricción que producirá una segunda proyección monocatenaria que comprende al
menos 2 nucleótidos, y
en el que dichas proyecciones primera y segunda son complementarias entre sí,
en el que tras el apareamiento de las proyecciones primera y segunda, se destruyen el primer sitio de restricción y el segundo sitio de restricción.
2. Vector según la reivindicación 1, en el que el polipéptido de interés es una proteína o un polipéptido de longitud completa, un dominio funcional de proteína, un dominio estructural de proteína, un dominio enzimático de proteína, un dominio de inhibición de proteína, un dominio de unión de proteína, un dominio de localización de proteína o un 45 epítopo.
3. Vector según la reivindicación 1 o la reivindicación 2, en el que dicho primer sitio de restricción comprende una secuencia polinucleotídica reconocida por la enzima de restricción NgoM IV.
4. Vector según una cualquiera de las reivindicaciones 1-3, en el que el segundo sitio de restricción comprende una secuencia polinucleotídica reconocida por la enzima de restricción Xma I.
5. Vector según una cualquiera de las reivindicaciones 1-4, en el que el tercer sitio de restricción comprende una
secuencia polinucleotídica reconocida por la enzima de restricción Cla I. 55
6. Vector según una cualquiera de las reivindicaciones 1-5, en el que la secuencia polinucleotídica espaciadora codifica para tres aminoácidos de glicina.
7. Vector según una cualquiera de las reivindicaciones 1-6, en el que se inserta al menos un marco de lectura abierto adicional entre el primer marco de lectura abierto y el segundo sitio de restricción, en el que cada uno del al menos un marco de lectura abierto adicional está en marco con tanto el primer marco de lectura abierto como el segundo grupo de restricción, y en el que cada uno de los marcos de lectura abiertos adicionales codifica para al menos un polipéptido de interés adicional.
8. Vector según la reivindicación 7, en el que no hay sitios de restricción en ninguno de los marcos de lectura abiertos entre los grupos de restricción primero y segundo que aparecen en los sitios de restricción o bien primero o bien segundo.
9. Método de preparación de un polipéptido de fusión, que comprende:
a) digerir el vector según una cualquiera de las reivindicaciones 3 a 8 con enzimas de restricción que reconocen los sitios de restricción segundo y tercero para crear un vector lineal con una proyección monocatenaria primera y segunda, en el que las proyecciones primera y segunda son incompatibles entre sí,
b) proporcionar un primer inserto, comprendiendo el primer inserto al menos un marco de lectura abierto adicional, en el que el al menos un marco de lectura abierto adicional comprende proyecciones en 5’ y 3’ que son compatibles con las proyecciones primera y segunda, en el que cada uno del al menos un marco de lectura abierto adicional codifica para al menos un polipéptido de interés adicional,
c) ligar el primer inserto en el vector lineal para formar un vector de expresión ligado, en el que el ligamiento de la proyección en 5’ del al menos un marco de lectura abierto se aparea con la primera proyección monocatenaria del vector lineal, en el que el ligamiento de la proyección en 3’ del al menos un marco de lectura abierto se aparea con la segunda proyección monocatenaria del vector lineal, y en el que el ligamiento de la proyección en 3’ recrea el tercer sitio de restricción;
en el que el primer marco de lectura abierto y el al menos un marco de lectura abierto adicional se ligan en marco entre sí.
10. Método de preparación de constructos de ADN de proteínas de fusión, que comprende:
a) proporcionar reactivos polinucleotídicos primero y segundo, conteniendo cada reactivo un marco de lectura abierto de interés, y estando flanqueado cada marco de lectura abierto de interés por al menos 3 sitios de endonucleasas de restricción predeterminados, en el que 2 de los sitios predeterminados tienen proyecciones compatibles, en el que el primer sitio de restricción es susceptible a una enzima de restricción que producirá una primera proyección monocatenaria que comprende al menos 2 nucleótidos, en el que el segundo sitio de restricción es susceptible a una enzima de restricción que producirá una segunda proyección monocatenaria que comprende al menos 2 nucleótidos, y en el que dichas proyecciones primera y segunda son complementarias entre sí, en el que tras el apareamiento de las proyecciones primera y segunda, se destruyen el primer sitio de restricción y el segundo sitio de restricción,
b) digerir el primer reactivo polinucleotídico con 2 endonucleasas de restricción diferentes que escinden en 2 de los sitios de endonucleasas de restricción predeterminados, generando un polinucleótido con 2 proyecciones diferentes,
c) digerir el segundo reactivo polinucleotídico con 2 endonucleasas de restricción diferentes que escinden en 2 de los sitios de endonucleasas de restricción predeterminados, en el que se libera un polinucleótido que contiene un marco de lectura abierto de interés, teniendo el polinucleótido liberado 2 proyecciones diferentes, y en el que una de las proyecciones es compatible con una proyección del polinucleótido digerido en la etapa b) ,
d) mezclar entre sí, aparear y ligar el polinucleótido generado en la etapa b) y el polinucleótido liberado que contiene un marco de lectura abierto de interés de la etapa c) , en el que se genera un tercer polinucleótido que contiene una 45 fusión de marcos de lectura abiertos, en el que una unión entre los marcos de lectura abiertos de interés ya no es susceptible a la digestión con cualquiera de las endonucleasas que cortan en los 3 sitios de restricción predeterminados, y en el que las secuencias que flanquean la fusión de marcos de lectura abiertos de interés contienen los 3 sitios de restricción predeterminados.
11. Método de preparación de constructos de ADN de proteínas de fusión, que comprende:
a) proporcionar reactivos polinucleotídicos primero y segundo, conteniendo cada reactivo un marco de lectura abierto de interés, y estando flanqueado cada marco de lectura abierto de interés por al menos 3 sitios de endonucleasas de restricción predeterminados, en el que 2 de los sitios predeterminados tienen proyecciones 55 compatibles, en el que el primer sitio de restricción es susceptible a una enzima de restricción que producirá una primera proyección monocatenaria que comprende al menos 2 nucleótidos, en el que el segundo sitio de restricción es susceptible a una enzima de restricción que producirá una segunda proyección monocatenaria que comprende al menos 2 nucleótidos, y en el que dichas proyecciones primera y segunda son complementarias entre sí, en el que tras el apareamiento de las proyecciones primera y segunda, se destruyen el primer sitio de restricción y el segundo sitio de restricción,
b) digerir el primer reactivo polinucleotídico con 2 endonucleasas de restricción diferentes que escinden en 2 de los sitios de endonucleasas de restricción predeterminados, generando un polinucleótido con 2 proyecciones incompatibles,
c) digerir el segundo reactivo polinucleotídico con 2 endonucleasas de restricción diferentes que escinden en 2 de los sitios de endonucleasas de restricción predeterminados, en el que 1 de las endonucleasas de restricción es idéntica a una endonucleasa de restricción de la etapa b) , y en el que se libera un polinucleótido que contiene un marco de lectura abierto de interés, y en el que el polinucleótido liberado tiene 2 proyecciones incompatibles,
d) mezclar entre sí, aparear y ligar el polinucleótido generado en la etapa b) y el polinucleótido liberado que contiene un marco de lectura abierto de interés de la etapa c) , en el que se genera un tercer polinucleótido que contiene una fusión de marcos de lectura abiertos, en el que la fusión de marcos de lectura abiertos de interés está flanqueada por dichos sitios de restricción predeterminados, y en el que una unión entre los marcos de lectura abiertos de interés carece de dichos 3 sitios de endonucleasas de restricción predeterminados.
12. Método según la reivindicación 10 u 11, en el que los sitios de restricción predeterminados son secuencias que pueden escindirse por NgoM IV, Xma I y Cla I.
Patentes similares o relacionadas:
Sistema de expresión bacteriano cistrónico doble, del 25 de Marzo de 2020, de LUPIN LIMITED: Un procedimiento para la producción de un anticuerpo o un fragmento del mismo que comprende las etapas de: (i) transformar una célula hospedadora bacteriana […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 15 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de células bacterianas para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no replicativa (NRTP) para su introducción […]
Método de construcción de biblioteca de péptidos y vectores relacionados, del 1 de Enero de 2020, de Hunan Zonsen Peplib Biotech Co., Ltd: Método integrado de construcción de una biblioteca de péptidos completa que contiene todos los péptidos posibles para el tamaño específico, comprendiendo el método: (i) […]
 Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Transporte de proteínas basado en bacterias, del 14 de Agosto de 2019, de Universität Basel: Un vector que comprende, en la dirección 5' a 3': un promotor; una primera secuencia de ADN que codifica una señal de transporte procedente […]
Sistema de expresión y secreción, del 26 de Junio de 2019, de F. HOFFMANN-LA ROCHE AG: Una molécula de ácido nucleico que codifica un primer polipéptido y un segundo polipéptido, enlazada de forma funcional a una secuencia señal que codifica […]
 Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Síntesis microbiana de aldehídos y alcoholes correspondientes, del 17 de Junio de 2019, de Easel Biotechnologies, LLC: Un método para producir un alcohol, que comprende: cultivar una pluralidad de células microbianas en un medio de fermentación, en donde las […]