Extensión de cebadores correctora.
Método de realización de una reacción de extensión de cebador,
comprendiendo el método:
a. poner en contacto un oligonucleótico con: (i) un ácido nucleico molde, e (ii) una ácido nucleico polimerasa quepresenta actividad exonucleasa 3' a 5', en la que:
i. el oligonucleótido comprende un nucleótido modificado que no resulta eliminado por la actividad exonucleasa 3' a5',
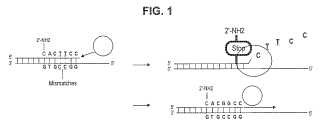
ii. el nucleótido modificado no es el nucleótido 3'-terminal ó 5'-terminal ni el penúltimo nucleótido del extremo 3' deloligonucleótido,
iii. el oligonucleótido presenta una parte 3' y una parte 5', en el que la parte 3' comprende los nucleótidos deloligonucleótido que se encuentran en el lado 3' del nucleótido modificado y la parte 5' comprende los nucleótidos deloligonucleótido que se encuentran en el lado 5' del nucleótido modificado, y
iv. la etapa de puesta en contacto se lleva a cabo bajo condiciones adecuadas para permitir que la polimerasa editela parte 3' del oligonucleótido de una manera específica de molde en el caso de que la parte 3' del oligonucleótido nosea 100% complementaria al ácido nucleico molde, y
b. llevar a cabo una reacción de extensión de cebador mediante la extensión del oligonucleótido de un mododependiente de molde, en la que el nucleótido modificado comprende una fracción 2' seleccionada de entre el grupoque consiste de amino, O-metilo, O-fosfato y fosforotioato.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2009/005770.
Solicitante: F. HOFFMANN-LA ROCHE AG.
Inventor/es: MYERS, THOMAS WILLIAM, FISS,Ellen.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2384681_T3.pdf
Fragmento de la descripción:
Extensión de cebadores correctora Antecedentes de la invención El desarrollo de la tecnología de amplificación de ácidos nucleicos ha revolucionado las ciencias del análisis e ingeniería genéticos. Por ejemplo, la reacción en cadena de la polimerasa (PCR) se utiliza rutinariamente para amplificar ácidos nucleicos diana utilizando ácidos nucleicos cebadores seleccionados, por ejemplo para facilitar la detección del ácido nucleico diana como parte de un análisis diagnóstico, forense u otra aplicación. Los cebadores típicamente funcionan en parejas diseñadas para la extensión de un componente de la pareja hacia el otro cubriendo la región diana seleccionada. Un ciclo de PCR típico incluye una etapa de desnaturalización a temperatura elevada (por ejemplo 85ºC o superior) durante la que las cadenas de ácidos nucleicos de doble cadena se separan uno de otro, una etapa de apareamiento a temperatura baja (por ejemplo entre 45ºC y 65ºC) durante la que los cebadores se hibridan con las cadenas individuales separadas, y una etapa de extensión a temperatura intermedia (por ejemplo aproximadamente 72ºC) durante la que una ácido nucleico polimerasa extiende los cebadores. También se utilizan procedimientos de termociclado a dos temperaturas. Estos procedimientos generalmente incluyen una etapa de desnaturalización a alta temperatura y una etapa de apareamiento-extensión a baja temperatura.
También se describen PCRs en muchas patentes US diferentes, entre ellas, por ejemplo, la patente US nº 4.683.195, titulada "Process for amplifying, detecting, and/or cloning nucleic acid sequences", concedida a Mullis et al. el 28 de julio de 1987, la patente US nº 4.683.202, titulada "Process for amplifying nucleic acid sequences", concedida a Mullis el 28 de julio de 1987, y la patente US nº 4.965.188, titulada "Process for amplifying, detecting, and/or cloning nucleic acid sequences using a thermostable enzyme", concedida a Mullis et al. el 23 de octubre de 1990. Además, también se describen técnicas relacionadas con la PCR en diversas otras publicaciones, tales como Innis et al. (editores) , PCR Protocols: A Guide to Methods and Applications, Elsevier Science & Technology Books, 1990, Innis et al. (editores) , PCR Applications: Protocols for Functional Genomics, Academic Press, 1999; Edwards et al., Real-Time PCR, Taylor & Francis, Inc., 2004, y Rapley et al., Molecular Analysis and Genome Discover y , John Wiley & Sons, Inc., 2004. La amplificación de ácidos nucleicos víricos utilizando cebadores degenerados que contienen inosina para la tolerancia a los desapareamientos se describe en Fujiwara et al., Genome Research 4:239-240, 1995; U/Tma es la única polimerasa Exo+ utilizada con éxito con cebadores degenerados.
Además, se describen métodos de tipo PCR que utilizan cebadores quiméricos en los que se sustituyen el sexto y séptimo desoxirribonucleótidos del extremo 3' por ribonucleótidos (patente EP nº 1.167.524) o cebadores que contienen modificaciones 2'-O-metilo en combinación con una ADN polimerasa que no presenta actividad exonucleasa 3' a 5' (patente WO nº 2005/012499) .
Breve descripción resumida de la invención La presente invención proporciona métodos para llevar a cabo una reacción de extensión de cebadores. En algunas realizaciones, los métodos comprenden:
a. poner en contacto un oligonucleótico con: (i) un ácido nucleico molde, e (ii) una ácido nucleico polimerasa que presenta actividad exonucleasa 3' a 5', en la que:
i. el oligonucleótido comprende un nucleótido modificado que no resulta eliminado por la actividad exonucleasa 3' a 5',
ii. el nucleótido modificado no es el nucleótido 3'-terminal ó 5'-terminal ni el penúltimo nucleótido del extremo 3' del oligonucleótido, iii. el oligonucleótido presenta una parte 3' y una parte 5', en el que la parte 3' comprende los nucleótidos del oligonucleótido que se encuentran en el lado 3' del nucleótido modificado y la parte 5' comprende los nucleótidos del oligonucleótido que se encuentran en el lado 5' del nucleótido modificado, y
iv. la etapa de puesta en contacto se lleva a cabo bajo condiciones adecuadas para permitir que la polimerasa edite la parte 3' del oligonucleótido de una manera específica de molde en el caso de que la parte 3' del oligonucleótido no sea 100% complementaria al ácido nucleico molde, y
b. llevar a cabo una reacción de extensión de cebador mediante la extensión del oligonucleótido de un modo dependiente de molde, en la que el nucleótido modificado comprende una fracción 2' seleccionada de entre el grupo que consiste de amino, O-metilo, O-fosfato y fosforotioato.
En algunas realizaciones, la parte 3' del oligonucleótido, antes de la edición, es entre 70% y 100% complementaria al ácido nucleico de molde.
En algunas realizaciones, el ácido nucleico de molde procede de una muestra biológica. En algunas realizaciones, el ácido nucleico de molde procede de un genoma vírico o bacteriano.
En algunas realizaciones, el ácido nucleico de molde es ARN. En algunas realizaciones, el ácido nucleico de molde es ADN.
En algunas realizaciones, el nucleótido modificado se encuentra 2 a 10 nucleótidos del extremo 3' del oligonucleótido. En algunas realizaciones, el nucleótido modificado se encuentra 5 a 7 nucleótidos del extremo 3' del oligonucleótido.
En algunas realizaciones, el oligonucleótido presenta:
entre 15 y 40 nucleótidos de longitud, es complementario por lo menos al 70% respecto al ácido nucleico de molde a lo largo de la longitud completa del oligonucleótido, y/o el nucleótido modificado se encuentra 2 a 10 nucleótidos del extremo 3' del oligonucleótido.
En algunas realizaciones, la polimerasa comprende una modificación covalente térmicamente reversible, en la que la incubación a una temperatura superior a 50ºC en un tampón acuoso a pH alcalino revierte la modificación covalente y resulta en un incremento de por lo menos dos veces de la actividad enzimática en menos de 20 minutos.
En algunas realizaciones, la etapa b comprende una reacción en cadena de la polimerasa. En algunas realizaciones, la etapa b comprende llevar a cabo una reacción en cadena de la polimerasa en tiempo real.
En algunas realizaciones, el nucleótido modificado no comprende una fracción fluorescente.
La presente invención también proporciona mezclas de reacción que comprenden uno o más de lo reactivos indicados en la presente memoria. Las mezclas de reacción comprenden un oligonucleótido, comprendiendo el oligonucleótido un nucleótido modificado que no resulta eliminado por la actividad exonucleasa 3' a 5' de una ácido nucleico polimerasa, en la que el nucleótido modificado no es el nucleótido 3'-terminal ó 5'-terminal ni el penúltimo nucleótido del extremo 3' del oligonucleótido, y el nucleótido modificado comprende una fracción 2' seleccionada de entre el grupo que consiste de amino, O-metilo, O-fosfato y fosforotioato, pero no comprende una fracción fluorescente, y comprende una ácido nucleico polimerasa que presenta una actividad exonucleasa 3' a 5'.
En algunas realizaciones, la mezcla de reacción comprende además un ácido nucleico molde. En algunas realizaciones, la mezcla de reacción comprende además desoxinucleósidos trifosfato.
En algunas realizaciones, el oligonucleótido presenta una longitud de entre 15 y 40 nucleótidos.
En algunas realizaciones, el nucleótido modificado se encuentra 3 a 10 nucleótidos del extremo 3' del oligonucleótido.
La presente invención incluye además kits que comprenden uno o más reactivos indicados en la presente memoria. El kit comprende un oligonucleótido, comprendiendo el oligonucleótido un nucleótido modificado que no resulta eliminado por la actividad exonucleasa 3' a 5' de una ácido nucleico polimerasa, en el que el nucleótido modificado no es el nucleótido 3'-terminal ó 5'-terminal ni el penúltimo nucleótido del extremo 3' del oligonucleótido y el nucleótido modificado comprende una fracción 2' seleccionada de entre el grupo que consiste de amino, O-metilo, Ofosfato y fosforotioato, pero no comprende una fracción fluorescente y comprende una ácido nucleico polimerasa que presenta una actividad exonucleasa 3' a 5'.
En algunas realizaciones, el kit comprende además desoxinucleósidos trifosfato.
En algunas realizaciones, el oligonucleótido presenta una longitud de entre 15 y 40 nucleótidos.
En algunas realizaciones, el nucleótido modificado se encuentra... [Seguir leyendo]
Reivindicaciones:
1. Método de realización de una reacción de extensión de cebador, comprendiendo el método:
a. poner en contacto un oligonucleótico con: (i) un ácido nucleico molde, e (ii) una ácido nucleico polimerasa que presenta actividad exonucleasa 3' a 5', en la que:
i. el oligonucleótido comprende un nucleótido modificado que no resulta eliminado por la actividad exonucleasa 3' a 5',
ii. el nucleótido modificado no es el nucleótid.
3. terminal .
5. terminal ni el penúltimo nucleótido del extremo 3' del oligonucleótido, iii. el oligonucleótido presenta una parte 3' y una parte 5', en el que la parte 3' comprende los nucleótidos del oligonucleótido que se encuentran en el lado 3' del nucleótido modificado y la parte 5' comprende los nucleótidos del oligonucleótido que se encuentran en el lado 5' del nucleótido modificado, y
iv. la etapa de puesta en contacto se lleva a cabo bajo condiciones adecuadas para permitir que la polimerasa edite la parte 3' del oligonucleótido de una manera específica de molde en el caso de que la parte 3' del oligonucleótido no sea 100% complementaria al ácido nucleico molde, y
b. llevar a cabo una reacción de extensión de cebador mediante la extensión del oligonucleótido de un modo dependiente de molde, en la que el nucleótido modificado comprende una fracción 2' seleccionada de entre el grupo que consiste de amino, O-metilo, O-fosfato y fosforotioato.
2. Método según la reivindicación 1, en el que el nucleótido modificado se encuentra 2 a 10 nucleótidos del extremo 3' del oligonucleótido.
3. Método según la reivindicación 1, en el que el nucleótido modificado no comprende una fracción fluorescente.
4. Mezcla de reacción que comprende un oligonucleótido, comprendiendo el oligonucleótido un nucleótido modificado que no resulta eliminado por la actividad de exonucleasa 3' a 5' de una ácido nucleico polimerasa, en la que el nucleótido modificado no es el nucleótido 3' .
5. terminal ni el penúltimo nucleótido del extremo 3' del oligonucleótido, y el nucleótido modificado comprende una fracción 2' seleccionada de entre el grupo que consiste de amino, O-metilo, O-fosfato y fosforotioato, pero no comprende una fracción fluorescente, y que comprende una ácido nucleico polimerasa que presenta actividad de exonucleasa 3' a 5'.
5. Mezcla de reacción según la reivindicación 4, que comprende además un ácido nucleico molde.
6. Kit que comprende un oligonucleotido, comprendiendo el oligonucleótido un nucleótido modificado que no resuta eliminado por la actividad de exonucleasa 3' a 5' de una ácido nucleico polimerasa, en el que el nucleótido modificado no es el nucleótido 3' .
5. terminal ni en el penúltimo nucleótido del extremo 3' del oligonucleótido y el nucleótido modificado comprende una fracción 2' seleccionada de entre el grupo que consiste de amino, O-metilo, Ofosfato y fosforotioato, pero no comprende una fracción fluorescente, y que comprende además una ácido nucleico polimerasa que presenta actividad de exonucleasa 3' a 5'.
7. Kit según la reivindicación 6, en el que el nucleótido modificado se encuentra 3 a 10 nucleótidos del extremo 3' del oligonucleótido.
8. Método para detectar la presencia, ausencia o cantidad de una entidad biológica en una muestra biológica, comprendiendo el método:
a. poner en contacto un oligonucleótido con (i) una muestra biológica que se sospecha que presenta un ácido nucleico de la entidad biológica, e (ii) una ácido nucleico polimerasa que presenta actividad de exonucleasa 3' a 5', en el que el oligonucleótido comprende un nucleótido modificado que no resulta eliminado por la actividad de exonucleasa 3' a 5' y que no es el nucleótid.
3. terminal .
5. terminal ni en el penúltimo nucleótido desde el extremo 3' del oligonucleótido, y en el que dicho nucleótido modificado comprende una fracción 2' seleccionada de entre el grupo que consiste de amino, O-metilo, O-fosfato y fosforotioato y en el que el oligonucleótido es sustancialmente complementario al ácido nucleico de la entidad biológica,
b. llevar a cabo una reacción en cadena de la polimerasa, extendiendo de esta manera el oligonucléotido de una manera dependiente de molde para producir un producto polinucleótido complementario al ácido nucleico de la entidad biológica,
c. cuantificar el producto polinucleótido, o el complemento del mismo, y
d. correlacionar la cantidad de producto polinucleótido, o complemento del mismo, con la cantidad o presencia o ausencia de la entidad biológica en la muestra biológica.
9. Método según la reivindicación 8, en el que la etapa de cuantificación comprende una PCR en tiempo real cuantitativa.
10. Método según la reivindicación 8, en el que la PCR comprende una etapa bajo condiciones que permiten la extensión del oligonucleótido con cero, uno, dos o tres desapareamientos entre el oligonucleótido y el ácido nucleico vírico y en el que el oligonucleótido presenta uno o más desapareamientos con el ácido nucleico de la entidad biológica y en el que la actividad de exonucleasa 3' a 5' de la polimerasa edita el oligonucleótido resultando en un producto polinucleótido que es totalmente complementario al producto polinucleótido.
11. Método según la reivindicación 8, en el que la etapa de ejecución comprende además una o más polimerasas, algunas de las cuales no presentan sustancialmente actividad de exonucleasa 3' a 5'.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]