Amplificación por recombinasa-polimerasa.
Procedimiento de amplificación por recombinasa-polimerasa de amplificación de ADN de una molécula de ácidonucleico diana bicatenario,
que comprende una primera y una segunda cadena de ADN, que comprende las etapassiguientes
(a) poner en contacto un agente de recombinasa con un primer y un segundo cebador de ácido nucleico paraformar un primer y un segundo cebador de nucleoproteína que comprende una región monocatenaria en suextremo 3';
(b) poner en contacto los cebadores de nucleoproteína primero y segundo con dicha molécula de ácido nucleicodiana formando así una primera estructura bicatenaria en una primera porción de dicha primera cadena; yuna segunda estructura bicatenaria en una segunda porción de dicha segunda cadena;
(c) extender el extremo 3' de dichos cebadores de nucleoproteína primero y segundo con una o más polimerasasy dNTP para generar un primer y un segundo ácido nucleico bicatenario y una primera y una segunda cadenadesplazada de ácido nucleico; y
(d) continuar la reacción mediante repetición de (b) y (c) hasta que se alcanza un grado de amplificacióndeseado,
realizándose dicho procedimiento en las etapas (b) y (c) en presencia de un agente de agrupamiento de tal maneraque el agente de agrupamiento estimula la amplificación,
en el que además dicho agente de agrupamiento
(i) se selecciona de entre el grupo que consiste en polietilenglicol (PEG), dextrano, Ficoll, PEG1450, PEG3000,PEG8000, PEG10000, compuesto de PEG con un peso molecular de entre 15.000 y 20.000 Dalton y unacombinación de los mismos; y/o
(ii) está presente a una concentración de entre el 1% y el 12% en volumen o en peso de la reacción.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/IB2005/001560.
Solicitante: Alere San Diego, Inc.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 9975 Summers Ridge Road San Diego, CA 92121 ESTADOS UNIDOS DE AMERICA.
Inventor/es: ARMES,NIALL,A, STEMPLE,DEREK,L, PIEPENBURG,OLAF, WILLIAMS,COLIN H.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2425354_T3.pdf
Fragmento de la descripción:
Amplificación por recombinasa-polimerasa.
Antecedentes de la invención La capacidad para amplificar ADN se encuentra en el centro de la investigación biológica y médica moderna. Esto es así porque la mayoría de las técnicas de biología molecular se basan en muestras que contienen muchas moléculas idénticas para aumentar la sensibilidad de un ensayo o para preparar suficiente material para un procesamiento adicional. Entre las diversas técnicas de amplificación de ácido nucleico, la reacción en cadena de la polimerasa (PCR) es la más común debido a su sensibilidad y eficacia en la amplificación de secuencias de ácido nucleico cortas.
Aunque la PCR es de gran utilidad, también está limitada de varias maneras. La primera limitación de la PCR es que se basa en múltiples ciclos de fusión térmica (desnaturalización) a altas temperaturas seguidos por hibridación y elongación a una temperatura reducida. Para maximizar la eficacia y minimizar el ruido, se requiere un complejo control de temperatura de múltiples reacciones. Esto necesita la utilización de un bloque de calentamiento/enfriamiento rápido controlable por termociclador fabricado de material exótico (por ejemplo, bloques de plata recubiertos con oro) , o un mecanismo robótico para mover muestras entre zonas de temperatura controlada. Debido a la alta temperatura requerida para fundir el ADN en condiciones salinas fisiológicas, la tecnología de PCR requiere o bien la adición de polimerasa nueva por ciclo o bien la utilización de polimerasas termoestables. El enfoque de añadir polimerasa nueva no se ha automatizado y por tanto requiere mucho trabajo y es propenso a errores (por ejemplo, contaminación, caída de tubos, errores de etiquetado) . Además, la necesidad de añadir enzimas y de mezclar cada reacción individualmente presenta graves inconvenientes que presentan una adaptación limitada de procedimientos de PCR con adición de enzimas a pequeña escala.
En comparación con procedimientos que implican la adición de polimerasa nueva, la utilización de polimerasas termoestables en PCR es el más comúnmente empleado. Este enfoque presenta el inconveniente de que las polimerasas termoestables se encuentran en un número limitado de organismos, y los mecanismos de replicación utilizados por organismos termófilos se entienden mal. El repertorio disponible de polimerasas termoestables está limitado a enzimas polimerasas de un único polipéptido que participan en la reparación de ADN, y/o síntesis de la cadena retrasada. Las polimerasas de reparación de ADN y/o cadena retrasada son malas opciones para la amplificación de ADN porque muestran una escasa capacidad de procesamiento (síntesis distributiva) . En parte como consecuencia de la utilización de polimerasas de reparación y/o cadena retrasada (por ejemplo polimerasas Taq, Pfu, Vent) , y debido a la formación de estructuras de ácido nucleico secundarias o terciarias inhibidoras tras la fusión térmica, los protocolos de PCR actuales no amplifican fácilmente secuencias de más de varios miles de pares de bases. La síntesis (y amplificación) fiable de moldes más largos se basará en polimerasas y complejos enzimáticos auxiliares que muestren colectivamente niveles muy superiores de capacidad de procesamiento, desplazamiento de cadena y resolución de estructuras secundarias, así como que limiten la formación de estructuras de ácido nucleico de orden superior inhibidoras que pueden formarse al enfriar ADN desnaturalizado por calor.
Una segunda limitación de la PCR es que se basa en hibridación en disolución entre oligonucleótidos (cebadores de PCR) y ADN de molde desnaturalizado (es decir, el ADN que va a amplificarse) en un entorno acuoso. Para ser eficaces, las reacciones de PCR se realizan en un tiempo corto porque las polimerasas termoestables presentan una actividad que disminuye rápidamente a las temperaturas de PCR. Además, para una hibridación eficaz en un tiempo corto, una característica crítica para un resultado rápido, es necesario realizar la PCR en un entorno con altas concentraciones de oligonucleótidos. La alta concentración de oligonucleótidos también garantiza la rápida interacción de secuencias diana con los oligonucleótidos en competición con la cadena complementaria desnaturalizada por calor que todavía está presente en disolución. Las altas concentraciones de cebadores oligonucleotídicos pueden provocar problemas, particularmente cuando el número de copias de la secuencia diana es bajo y está presente en una mezcla compleja de moléculas de ADN. Este sería el caso, por ejemplo, en una PCR de un genoma para determinar el polimorfismo genético en un locus.
Un problema con la utilización de altas concentraciones de oligonucleótidos es que potencia el grado de falso cebado en secuencias apareadas sólo parcialmente en la mezcla de ADN compleja. El falso cebado se refiere a la hibridación de un cebador con un ADN de molde en PCR incluso cuando la secuencia del cebador no es completamente complementaria al ácido nucleico de molde, lo cual puede conducir a una amplificación no específica de ácidos nucleicos. El ruido, debido al falso cebado, aumenta con la concentración de oligonucleótidos y la complejidad del ADN de partida total. Además, la posibilidad de falso cebado aumenta a medida que disminuye el número de copias de las secuencias diana. Cuando las condiciones para el falso cebado son favorables (es decir, alta concentración de oligonucleótidos, alta complejidad, bajo número de copias) , secuencias amplificadas errantes pueden convertirse en un producto de reacción dominante. Por consiguiente, puede ser difícil identificar condiciones, y oligonucleótidos, para una amplificación limpia de secuencias diana a partir de un ADN de muestra sin un exceso de fondo de falso cebado. Por tanto una desventaja adicional de utilizar PCR es el limitado éxito para amplificar de manera limpia ADN diana poco frecuentes a partir de mezclas de secuencias complejas.
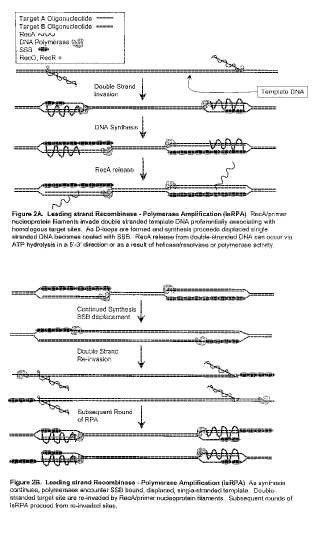
Una solución a los problemas de especificidad y al problema de fusión de molde que presenta la PCR es emplear procedimientos que se basen en las propiedades biológicas de la proteína recombinasa RecA bacteriana, o sus proteínas relacionadas procariotas y eucariotas. Estas proteínas recubren ADN monocatenario (ADNmc) para formar filamentos, que después barren ADN bicatenario (ADNbc) para detectar regiones de homología de secuencia. Cuando se localizan secuencias homólogas, la cadena de filamento de nucleoproteína invade el ADNbc creando un híbrido corto y una burbuja de cadena desplazada conocida como bucle D. El extremo 3’ libre de la cadena de filamento en el bucle D puede extenderse mediante ADN polimerasas para sintetizar una nueva cadena complementaria. La cadena complementaria desplaza a la cadena originalmente apareada a medida que se elonga. Utilizando pares de oligonucleótidos de una manera similar a la utilizada en PCR debe ser posible amplificar secuencias de ADN diana de una manera análoga pero sin ningún requisito de fusión térmica (termociclado) . Esto presenta la ventaja tanto de permitir la utilización de polimerasas termolábiles que anteriormente no podían utilizarse en PCR, como de aumentar la fidelidad y sensibilidad mediante barrido de molde e invasión de cadena en vez de hibridación.
Aunque la utilización de RecA y sus homólogos para la amplificación in vitro de ácidos nucleicos se ha descrito anteriormente (patente US 5.223.414 concedida a Zarling et al., mencionada en la presente memoria “Zarling”) , el procedimiento y los resultados son limitados. El procedimiento de Zarling presenta fallos críticos que limitan su capacidad para alcanzar una amplificación exponencial de ADN bicatenario. El fallo del procedimiento de Zarling en alcanzar la amplificación exponencial puede deberse a su especificación de la utilización de ATPyS en vez de ATP. El procedimiento de Zarling insta a utilizar ATPyS, en vez de ATP, en el ensamblaje de filamentos de nucleoproteína de RecA porque da como resultado una estructura de filamento de RecA/ADNmc más estable. Normalmente, los filamentos se ensamblan en el sentido de 5’ a 3’ y se desensamblarán espontáneamente en el mismo sentido de 5’ a 3’ a medida que RecA hidroliza ATP. Este procedimiento es dinámico porque el ensamblaje y el desensamblaje se producen al mismo tiempo y la cantidad de filamentos ensamblados está en equilibrio. Si se utiliza el análogo de ATP no hidrolizable, ATPyS, se inhiben la hidrólisis de ATPyS y el desensamblaje de 5’ a 3’ de los filamentos. La gran estabilidad de filamentos de RecA/ATPyS, tanto antes como después del intercambio de cadena, aunque es útil en el procedimiento de selección como diana (es decir, el procedimiento de... [Seguir leyendo]
Reivindicaciones:
1. Procedimiento de amplificación por recombinasa-polimerasa de amplificación de ADN de una molécula de ácido nucleico diana bicatenario, que comprende una primera y una segunda cadena de ADN, que comprende las etapas siguientes
(a) poner en contacto un agente de recombinasa con un primer y un segundo cebador de ácido nucleico para formar un primer y un segundo cebador de nucleoproteína que comprende una región monocatenaria en su extremo 3’;
(b) poner en contacto los cebadores de nucleoproteína primero y segundo con dicha molécula de ácido nucleico diana formando así una primera estructura bicatenaria en una primera porción de dicha primera cadena; y una segunda estructura bicatenaria en una segunda porción de dicha segunda cadena;
(c) extender el extremo 3’ de dichos cebadores de nucleoproteína primero y segundo con una o más polimerasas y dNTP para generar un primer y un segundo ácido nucleico bicatenario y una primera y una segunda cadena desplazada de ácido nucleico; y
(d) continuar la reacción mediante repetición de (b) y (c) hasta que se alcanza un grado de amplificación deseado,
realizándose dicho procedimiento en las etapas (b) y (c) en presencia de un agente de agrupamiento de tal manera que el agente de agrupamiento estimula la amplificación,
en el que además dicho agente de agrupamiento
(i) se selecciona de entre el grupo que consiste en polietilenglicol (PEG) , dextrano, Ficoll, PEG1450, PEG3000, PEG8000, PEG10000, compuesto de PEG con un peso molecular de entre 15.000 y 20.000 Dalton y una combinación de los mismos; y/o
(ii) está presente a una concentración de entre el 1% y el 12% en volumen o en peso de la reacción.
2. Procedimiento según la reivindicación 1, en el que dicho agente de agrupamiento se selecciona de entre el grupo que consiste en polietilenglicol (PEG) , dextrano, Ficoll, PEG1450, PEG3000, PEG8000, PEG10000, compuesto de PEG con un peso molecular comprendido entre 15.000 y 20.000 Dalton y una combinación de los mismos.
3. Procedimiento según la reivindicación 1, en el que las etapas (b) y (c) se realizan en presencia de uno o más agentes auxiliares seleccionados del grupo que consiste en proteína de unión monocatenaria, helicasa, resolvasa, RuvA, RuvB, RuvC, RecG, PriA, PriB, PriC, DnaT, DnaB, DnaC, DnaG, cargador de abrazadera de DnaX, complejo de núcleo de polimerasa, ADN ligasa, una abrazadera deslizante y una combinación de los mismos.
4. Procedimiento según la reivindicación 1, en el que dichos cebadores de ácido nucleico se seleccionan de entre el grupo que consiste en ADN, ARN, ANP, ANB, ácido nucleico con estructura principal de morfolino, ácido nucleico con estructura principal de fosforotiorato y una combinación de los mismos.
5. Procedimiento según la reivindicación 1, en el que dicho procedimiento se realiza en presencia de un agente estabilizante de filamentos de nucleoproteínas de RecA/ADNmc.
6. Procedimiento según la reivindicación 5, en el que dicho agente estabilizante se selecciona de entre el grupo que consiste en RecR, RecO, RecF y una combinación de los mismos.
7. Procedimiento según la reivindicación 1, en el que dicho procedimiento se realiza en presencia de un agente estabilizante monocatenario seleccionado del grupo que consiste en gp32, proteína SSB de E. coli, proteína gp32 de T4 y derivados de los mismos.
8. Procedimiento según la reivindicación 7, en el que el derivado de la proteína gp32 se selecciona de entre el grupo que consiste en gp32 con aminoácidos adicionales en el extremo N-terminal, gp32 con aminoácidos adicionales en el extremo C-terminal, gp32 con la sustitución de lisina 3 por un aminoácido distinto de lisina, y gp32 con la sustitución de arginina 4 por un aminoácido distinto de arginina y una combinación de los mismos.
9. Procedimiento según la reivindicación 7, en el que dicho derivado de gp32 se selecciona de entre el grupo que consiste en gp32 (N) , gp32 (C) , gp32 (C) K3A, gp32 (C) R4Q, gp32 (C) R4T, gp32K3A, gp32R4Q, gp32R4T y una combinación de los mismos.
10. Procedimiento según la reivindicación 1, en el que dicho procedimiento se realiza en presencia de una proteína de carga de recombinasa.
11. Procedimiento según la reivindicación 10, en el que la proteína de carga de recombinasa se selecciona de entre el grupo que consiste en uvsY de T4, recO de E. coli, recR de E. coli y una combinación de los mismos.
12. Procedimiento según la reivindicación 1, en el que dicho procedimiento de amplificación por recombinasapolimerasa comprende además un cofactor para el agente de recombinasa seleccionado del grupo que consiste en ATP, análogo de ATP y una combinación de los mismos.
13. Procedimiento según la reivindicación 1, en el que dicho procedimiento de amplificación por recombinasapolimerasa comprende además un sistema de regeneración de ATP para convertir ADP en ATP.
14. Procedimiento según la reivindicación 13, en el que dicho sistema de regeneración de ATP comprende fosfocreatina y creatina cinasa.
15. Procedimiento según la reivindicación 1, en el que dicho procedimiento de amplificación por recombinasapolimerasa comprende además pirofosfatasa para convertir pirofosfato en fosfato.
16. Procedimiento según la reivindicación 1, en el que una o más ADN polimerasa se seleccionan de entre el grupo que consiste en polimerasa procariota, polimerasa eucariota y polimerasa codificada por fago.
17. Procedimiento según la reivindicación 16, en el que dicha polimerasa eucariota se selecciona de entre el grupo que consiste en pol-a, pol-H, pol-a, pol-E y una combinación de las mismas.
18. Procedimiento según la reivindicación 16, en el que la polimerasa procariota se selecciona de entre el grupo que consiste en fragmento de Klenow de ADN polimerasa I de E. coli, ADN polimerasa gp43 de bacteriófago T4, fragmento grande de polimerasa I de Bacillus stearothermophilus, ADN polimerasa de Phi-29, ADN polimerasa de T7, fragmento grande de Pol I de Bacillus subtilis (polimerasa de Bsu) , ADN polimerasa I de E. coli, ADN polimerasa II de E. coli, ADN polimerasa III de E. coli, ADN polimerasa IV de E. coli, ADN polimerasa V de E. coli y una combinación de las mismas.
19. Procedimiento según la reivindicación 1, en el que el agente de recombinasa es sensible a la temperatura, en el que dicho agente de recombinasa sensible a la temperatura presenta actividad de invasión de cadena a una temperatura permisiva y ausencia de actividad de invasión de cadena a una temperatura no permisiva.
20. Procedimiento de amplificación por recombinasa-polimerasa anidada, que comprende las etapas siguientes:
(a) amplificar una región de ADN utilizando el procedimiento de amplificación por recombinasa-polimerasa según la reivindicación 1 utilizando un primer y un segundo cebador para producir un primer producto amplificado;
(b) amplificar dicho producto amplificado utilizando un tercer y un cuarto cebador utilizando el procedimiento de amplificación por recombinasa-polimerasa según la reivindicación 1 para producir un segundo producto amplificado, en el que dicho segundo producto amplificado es una secuencia más pequeña contenida dentro de dicho primer producto amplificado.
21. Procedimiento según la reivindicación 1, en el que cualquier ácido nucleico está marcado con un marcador detectable.
22. Procedimiento según la reivindicación 21, en el que dicho marcador se selecciona de entre el grupo que consiste en un fluorocromo, una enzima, un extintor de fluorescencia, un inhibidor enzimático, un marcador radiactivo y una combinación de los mismos.
23. Procedimiento según la reivindicación 1, en el que dicho agente de recombinasa es uvsX o uvsX de T4.
24. Procedimiento de amplificación por recombinasa-polimerasa de amplificación de ADN según la reivindicación 1, que comprende las etapas siguientes:
(a) combinar los siguientes reactivos en una reacción
(1) al menos una recombinasa;
(2) al menos una proteína de unión a ADN monocatenario;
(3) al menos una ADN polimerasa;
(4) dNTP o una mezcla de dNTP y ddNTP;
(5) un agente de agrupamiento;
(6) un tampón;
(7) un agente reductor;
(8) ATP o un análogo de ATP hidrolizable;
(9) al menos una proteína de carga de recombinasa;
(10) un primer cebador y opcionalmente un segundo cebador; y
(11) una molécula de ácido nucleico diana;
(b) incubar dicha reacción hasta que se alcanza un grado de amplificación deseado y en el que el agente de agrupamiento estimula la amplificación.
25. Procedimiento de amplificación por recombinasa-polimerasa de amplificación de ADN según la reivindicación 1, que comprende las etapas siguientes:
(a) combinar los siguientes reactivos en una reacción
(1) una recombinasa uvsX a una concentración comprendida entre 0, 2 y 12 lM;
(2) una proteína de unión a ADN monocatenario gp32 a una concentración comprendida entre 1 y 30 lM;
(3) polimerasa de Bsu a una concentración comprendida entre 500 y 5000 unidades por ml;
(4) dNTP o una mezcla de dNTP y ddNTP a una concentración comprendida entre 1-500 lM;
(5) polietilenglicol a una concentración comprendida entre el 1% y el 12% en peso o en volumen;
(6) tampón Tris-acetato a una concentración comprendida entre 25 mM y 60 mM;
(7) DTT a una concentración comprendida entre 1 mM - 5 mM;
(8) ATP a una concentración comprendida entre 1, 5 mM - 5 mM;
(9) uvsY a una concentración comprendida entre 0, 2 lM - 8 lM;
(10) un primer cebador y un segundo cebador, en el que dichos cebadores están a una concentración de entre 10 nM y 1 lM; y
(11) una molécula de ácido nucleico diana de al menos una copia;
(b) incubar dicha reacción hasta que se alcanza un grado de amplificación deseado; y en el que dicho polietilenglicol estimula la amplificación.
26. Procedimiento de amplificación por recombinasa-polimerasa de amplificación de ADN según la reivindicación 1, que comprende las etapas siguientes:
(a) combinar los siguientes reactivos en una reacción;
(1) recombinasa uvs.
10. 200 ng/ll;
(2) gp32 600 ng/ll;
(3) polimerasa de Bsu 20 ng/ll;
(4) dNTP 200 lM;
(5) DTT 1 mM;
(6) ATP o un análogo de ATP hidrolizable 3 mM;
(7) uvsY de 16 ng/ll a 60 ng/ll;
(8) 300 nM de un primer cebador y 300 nM de un segundo cebador;
(9) acetato de potasio 100 mM
(10) acetato de magnesio 8-16 mM
(11) fosfocreatina 25 mM;
(12) creatina cinasa 100 ng/ll
(b) liofilizar los reactivos de dicha etapa (a) para formar reactivos liofilizados;
(c) reconstituir dichos reactivos liofilizados con
(1) tampón Tris-acetato a una concentración de entre 1 mM y 60 mM;
(2) polietilenglicol a una concentración de entre el 1% y el 12% en peso o en volumen;
(3) un ácido nucleico diana;
(d) incubar dicha reacción hasta que se alcance un grado de amplificación deseado;
y en el que dicho polietilenglicol estimula la amplificación.
27. Procedimiento de amplificación por recombinasa-polimerasa de amplificación de ADN según la reivindicación 1, que comprende las etapas siguientes:
(a) combinar los siguientes reactivos en una reacción;
(1) recombinasa uvs.
10. 200 ng/ll;
(2) gp3.
30. 1000 ng/ll;
(3) polimerasa de Bsu o polimerasa de T4 10-50 ng/ll;
(4) dNT.
5. 500 lM;
(5) DTT de 0, 1 a 10 mM;
(6) ATP o un análogo de ATP hidrolizable 3 mM;
(7) uvsY de 16 ng/ll a 60 ng/ll;
(8) 50-1000 nM de un primer cebador .
5. 1000 nM de un segundo cebador;
(9) acetato de potasi.
4. 160 mM;
(10) acetato de magnesio 5-20 mM;
(11) fosfocreatina 10-40 mM;
(12) creatina cinas.
5. 200 ng/ll;
(b) liofilizar los reactivos de dicha etapa (a) para formar reactivos liofilizados;
(c) reconstituir dichos reactivos liofilizados con
(1) tampón Tris-acetato a una concentración de entre 1 mM y 60 mM;
(2) polietilenglicol a una concentración de entre el 1% y el 12% en peso o en volumen;
(3) un ácido nucleico diana;
(d) incubar dicha reacción hasta que se alcance un grado de amplificación deseado;
y en el que dicho polietilenglicol estimula la amplificación.
28. Procedimiento de amplificación por recombinasa-polimerasa de amplificación de ADN según la reivindicación 1, que comprende las etapas siguientes:
(a) combinar los siguientes reactivos en una reacción;
(1) recombinasa uvs.
10. 200 ng/ll;
(2) gp3.
30. 1000 ng/ll;
(3) polimerasa de Bsu o polimerasa de T4 10-50 ng/ll;
(4) dNT.
5. 500 lM;
(5) DTT de 0, 1 a 10 mM;
(6) ATP o un análogo de ATP 3 mM;
(7) uvsY de 16 ng/ll a 60 ng/ll;
(8) 50-1000 nM de un primer cebador .
5. 1000 nM de un segundo cebador;
(9) acetato de potasi.
4. 160 mM;
(10) acetato de magnesio 5-20 mM;
(11) fosfocreatina 10-40 mM;
(12) creatina cinas.
5. 200 ng/ll;
(13) polietilenglicol a una concentración de entre el 1% y el 12% en peso o en volumen
(b) liofilizar los reactivos de dicha etapa (a) para formar reactivos liofilizados;
(c) reconstituir dichos reactivos liofilizados con
(1) tampón Tris-acetato a una concentración de entre 1 mM y 60 mM;
(2) un ácido nucleico diana;
(d) incubar dicha reacción hasta que se alcance un grado de amplificación deseado;
y en el que dicho polietilenglicol estimula la amplificación.
29. Procedimiento según cualquiera de las reivindicaciones anteriores, en el que los cebadores están marcados con un marcador detectable.
30. Procedimiento según cualquiera de las reivindicaciones anteriores, que comprende además detectar la molécula de ácido nucleico diana amplificado.
31. Procedimiento según la reivindicación 30, en el que dicha detección se realiza utilizando una tira reactiva de flujo lateral para evaluar si se han asociado físicamente dos cebadores marcados dentro del ácido nucleico diana amplificado.
32. Procedimiento según cualquiera de las reivindicaciones anteriores, que comprende convertir en primer lugar ARN 10 en dicha molécula de ácido nucleico diana bicatenario utilizando actividad de transcriptasa inversa.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]