Examen genético fetal no invasivo mediante análisis digital.
Método de detección de aneuploidía fetal en una mezcla de material genético materno y fetal,
en unamuestra de tejido materno, caracterizado por:
(a) distribuir el material genético en muestras de reacción, en las que el ADN que va a analizarse estará obien presente o bien ausente en una muestra de reacción, debido a variaciones aleatorias entre muestrasde reacción;
(b) medir la presencia de diferentes secuencias diana en las muestras de reacción mediante análisis digitalpara obtener resultados binarios que proporcionan la detección diferencial de las secuencias diana en unamezcla de material genético materno y fetal, en el que dichas secuencias diana comprenden secuencias dedos cromosomas, uno de los cuales es posiblemente aneuploide y uno de los cuales es presumiblementediploide;
(c) analizar los resultados binarios de la etapa (b) contando la frecuencia de respuestas positivas a partir desecuencias diana seguido por
(d) análisis estadístico de los resultados de la etapa (c) mediante lo cual la frecuencia de respuestaspositivas a partir de secuencias diana proporciona datos suficientes para distinguir secuencias dianaeuploides de aneuploides
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2007/003209.
Solicitante: THE BOARD OF TRUSTEES OF THE LELAND STANFORD JUNIOR UNIVERSITY.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 1705 EL CAMINO REAL PALO ALTO CA 94306-1106 ESTADOS UNIDOS DE AMERICA.
Inventor/es: QUAKE,STEPHEN, FAN,HEI-MUN CHRISTINA.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12P19/34 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2429408_T3.pdf
Fragmento de la descripción:
Examen genético fetal no invasivo mediante análisis digital
Referencia cruzada a solicitudes relacionadas Esta solicitud reivindica prioridad de la solicitud de patente provisional estadounidense n.º 60/764.420 presentada el 2 de febrero de 2006.
Declaración de apoyo gubernamental
Esta invención se realizó con el apoyo del gobierno estadounidense. El gobierno estadounidense puede tener determinados derechos en esta invención.
Referencia a la lista de secuencias Los solicitantes afirman que la copia en papel de la lista de secuencias es idéntica a la lista de secuencias en forma legible por ordenador que se encuentra en el disco de ordenador adjunto. Los solicitantes incorporan el contenido de la lista de secuencias como referencia en su totalidad.
Antecedentes de la invención Campo de la invención La presente invención se refiere al campo del examen genético fetal y al campo del análisis cuantitativo de ácidos nucleicos.
Técnica relacionada Se reconoce ahora que se libera ADN fetal de la placenta y se mezcla con la sangre de la madre a niveles bastante altos, entre el 3% y el 6% del ADN en la sangre de la madre procede del feto. Esta observación se ha usado conjuntamente con ensayos de PCR para una variedad de exámenes genéticos fetales, género, Rh y talasemia. Sin embargo, la técnica sigue siendo limitada por dos motivos principales: en primer lugar, los ensayos de PCR sacrifican sensibilidad por especificidad, haciendo difícil identificar mutaciones particulares, y en segundo lugar, el trastorno genético más común, el síndrome de Down, es una trisomía cromosómica y por tanto no puede detectarse mediante PCR convencional en una muestra mixta.
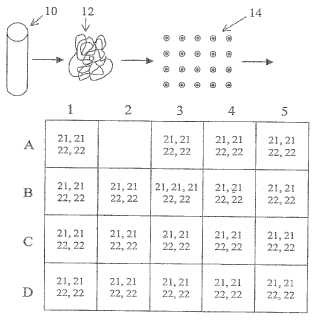
Se ha encontrado ahora que estos problemas pueden solucionarse mediante el examen cuantitativo de grandes números de muestras de cromosomas a través del uso de técnicas altamente escalables. Este enfoque se denomina en el presente documento “análisis digital”, e implica la separación del material genómico extraído en unidades diferenciadas de modo que la detección de una secuencia diana (por ejemplo, cromosoma 21) puede cuantificarse simplemente como binario (0, 1) o múltiplos sencillos, 2, 3, etc. El ejemplo principal de una técnica que puede usarse para producir tales resultados “digitales” es la “PCR digital”, que permite una amplificación eficaz a partir de moléculas individuales, seguido por análisis cuantitativo posterior. La PCR digital, tal como se usa el término en el presente documento, se refiere a una dilución cuantitativa, limitada de una muestra de ácido nucleico, tal como en placas de múltiples pocillos, luego la amplificación de una molécula de ácido nucleico en un pocillo, que debido a la dilución, debe ser o bien 0 o bien 1 molécula. La PCR digital usando placas de múltiples pocillos se ha usado anteriormente para detectar mutaciones poco comunes o bien mediante análisis en serie de amplicones de una molécula individual (es decir, clonal) (Vogelstein B, Kinzler KW. Proc Natl Acad Sci U S A. 3 de agosto de 1999; 96 (16) : 9236-41) o bien mediante potenciación de la sensibilidad de la amplificación diferencial (http://www.fluidigm.com/didIFC.htm) . Se describe a continuación una invención mediante la cual puede aplicarse PCR digital a diagnósticos fetales no invasivos con el fin de detectar mutaciones fetales con especificidad y sensibilidad más allá de los que es posible con análisis de PCR convencional.
Además, como también se describe en relación con la invención descrita a continuación, la PCR digital puede usarse para detectar aneuploidía, tal como la trisomía que provoca el síndrome de Down. Puesto que las aneuploidías no presentan un cambio mutacional en la secuencia, y son meramente un cambio en el número de cromosomas, no ha sido posible detectarlas en un feto sin recurrir a técnicas invasivas tales como amniocentesis o toma de muestras de vellosidades coriónicas (Science 309, 2 de septiembre de 2005 págs. 1476-8) .
Otra forma de PCR digital se ha descrito como PCR en emulsión, que se ha usado para preparar pequeñas perlas con ADN amplificado de manera clonal (en esencia, cada perla contiene un amplicón de PCR digital) . (Dressman et al, Proc Natl Acad Sci USA. 100, 8817 (22 de julio de 2003) ) .
Otra forma de PCR digital puede llevarse a cabo usando dispositivos microfluídicos. En esta realización, descrita a continuación, se diluye ADN y se separa en muestras pequeñas, diferenciadas para formar muestras de reacción mediante una serie canales y válvulas.
Un ejemplo de un método adecuado para el análisis de moléculas individuales que puede adaptarse a los presentes
métodos se facilita en Braslavsky et al., “Sequence information can be obtained from single DNA molecules”, Proc. Nat. Acad. Sci. 100 (7) : 3960-3964 (2003) , que usa incorporación secuencial de nucleótidos marcados sobre un molde de ADN monocatenario inmovilizado y monitorización mediante microscopía de fluorescencia.
Otro aspecto de la técnica relevante implica la preparación de muestras con el fin de llevar a cabo los presentes procedimientos. Es decir, puede enriquecerse el ADN fetal en relación con el ADN materno. Chan, et al., “Size Distribution of Maternal and Fetal DNA in Maternal Plasma”, Clin. Chem. 50 (1) : 88-92 (2004) notifican que las moléculas de ADN en plasma son principalmente fragmentos de ADN cortos. Los fragmentos de ADN en el plasma de mujeres embarazadas son significativamente más largos que los fragmentos de ADN de mujeres no embarazadas, y más largos que el ADN fetal.
Publicaciones y patentes relacionadas Vogelstein et al., “Digital Amplification”, documento US 6.440.706, expedido el 27 de agosto de 2002, dan a conocer la identificación de mutaciones predefinidas que se espera que estén presentes en una fracción minoritaria de una población celular.
Lo, “Fetal DNA in Maternal Plasma: Biology and Diagnostic Applications”, Clin. Chem. 46:1903-1906 (2000) da a conocer la demostración de ADN fetal en plasma materno. Los autores encontraron un nivel fraccional medio del 3, 4% de ADN fetal en ADN materno en el plasma durante el embarazo temprano. Los autores notifican la detección del gen RhD y polimorfismos de microsatélites en el plasma de mujeres embarazadas.
Li et al., “Detection of Paternally Inherited Fetal Point Mutations for !-Thalassemia Using Size Fractionated Cell-Free DNA in Maternal Plasma”, J. Amer. Med. Assoc. 293:843-849 (16 de febrero de 2005) dan a conocer que el análisis de ADN fetal libre de células en plasma materno ha demostrado ser notablemente fiable para la evaluación de loci fetales ausentes del genoma materno, tales como secuencias específicas del cromosoma Y o el gen RhD en mujeres embarazadas que son negativas para Rh. Los autores notifican la extracción y el fraccionamiento por tamaño de ADN de plasma materno usando electroforesis en gel de agarosa. Entonces, se usaron ácidos nucleicos peptídicos (PNA) para unirse específicamente a un alelo materno para suprimir la amplificación por PCR del alelo materno de tipo natural, enriqueciendo de ese modo la presencia de secuencias mutantes heredadas por vía paterna. Se examinaron cuatro mutaciones puntuales distintas en el gen de !-globina. Se encontró que la etapa de PNA era necesaria para la detección de alelos mutantes usando PCR específica de alelo.
Lo et al., “Quantitative Analysis of Fetal DNA in Maternal Plasma and Serum: Implications for Noninvasive Prenatal Diagnosis”, Am. J. Hum. Genet. 62:768-775 (1998) dan a conocer un ensayo de PCR cuantitativa en tiempo real para medir la concentración de ADN fetal en suero y plasma materno. Los autores encontraron una media de 25, 4 equivalentes de genoma/ml de ADN fetal en embarazo temprano. Esto corresponde a aproximadamente el 3, 4% del ADN total en embarazo temprano.
Chan et al., “Size Distribution of Maternal and Fetal DNA in Maternal Plasma”, Clin Chem. 50:89-92 (enero de 2004) investigaron la distribución de tamaño de ADN en plasma en mujeres no embarazadas y mujeres embarazadas, usando un panel de ensayos de PCR cuantitativa con diferentes tamaños de amplicones que seleccionan como diana el gen leptina. Encontraron que los fragmentos de ADN en el plasma de mujeres embarazadas son significativamente más largos que aquellos en el plasma de mujeres no embarazadas, y las moléculas de ADN derivadas de la madre son más largas que las derivadas del feto.
Tufan et al., “Analysis of Cell-Free Fetal DNA from Maternal Plasma and Serum Using a Convencional Multiplex PCR: Factors Influencing Success”, Turk. J. Med Sci. 35: 85-92 (2005) compararon... [Seguir leyendo]
Reivindicaciones:
1. Método de detección de aneuploidía fetal en una mezcla de material genético materno y fetal, en una muestra de tejido materno, caracterizado por:
(a) distribuir el material genético en muestras de reacción, en las que el ADN que va a analizarse estará o bien presente o bien ausente en una muestra de reacción, debido a variaciones aleatorias entre muestras de reacción;
(b) medir la presencia de diferentes secuencias diana en las muestras de reacción mediante análisis digital para obtener resultados binarios que proporcionan la detección diferencial de las secuencias diana en una mezcla de material genético materno y fetal, en el que dichas secuencias diana comprenden secuencias de dos cromosomas, uno de los cuales es posiblemente aneuploide y uno de los cuales es presumiblemente diploide;
(c) analizar los resultados binarios de la etapa (b) contando la frecuencia de respuestas positivas a partir de secuencias diana seguido por
(d) análisis estadístico de los resultados de la etapa (c) mediante lo cual la frecuencia de respuestas
positivas a partir de secuencias diana proporciona datos suficientes para distinguir secuencias diana euploides de aneuploides.
2. Método según la reivindicación 1, en el que la etapa de medición comprende la secuenciación directa del material genético materno y fetal.
3. Método según la reivindicación 1 ó 2, en el que una de las diferentes secuencias diana es diploide en
material genético materno y aneuploide en material genético fetal y otra de las diferentes secuencias diana es diploide en material genético tanto materno como fetal.
4. Método según la reivindicación 1 ó 2, en el que el tejido materno es plasma sanguíneo o sangre periférica materna.
5. Método según la reivindicación 1 ó 2, en el que el número de muestras de reacción es de al menos 25 aproximadamente 10.000 y/o en el que el material genético es ADNc derivado de ARN en el tejido.
6. Método según la reivindicación 1 o la reivindicación 2, en el que las muestras de reacción se seleccionan del grupo que consiste en: pocillos en una placa de microtitulación, fases acuosas en una emulsión, una zona sobre una superficie de matriz y cámaras de reacción en un dispositivo microfluídico.
7. Método según la reivindicación 1, en el que las muestras de reacción se ponen en contacto con una
pluralidad de cebadores de PCR, incluyendo al menos un cebador dirigido específicamente a una secuencia materna y al menos un cebador dirigido específicamente a una secuencia fetal, y que incluye además la etapa de amplificar la secuencia materna y la secuencia fetal.
8. Método según la reivindicación 7, en el que dicha medición comprende la hibridación de una secuencia diana con un ácido nucleico que tiene un marcador fluorescente.
9. Método según la reivindicación 8, en el que dicho ácido nucleico está en una muestra para unirse a una secuencia diana y generar un cambio resultante en la fluorescencia.
10. Método según la reivindicación 1 o reivindicación 2, en el que dicha medición comprende el uso de ácidos nucleicos específicos para el cromosoma 21 humano y específicos para otro cromosoma humano.
11. Método según la reivindicación 1 ó 2, en el que dicha medición comprende la hibridación con ácidos
nucleicos que se hibridan con una secuencia seleccionada del grupo que consiste en uno o más de los genes: CFTR, factor VIII (gen F8) , beta globina, hemocromatosis, G6PD, neurofibromatosis, GAPDH, beta amiloide y piruvato cinasa.
12. Método según la reivindicación 1 o la reivindicación 2, en el que dicha medición detecta una secuencia delecionada en una deleción de cromosoma humano, movida en una translocación o inversión, o duplicada 45 en una duplicación cromosómica, en el que dicha secuencia se caracteriza en un trastorno genético conocido porque el material genético fetal no está presente en el material genético materno.
13. Método según la reivindicación 1 ó 2, en el que dicha medición detecta una secuencia de una forma mutada de un gen humano que tiene una anomalía conocida.
14. Método según la reivindicación 1, que comprende además la etapa de enriquecer la mezcla en material
genético fetal mediante separación por tamaño, mediante lo cual se usa una preparación que comprende sólo fragmentos de ADN inferiores a aproximadamente 300 pb para medir en la etapa (b) .
15. Método según la reivindicación 2, en el que la secuenciación comprende secuenciación de una única molécula.
16. Método según la reivindicación 2, en el que la secuenciación comprende secuenciación masiva en paralelo.
17. Método según la reivindicación 1, caracterizado además por las etapas de:
a) distribuir la mezcla de material genético materno y fetal en al menos quinientas muestras de reacción separadas, conteniendo cada muestra de reacción menos de aproximadamente una molécula de secuencia diana;
b) hibridar el ADN en cada muestra de reacción separada con ácidos nucleicos que se hibridan con una de dos diferentes secuencias diana, una de las cuales se usa como secuencia de control en un cromosoma presumiblemente diploide para detectar dianas presentes por igual en ADN tanto materno como fetal y la otra se usa para detectar dianas posiblemente no presentes por igual en ADN tanto materno como fetal en un cromosoma posiblemente aneuploide con el fin de detectar una aneuploidía fetal; y
c) analizar los resultados de dicho marcaje para obtener resultados que distinguen una diferencia en los números de secuencias diana en el cromosoma presumiblemente diploide y el cromosoma posiblemente aneuploide.
18. Método según la reivindicación 17, en el que la distribución es en al menos 10.000 muestras de reacción separadas, en el que dicha diferencia en secuencias diana es una aneuploidía fetal.
19. Método según la reivindicación 17, en el que la distribución es en al menos 10.000 muestras de reacción separadas, en el que dichas sondas comprenden una sonda específica para el cromosoma 21 humano.
Fig. 4
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]