Composiciones y métodos de detección.
Un conjunto de perlas para detectar una cepa del VPH y/o para diferenciar entre dos o más cepas del VPH,
en los que el conjunto de perlas comprende una pluralidad de familias o una pluralidad de subconjuntos de perlas que tienen:
(a) al menos dos subconjuntos de perlas, en los que cada subconjunto de perlas se puede distinguir fisioquímicamente de cualquier otro subconjunto de perlas en base, al menos, al tamaño, o
(b) al menos un subconjunto de perlas que tienen al menos dos familias, en el que las perlas en un subconjunto de perlas son homogéneas en tamaño unas con respecto a otras, y se pueden distinguir fisioquímicamente en dos o más familias de perlas en base al nivel de marcado que contiene cada una, o
(c) una combinación de dichas familias de perlas y dichos subconjuntos de perlas;
y en los que:
las perlas dentro de cada familia de perlas de un subconjunto están cada una acopladas a una sonda de captura de ácido nucleico opcionalmente marcada que se puede unir a una región específica de cepa de VPH de un genoma de VPH u, opcionalmente, a al menos una de una secuencia de ácido nucleico de control, siempre que cualquier sonda de captura de ácido nucleico o secuencia de ácido nucleico de control esté marcada con la misma marca que otras sondas de captura marcadas o secuencia de ácido nucleico en el subconjunto de perlas, y que dentro de cualquier subconjunto de perlas individual, cada familia de perlas del mismo tiene una intensidad fluorescente diferente, en los que la sonda de captura de ácido nucleico se selecciona de SEQ ID NOs:46-63; y
en los que la región específica de cepa del VPH de un genoma del VPH es un amplicón generado amplificando ácido nucleico a partir de una muestra biológica que se ha obtenido de un sujeto humano, que comprende putativamente VPH, en los que el amplicón es distinto para dicho VPH o una cepa particular de dicho VPH, y en los que el amplicón se genera usando un par de cebadores de amplificación de oligonucleótidos de un cebador directo y un cebador inverso, en los que el cebador directo del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:1-8, y el cebador inverso del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:9-12, y en los que al menos uno de los cebadores directos e inversos está conjugado a una protuberancia en el extremo 5' terminal del cebador; y
en los que cada familia de perlas es específica para la detección de una cepa de VPH diferente de cualquier otra familia de perlas en el conjunto de perlas; en los que dicho conjunto de perlas puede identificar una o más cepas de VPH específicas por medio de análisis del tamaño de perla, intensidad fluorescente, o discriminación de secuencias de la al menos una familia de perlas o subconjunto de perlas, usando citometría de flujo.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2008/004441.
Solicitante: GENERA BIOSYSTEMS LIMITED.
Nacionalidad solicitante: Australia.
Dirección: 1 DALMORE DRIVE SCORESBY, VICTORIA 3179 AUSTRALIA.
Inventor/es: PARK, DANIEL J., KHAN,ZAHEER, POETTER,KARL F.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/70 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen virus o bacteriófagos.
- G01N33/53 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › Ensayos inmunológicos; Ensayos en los que interviene la formación de uniones bioespecíficas; Materiales a este efecto.
PDF original: ES-2403729_T3.pdf
Fragmento de la descripción:
Composiciones y métodos de detección
Campo de la invención La presente invención se refiere, en general, al campo de ensayos de diagnóstico y detección. Más en particular, la presente invención proporciona métodos y reactivos incluidos biochips para detectar la presencia de, o distinguir entre, uno o más analitos en una muestra.
Antecedentes de la invención Los detalles bibliográficos de referencias proporcionadas en la memoria descriptiva objeto se enumeran al final de la memoria descriptiva.
Las referencias a cualquier técnica anterior en esta memoria descriptiva no son, y no deberían considerarse como, un reconocimiento de ninguna forma de sugerencia de que la técnica anterior forma parte del saber popular en ningún país.
La necesidad de métodos de cribado rápidos y fiables para detectar múltiples analitos en un ensayo individual es vital no sólo en los campos de diagnóstico clínico sino también para su uso en el cribado, por ejemplo, para el cribado de fármacos y toxinas ambientales.
Un área de este tipo que necesita urgentemente métodos de cribado mejorados y reactivos está en el campo de las enfermedades infecciosas. Por ejemplo, una estimación conservadora del uso mundial de pruebas de diagnóstico para enfermedades de transmisión sexual, tales como el virus del papiloma humano (VPH) , es de aproximadamente 20.000.000 pruebas por año.
Muchas de las pruebas existentes para el cribado de las causas de las enfermedades infecciosas consumen tiempo, son laboriosas, caras, a menudo específicas sólo para un patógeno y/o no pueden diferenciar entre diferentes cepas de patógenos.
El VPH es el principal patógeno causante de cáncer cervical. Sin embargo, el taxón del VPH comprende muchas "cepas" del patógeno, de las que sólo algunas están asociadas con el desarrollo de cáncer cervical y otros carcinomas. En consecuencia, las cepas de VPH se clasifican típicamente como cepas de "alto riesgo", incluidas las 13 cepas que representan aproximadamente el 98 % de los casos de cáncer cervical, o cepas de "bajo riesgo" que no están típicamente asociadas con el desarrollo de cáncer cervical. El documento WO 2006/060872 A1 divulga un ensayo para el VPH en sujetos humanos para la diferenciación entre taxones del VPH para distinguir infecciones por VHP de "alto riesgo" de las infecciones de "bajo riesgo".
Actualmente, el cáncer cervical se detecta por un frotis de Papanicolaou. En esta técnica, se recogen células del cérvix por raspado o lavado. A continuación, estas células se sitúan sobre un portaobjetos de microscopio de vidrio para producir el "frotis". Después, un patólogo examina el portaobjetos, buscando células aberrantes. El frotis de Papanicolaou, sin embargo, es de algún modo un ensayo no satisfactorio para determinar de forma inequívoca un riesgo de cáncer cervical, ya que la técnica tiene una tasa de falso negativo de aproximadamente el 20 % y la técnica no puede distinguir el taxón de "alto riesgo" y de "bajo riesgo".
Muchos de los reactivos y/o métodos para la detección del VPH sufren de altos costes asociados y/o consumen cantidades significativas de tiempo hasta su terminación. Se necesitan análisis más rápidos y/o simplificados con menores costes globales y una aplicación más fácil. La presente invención se refiere a estos y otros fines importantes.
Sumario de la invención La presente invención proporciona ensayos que permiten la detección de uno o más analitos y/o que diferencian entre miembros dentro de una clase de analitos. En particular, se emplea un análisis de multiplexación basado en las propiedades de los analitos y de los componentes del ensayo para identificar o distinguir entre analitos.
En consecuencia, determinados aspectos de la presente invención proporcionan conjuntos de perlas para detectar uno o más analitos y/o para diferenciar entre dos o más miembros dentro de una clase de analitos, en los que el conjunto de perlas comprende una pluralidad de familias o subconjuntos de perlas en el que:
(a) las perlas de cada subconjunto son homogéneas con respecto al tamaño;
(b) las perlas dentro de cada subconjunto se acoplan a un reactivo que reaccionará específicamente con un analito de interés dado en una muestra que se va a probar;
(c) el reactivo sobre cada perla se marca con la misma marca con cada subconjunto de perlas que tienen una intensidad fluorescente diferente para crear una mezcla heterogénea de subconjuntos de perlas basada en la intensidad fluorescente; y
(d) al menos dos subconjuntos de perlas se mezclan juntas para producir un conjunto de perlas, en el que la identidad del subconjunto y por lo tanto el reactivo al que se ha acoplado la perla se puede identificar por citometría de flujo en base al tamaño, intensidad fluorescente y discriminación de analitos.
En otros aspectos, la presente invención contempla métodos in vitro para detectar y/o diferenciar entre dos o más analitos en una muestra, que comprenden las etapas de:
(a) poner en contacto la muestra con un conjunto de perlas específicas para los analitos de interés;
(b) incubar dicho conjunto de perlas con dicha muestra durante un tiempo y bajo condiciones suficientes para permitir que dicho (s) analito (s) en dicha muestra reacciones específicamente con un reactivo sobre una perla dentro de dicho conjunto de perlas; y
(c) detectar y/o diferenciar analitos en la muestra que se unen a un reactivo sobre dicha perla.
En algunos otros aspectos, los reactivos se pueden marcar con uno o más fluorocromos que permiten adicionalmente la diferenciación entre miembros dentro de una clase de analitos. En determinados aspectos preferidos, la presente invención proporciona métodos y conjuntos de perlas que pueden detectar y/o distinguir entre analitos dentro de una muestra biológica, en los que los analitos son específicos para un patógeno infeccioso.
Las muestras biológicas contempladas en el presente documento incluyen sangre, suero, saliva, heces, orina, fluido tisular, semen, exudado, pus y fluido respiratorio y moco y frotis de llagas, cánceres y lesiones tópicas.
El término "patógeno" se refiere a un microorganismo o virus que infecta o coloniza una muestra. Los patógenos ejemplares incluyen virus, bacterias, hongos y microorganismos eucariotas. Un virus incluye un Lentivirus (por ejemplo virus del sida, VIH-I, VIH-II, HTLV-IV) , Retrovirus y virus de la gripe aviar. En algunas realizaciones preferidas, "patógeno" incluye un microorganismo o virus que infecta un organismo multicelular tal como un animal o una planta. En consecuencia, en algunas realizaciones, el analito se puede considerar como un patógeno de animal
o de planta. Sin embargo, la presente invención engloba la detección y/o diferenciación de entidades no patógenas que colonizan organismos multicelulares tales como simbiontes, endófitos, colonizadores gastrointestinales, y similares. Los métodos de la presente invención también son aplicables a la detección de analitos en una muestra que sea indicativa de la presencia de agentes terapéuticos o sustancias de abuso. Los métodos y reactivos de la presente invención también se pueden usar en la detección de un analito en una muestra que no se deriva de una muestra biológica aislada a partir de un animal o una planta. Como tales, los reactivos y métodos de la presente invención también se extienden a la detección de uno o más analitos y/o diferenciación entre analitos en muestras ambientales, incluidas muestras de aire, agua y tierra, incluidas muestras extraterrestres de tierra, polvo o similar, muestras industriales y similares, además de las muestras biológicas enumeradas anteriormente.
En determinadas realizaciones, la presente invención proporciona métodos in vitro de diagnóstico y reactivos para VPH en sujetos humanos y puede detectar y diferenciar entre cepas diferentes para distinguir un taxón del VPH de "alto riesgo" de un taxón del VPH de "bajo riesgo". En consecuencia, en algunas realizaciones preferidas, la presente invención proporciona conjuntos de perlas que pueden distinguir entre pluralidades de cepas de VPH diferentes. Como tales, en un aspecto, los analitos son específicos para una pluralidad de taxones del VPH y los métodos y/o reactivos son específicos para la detección de ácido nucleico o antígenos o anticuerpos que sean específicos para la pluralidad de taxones del VPH.
Aún más preferido, los cebadores o sondas de ácido nucleico que se pueden unir a una porción específica de cepa de un genoma del VPH se inmovilizan sobre perlas en cada subconjunto de perlas. Los cebadores dirigidos a regiones conservadas dentro de un genoma del VPH que... [Seguir leyendo]
Reivindicaciones:
1. Un conjunto de perlas para detectar una cepa del VPH y/o para diferenciar entre dos o más cepas del VPH, en los que el conjunto de perlas comprende una pluralidad de familias o una pluralidad de subconjuntos de perlas que tienen:
(a) al menos dos subconjuntos de perlas, en los que cada subconjunto de perlas se puede distinguir fisioquímicamente de cualquier otro subconjunto de perlas en base, al menos, al tamaño, o
(b) al menos un subconjunto de perlas que tienen al menos dos familias, en el que las perlas en un subconjunto de perlas son homogéneas en tamaño unas con respecto a otras, y se pueden distinguir fisioquímicamente en dos o más familias de perlas en base al nivel de marcado que contiene cada una, o
(c) una combinación de dichas familias de perlas y dichos subconjuntos de perlas;
y en los que:
las perlas dentro de cada familia de perlas de un subconjunto están cada una acopladas a una sonda de captura de ácido nucleico opcionalmente marcada que se puede unir a una región específica de cepa de VPH de un genoma de VPH u, opcionalmente, a al menos una de una secuencia de ácido nucleico de control, siempre que cualquier sonda de captura de ácido nucleico o secuencia de ácido nucleico de control esté marcada con la misma marca que otras sondas de captura marcadas o secuencia de ácido nucleico en el subconjunto de perlas, y que dentro de cualquier subconjunto de perlas individual, cada familia de perlas del mismo tiene una intensidad fluorescente diferente, en los que la sonda de captura de ácido nucleico se selecciona de SEQ ID NOs:46-63; y
en los que la región específica de cepa del VPH de un genoma del VPH es un amplicón generado amplificando ácido nucleico a partir de una muestra biológica que se ha obtenido de un sujeto humano, que comprende putativamente VPH, en los que el amplicón es distinto para dicho VPH o una cepa particular de dicho VPH, y en los que el amplicón se genera usando un par de cebadores de amplificación de oligonucleótidos de un cebador directo y un cebador inverso, en los que el cebador directo del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:1-8, y el cebador inverso del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:912, y en los que al menos uno de los cebadores directos e inversos está conjugado a una protuberancia en el extremo 5' terminal del cebador; y
en los que cada familia de perlas es específica para la detección de una cepa de VPH diferente de cualquier otra familia de perlas en el conjunto de perlas; en los que dicho conjunto de perlas puede identificar una o más cepas de VPH específicas por medio de análisis del tamaño de perla, intensidad fluorescente, o discriminación de secuencias de la al menos una familia de perlas o subconjunto de perlas, usando citometría de flujo.
2. El conjunto de perlas de la reivindicación 1, en el que la protuberancia del cebador directo se selecciona del grupo que consiste en:
CAATCAGC, ACAAT, GGAACAAT, GGAAC, CAGCTT, ATTACC, CTGTT, /5Phos/CAATCAGC, /5Phos/ACAAT, /5Phos/GGAACAAT, /5Phos/GGAAC, /5Phos/CAGCTT, 5Phos/ATTACC, y /5Phos/CTGTT.
4. El conjunto de perlas de una de las reivindicaciones 1-3 en el que las perlas marcadas se marcan con un fluorocromo seleccionado del grupo que consiste en: hidroxicumarina, aminocumarina, metoxicumarina, azul Cascade, amarillo Lucifer, NBD, ficoeritrina (PE) , PerCP, aloficocianina, Hoechst 33342, DAP1, azul SYTOX, Hoechst 33258, cromomicina A3, mitramicina, YOYO-1, verde SYTOX, naranja SYTOX, 7-AAD, naranja de acridina, TOTO-1, To-PRO-1, naranja de tiazol, TOTO-3, TO-PRO-3, LDS 751, Alexa Fluro-350, Alexa Fluro-430, Alexa Fluro488, Alexa Fluro-532, Alexa Fluro-546, Alexa Fluro-555, Alexa Fluro-556, Alexa Fluro-594, Alexa Fluro-633, Alexa Fluro-647, Alexa Fluro-660, Alexa Fluro-680, Alexa Fluro-700 y Alexa Fluro-750, BoDipy 630/650 y BoDipy 650/665, los colorantes CY Cy2, Cy3, Cy3, 5, Cy5, Cy-5, 5 y Cy7, 6-FAM (fluoresceína) , PE-Cy5, PE-Cy7, fluoresceína dT, Hexaclorofluoresceína (Hex) , 6-carboxi-4', 5'-dicloro-2', 7'-dimetoxifluoresceína (JOE) , colorantes Oregon Gree.
48. X y 514, colorantes rodamina X-rodamina, lisamina rodamina B, verde rodamina, rojo rodamina, y ROX, TRITC, tetrametilrodamina (TMR) , carboxitetrametilrodamina (TAMRA) , tetraclorofluoresceína (TET) , Rojo 6E, FluorX, BODIPY-FL, colorante SYBR Green I, y rojo Texas.
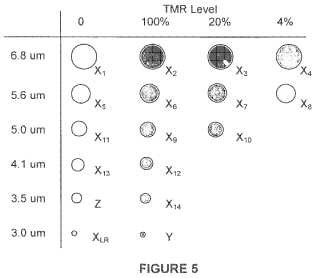
5. El conjunto de perlas de la reivindicación 4 en el que las perlas marcadas se marcan con TMR.
6. El conjunto de perlas de la reivindicación 1 que comprende de 2 a 17 familias de perlas.
7. El conjunto de perlas de una cualquiera de las reivindicaciones 1 a 6, en el que el conjunto de perlas comprende 17 familias de perlas.
8. El conjunto de perlas de la reivindicación 1, en el que las dos o más cepas del VPH se seleccionan de las cepas 6, 11, 16, 18, 31, 33, 35, 39, 45, 51, 52, 56, 58, 59, 66 y 68.
9. Un método para preparar un conjunto de perlas para su uso en la detección de una cepa de VPH y/o diferenciación entre dos o más cepas de VPH, comprendiendo el método seleccionar una pluralidad de familias o subconjuntos de perlas en base al tamaño, intensidad fluorescente, o ambos, que comprende proporcionar:
(a) al menos dos subconjuntos de perlas, en los que cada subconjunto de perlas se puede distinguir fisioquímicamente de cualquier otro subconjunto de perlas en base, al menos, al tamaño, o
(b) al menos un subconjunto de perlas que tienen al menos dos familias, en el que las perlas en un subconjunto de perlas son homogéneas en tamaño unas con respecto a otras, y se pueden distinguir fisioquímicamente en dos o más familias de perlas en base al nivel de marcado que contiene cada una, o
(c) una combinación de dichas familias de perlas y dichos subconjuntos de perlas;
y en los que:
las perlas dentro de cada familia de perlas de un subconjunto están cada una acopladas a una sonda de captura de ácido nucleico opcionalmente marcada que se puede unir a una región específica de cepa de VPH de un genoma de VPH u, opcionalmente, a al menos una de una secuencia de ácido nucleico de control, siempre que cualquier sonda de captura de ácido nucleico o secuencia de ácido nucleico de control esté marcada con la misma marca que otras sondas de captura marcadas o secuencia de ácido nucleico en el subconjunto de perlas, y que dentro de cualquier subconjunto de perlas individual, cada familia de perlas del mismo tiene una intensidad fluorescente diferente, en los que la sonda de captura de ácido nucleico se selecciona de SEQ ID NOs:46-63; y
en los que la región específica de cepa del VPH de un genoma del VPH es un amplicón generado amplificando ácido nucleico a partir de una muestra biológica que se ha obtenido de un sujeto humano, que comprende putativamente VPH, en los que el amplicón es distinto para dicho VPH o una cepa particular de dicho VPH, y en los que el amplicón se genera usando un par de cebadores de amplificación de oligonucleótidos de un cebador directo y un cebador inverso, en los que el cebador directo del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:1-8, y el cebador inverso del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:912, y en los que al menos uno de los cebadores directos e inversos está conjugado a una protuberancia en el extremo 5' terminal del cebador; y
en los que cada familia de perlas es específica para la detección de una cepa de VPH diferente de cualquier otra familia de perlas en el conjunto de perlas; y
mezclar las al menos dos familias o al menos dos subconjuntos de perlas juntas para producir un conjunto de perlas que permita la identificación de una cepa específica de VPH por medio del análisis del tamaño de perla, intensidad fluorescente, o discriminación de secuencias de la al menos una familia de perlas o subconjunto de perlas, usando citometría de flujo.
10. El método de la reivindicación 9, en el que la protuberancia del cebador directo se selecciona del grupo que consiste en:
CAATCAGC, ACAAT, GGAACAAT, GGAAC, CAGCTT, ATTACC, CTGTT, /5Phos/CAATCAGC, /5Phos/ACAAT, /5Phos/GGAACAAT, /5Phos/GGAAC, /5Phos/CAGCTT, 5Phos/ATTACC, y /5Phos/CTGTT.
11. Un método in vitro para detectar y/o diferenciar entre una o más particular cepas de VPH en una muestra biológica, comprendiendo dicho método:
(i) aislar el ácido nucleico de una muestra biológica se había obtenido a partir de un sujeto humano, que comprende putativamente VPH,
(ii) amplificar el ácido nucleico de dicha muestra usando cebadores que generan un amplicón que es distinto para dicho analito o una cepa particular de dicho analito,
(iii) amplificar opcionalmente una secuencia de ácido nucleico de control del ADN genómico del sujeto humano,
(iv) efectuar opcionalmente el marcado del/de los amplicón/amplicones enumerados en las etapas (ii) y/o (iii) ,
(v) hibridar el/los amplicón/amplicones marcados a un conjunto de perlas de reactivos en los que cada miembro del conjunto de perlas comprende una molécula de ácido nucleico que tiene complementariedad con una secuencia de nucleótidos del VPH o una cepa de VPH particular o una secuencia de nucleótidos de control, unida o asociada de otro modo con un sustrato que se puede distinguir fisioquímicamente, en el que la hibridación se produce en presencia de al menos una secuencia de oligonucleótidos de bloqueo, y
(vi) determinar a qué reactivo se ha unido un amplicón;
en el que la asociación de un amplicón con un reactivo particular es indicativa de una infección por VPH en el sujeto humano;
en el que los cebadores incluyen un cebador directo y un cebador inverso, en el que el cebador directo del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:1-8, y el cebador inverso del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:9-12, y en el que al menos uno de los cebadores directos e inversos está conjugado a una protuberancia en el extremo 5' terminal del cebador.
12. El método de la reivindicación 11, en el que la protuberancia del cebador directo se selecciona del grupo que consiste en:
CAATCAGC, ACAAT, GGAACAAT, GGAAC, CAGCTT, ATTACC, CTGTT, /5Phos/CAATCAGC, /5Phos/ACAAT, /5Phos/GGAACAAT, /5Phos/GGAAC, /5Phos/CAGCTT, 5Phos/ATTACC, y /5Phos/CTGTT.
13. Un método in vitro para detectar y/o diferenciar entre una o más particular cepas de VPH en una muestra biológica, comprendiendo dicho método:
(i) aislar el ácido nucleico de una muestra biológica se había obtenido a partir de un sujeto humano, que comprende putativamente VPH,
(ii) amplificar el ácido nucleico de dicha muestra usando cebadores que generan un amplicón que es distinto para dicho analito o una cepa particular de dicho analito,
(iii) amplificar opcionalmente una secuencia de ácido nucleico de control del ADN genómico del sujeto humano,
(iv) hibridar el/los amplicón/amplicones marcados a un conjunto de perlas de reactivos en los que cada miembro del conjunto de perlas comprende una molécula de ácido nucleico que tiene complementariedad con una secuencia de nucleótidos de VPH o una cepa de VPH particular o una secuencia de nucleótidos de control, unida o asociada de otro modo con un sustrato que se puede distinguir fisioquímicamente y en el que la hibridación se produce en presencia de al menos una secuencia de oligonucleótidos de señal y en presencia de al menos una secuencia de oligonucleótidos de bloqueo, y
(v) determinar a qué reactivo se ha unido un amplicón;
en el que la asociación de un amplicón con un reactivo particular es indicativa de una infección por VPH en el sujeto humano;
en el que los cebadores incluyen un cebador directo y un cebador inverso, en el que el cebador directo del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:1-8, y el cebador inverso del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:9-12, y en el que al menos uno de los cebadores directos e inversos está conjugado a una protuberancia en el extremo 5' terminal del cebador.
14. El método de la reivindicación 13, en el que la protuberancia del cebador directo se selecciona del grupo que consiste en:
CAATCAGC, ACAAT, GGAACAAT, GGAAC, CAGCTT, ATTACC, CTGTT, /5Phos/CAATCAGC, /5Phos/ACAAT, /5Phos/GGAACAAT, /5Phos/GGAAC, /5Phos/CAGCTT, 5Phos/ATTACC, y /5Phos/CTGTT.
15. Un método para determinar el riesgo de que un sujeto humano desarrolle una enfermedad asociada con una o más cepas de VPH comprendiendo dicho método las etapas del método de la reivindicación 11 o 13, en el que la asociación de un amplicón con un reactivo particular que comprende un polinucleótido que es complementario a una cepa de VPH asociada con una enfermedad particular, es indicativa de un incremento en el riesgo de dicha enfermedad en el sujeto.
16. Un método de una cualquiera de las reivindicaciones 11 a 15, en el que el conjunto de perlas de reactivos comprende una pluralidad de familias o una pluralidad de subconjuntos de perlas que tienen:
(a) al menos dos subconjuntos de perlas, en los que cada subconjunto de perlas se puede distinguir fisioquímicamente de cualquier otro subconjunto de perlas en base, al menos, al tamaño, o
(b) al menos un subconjunto de perlas que tienen al menos dos familias, en el que las perlas en un subconjunto de perlas son homogéneas en tamaño unas con respecto a otras, y se pueden distinguir fisioquímicamente en dos o más familias de perlas en base al nivel de marcado que contiene cada una, o
(c) una combinación de dichas familias de perlas y dichos subconjuntos de perlas;
y en los que:
las perlas dentro de cada familia de perlas de un subconjunto están cada una acopladas a una sonda de captura de ácido nucleico opcionalmente marcada que se puede unir a una región específica de cepa de VPH de un genoma de VPH u, opcionalmente, a al menos una de una secuencia de ácido nucleico de control, siempre que cualquier sonda de captura de ácido nucleico o secuencia de ácido nucleico de control esté marcada con la misma marca que otras sondas de captura marcadas o secuencia de ácido nucleico en el subconjunto de perlas, y que dentro de cualquier subconjunto de perlas individual, cada familia de perlas del mismo tiene una intensidad fluorescente diferente; y
cada familia de perlas es específica para la detección de una cepa de VPH diferente de cualquier otra familia de perlas en el conjunto de perlas; en los que dicho conjunto de perlas puede identificar una o más cepas de VPH específicas por medio de análisis del tamaño de perla, intensidad fluorescente, o discriminación de secuencias de la al menos una familia de perlas o subconjunto de perlas, usando citometría de flujo.
17. Un par de cebadores de amplificación de oligonucleótidos para dirigir VPH oncogénico, en el que el cebador directo del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:1-8, y el cebador inverso del par de cebadores se selecciona del grupo que consiste en SEQ ID NO:9-12, y en el que al menos uno de los cebadores directos e inversos está conjugado con una protuberancia en el extremo 5' terminal del cebador.
18. Un par de cebadores de la reivindicación 17, en el que la protuberancia del cebador directo se selecciona del grupo que consiste en:
CAATCAGC, ACAAT, GGAACAAT, GGAAC, CAGCTT, ATTACC, CTGTT, /5Phos/CAATCAGC, /5Phos/ACAAT, /5Phos/GGAACAAT, /5Phos/GGAAC, /5Phos/CAGCTT, 5Phos/ATTACC, y /5Phos/CTGTT.
19. Un par de cebadores de la reivindicación 17, en el que la protuberancia del cebador inverso se selecciona del grupo que consiste en SEQ ID NO:87-94.
20. Un par de cebadores de la reivindicación 17, que contienen una secuencia de protuberancia de ácido nucleico opcional, en el que el cebador directo del par de cebadores se selecciona del grupo que consiste en:
CAR YTR TTT GTT ACT GTK GTD GA (SEQ ID NO:3) , que tiene opcionalmente una ACAAT (SEQ ID NO:110) , /5Phos/ACAAT (SEQ ID NO:35) , GGAACAAT (SEQ ID NO:111) , o una /5Phos/GGAACAAT (SEQ ID NO:36) conjugada al extremo 5' terminal del cebador; y
GTD GAT ACY AC HCG YAG TAC HAA (SEQ ID NO:8) , que tiene opcionalmente una CTGTT (SEQ ID NO:112) o /5Phos/CTGTT (SEQ ID NO:41) conjugada con el extremo 5' terminal del cebador.
21. Un par de cebadores de la reivindicación 17, que contienen una secuencia de protuberancia de ácido nucleico opcional, en el que el cebador inverso del par de cebadores se selecciona del grupo que consiste en:
TGA AAA ATA AAY TGY AAA TCA TAT TCY TCM MCA TG (SEQ ID NO:9) que tiene opcionalmente una ACTCACTATAGG (SEQ ID NO:113) o /5AmMC6/ACTCACTATAGG (SEQ ID NO:42) conjugada al extremo 5' terminal del cebador,
TR CAY ARY TGA AAA ATA AAY TG (SEQ ID NO:11) , que tiene opcionalmente una TCTAATACGACTCACTATAGG (SEQ ID NO:114) o /5AmMC6/TCTAATACGACTCACTATAGG (SEQ ID NO:44) conjugada al extremo 5' terminal del cebador.
Patentes similares o relacionadas:
Dispositivos modulares para puntos de atención y usos de los mismos, del 29 de Julio de 2020, de Labrador Diagnostics LLC: Un sistema para la detección automatizada de un analito a partir de una muestra de fluido corporal, que comprende: un dispositivo de fluidos que comprende: […]
Anticuerpos del OPGL, del 15 de Julio de 2020, de AMGEN FREMONT INC.: Un anticuerpo, que comprende una cadena pesada y una cadena ligera, donde: a) la cadena pesada comprende: 1) una secuencia de aminoácidos recogida […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
ANTICUERPO MONOCLONAL O UNA PORCIÓN DE UNIÓN A ANTÍGENO DEL MISMO QUE SE UNE A LA PROTEÍNA L DEL VIRUS PARAINFLUENZA HUMANO (PIV); MÉTODO Y KIT PARA DETECTAR AL VIRUS PIV, del 2 de Julio de 2020, de PONTIFICIA UNIVERSIDAD CATÓLICA DE CHILE: La invención presenta la generación de anticuerpos monoclonales, o fragmentos de los mismos, que reconocen la proteína L del virus parainfluenza humano (PIV), donde dichos […]
ANTICUERPOS MONOCLONALES ESPECÍFICOS PARA EL ANTÍGENO PB2 DEL VIRUS DE LA INFLUENZA HUMANA (FLU), SECUENCIAS NUCLEOTÍDICAS; MÉTODO Y KIT DE DIAGNÓSTICO DE INFECCIÓN PRODUCIDA POR FLU, del 2 de Julio de 2020, de PONTIFICIA UNIVERSIDAD CATÓLICA DE CHILE: La invención presenta la generación de anticuerpos monoclonales, o fragmentos de los mismos, que reconocen la proteína PB2 del virus de la influenza humana (Flu), […]
Dispositivo para la detección de analitos, del 1 de Julio de 2020, de TECHLAB, INC.: Un dispositivo para detectar por lo menos una sustancia de interés en una muestra líquida, comprendiendo el dispositivo: (a) una unidad que […]
Un ensayo de la respuesta inmunológica mediada por células, del 17 de Junio de 2020, de Cellestis Limited: Un método para medir una actividad de respuesta inmunológica mediada por células, comprendiendo dicho método: (a) proporcionar una composición de incubación […]
Método de deducción de un valor de positividad de biomarcador en porcentaje para células seleccionadas presentes en un campo de visión, del 10 de Junio de 2020, de NOVARTIS AG: Método de deducción de un valor para el % de positividad de biomarcador (PBP) para todas las células u, opcionalmente, uno o más subconjuntos de las […]