Secuenciación y genotipado utilizando nucleótidos modificados en 2'' de forma reversible.
Método para la secuenciación de al menos una parte de un ácido nucleico molde,
que comprende:
(a) Incubar al menos un ácido nucleico molde con al menos una polimerasa, al menos un nucleótidoterminador reversible modificado en 2' que comprende un grupo bloqueador en la posición 2' de lafracción azúcar, en el que el grupo bloqueador en la posición 2' es un fosfato, y al menos un ácidonucleico cebador complementario a al menos una subsecuencia del ácido nucleico molde, demanera que la polimerasa extiende el ácido nucleico cebador produciendo al menos un producto deextensión del cebador que incorpora el nucleótido terminador reversible modificado en 2' en elextremo 3' del producto de extensión del cebador;
(b) Eliminar el grupo bloqueador de la posición 2' del nucleótido terminador reversible modificado en 2'en el extremo 3' del producto de extensión del cebador, y,
(c) Identificar el nucleótido terminador reversible modificado en 2' en el producto de extensión delcebador antes y/o durante (b), de manera que al menos una parte de la secuencia de bases delácido nucleico molde es determinable a partir del nucleótido terminador reversible modificado en 2'identificado, secuenciándose de este modo al menos una parte del ácido nucleico molde.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2006/048933.
Solicitante: F. HOFFMANN-LA ROCHE AG.
Nacionalidad solicitante: Suiza.
Dirección: GRENZACHERSTRASSE 124 4070 BASEL SUIZA.
Inventor/es: GELFAND, DAVID, H., Gupta,Amar P.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2388722_T3.pdf
Fragmento de la descripción:
Secuenciación y genotipado utilizando nucleótidos modificados en 2’ de forma reversible.
ANTECEDENTES DE LA INVENCIÓN
Las técnicas de análisis de secuencia del ADN han evolucionado para manejar de forma eficiente proyectos de secuenciación a gran escala. Sin embargo, existen limitaciones en las técnicas actualmente disponibles cuando se aplican a proyectos de secuenciación de alto rendimiento en los que es deseable limitar los costes manteniendo una velocidad suficiente. Por ejemplo, los métodos clásicos de secuenciación didesoxi de Sanger utilizan una etapa de separación de fragmentos de ADN sobre un gel. Esta etapa no se presta a la multiplexación a gran escala o al procesamiento en paralelo y además, presenta el problema de la compresión de bandas durante la electroforesis. Se han desarrollado otras técnicas para incrementar la velocidad y disminuir el coste de la secuenciación. Entre ellas se incluyen la secuenciación mediante hibridación (ver, por ejemplo, Bains y Smith, J. Theoret. Biol. 135:303-307, 1988; Drmanac y otros Genomics 4:114-128, 1989; Khrapko y otros FEBS Lett. 256:118-122, 1989; Southern, WO10977, 1989) ; secuenciación de firmas en paralelo basada en el enlazado y escisión (por ejemplo, Brenner y otros Proc. Natl. Acad. Sci. 97:1665-1670, 2000) ; secuenciación utilizando nucleótidos terminadores de cadena reversibles (ver, por ejemplo, Patentes EEUU nº 5.902.723 y 5.547.835; Canard y Arzumanov, Gene 11:1 (1994) ; y Dyatkina Arzumanov, Nucleic Acids Symp Ser 18:117 (1987) ) ; terminación de cadena reversible con ADN ligasa (ver, por ejemplo, Patente EEUU nº 5.403.708) ; secuenciación resuelta en el tiempo (ver, por ejemplo, Johnson y otros, Anal. Biochem. 136:192 (1984) ; y la pirosecuenciación (por ejemplo, Ronaghi y otros Anal. Biochem 242:84-89, 1996) .
La pirosecuenciación se basa en el concepto de la secuenciación mediante síntesis (por ejemplo, Patente EEUU nº 4.863.849) . La técnica puede aplicarse a proyectos de secuenciación en paralelo masivos. Por ejemplo, utilizando una plataforma automatizada, es posible realizar cientos de miles de reacciones de secuenciación de forma simultánea. La secuenciación mediante síntesis se diferencia de la estrategia de secuenciación didesoxi clásica en que en lugar de generar un gran número de secuencias de forma simultánea para caracterizarlas en una etapa posterior, se utiliza la monitorización en tiempo real de la incorporación de cada base dentro de una cadena en crecimiento. Aunque esta estrategia es lenta en el contexto de una reacción de secuenciación individual, puede utilizarse para generar una gran cantidad de información de secuencias en cada ciclo cuando se realizan entre cientos de miles y millones de reacciones en paralelo. A pesar de estas ventajas, existen aún limitaciones en la estrategia de pirosecuenciación. Por ejemplo, existen dificultades para determinar el número de nucleótidos incorporados en regiones homopoliméricas, debido a la respuesta de señal no lineal tras la incorporación de múltiples moléculas idénticas. Otras estrategias de secuenciación mediante síntesis sobre matrices en fase sólida que no utilizan terminadores reversibles presentan desventajas similares.
Recientemente, se ha descrito un método de secuenciación que utiliza terminadores reversibles utilizando análogos de nucleótido con modificaciones 3’-O-alilo (Ruparel y otros, Proc. Natl. Acad. Sci. 102:5932-5937, 2005) . En este método, el análogo de nucleótido contiene un grupo alilo que encapucha el grupo 3’-OH y un fluoróforo unido a la posición 5’ del uracilo a través de un enlazador fotoescindible. Este nucleótido es un substrato para la ADN polimerasa. Tras la incorporación dentro de la cadena de ADN y la fotoescisión del enlazador, se elimina el grupo alilo utilizando una reacción catalizada por Pd, y se reinicia la reacción de la polimerasa. De este modo, estos análogos actúan como terminadores reversibles en las reacciones de secuenciación mediante síntesis. Se describen otros terminadores reversibles en, por ejemplo, las Patentes EEUU nº 5.872.244; 6.232.465; 6.214.987; 5.808.045; 5.763.594, y 5.302.509; y la Publicación de solicitud de patente EEUU nº 20030215862. Los terminadores reversibles del tipo 3’-OH bloqueados presentan diversos inconvenientes incluyendo unas bajas eficiencias de incorporación y desbloqueo y las condiciones tediosas utilizadas para el desbloqueo. Un método altamente deseable para la secuenciación de alto rendimiento basada en terminadores reversibles debe demostrar unas eficiencias de incorporación, terminación de cadena y desbloqueo casi perfectas para minimizar los problemas y las señales de fondo generadas por las reacciones desfasadas.
Recientemente se han descrito nucleósidos 5’ trifosfato modificados en 2’ (por ejemplo 2’-fosfato) que pueden utilizarse como substratos para ciertas enzimas de polimerización de ácidos nucleicos para la incorporación de una sola base (ver, por ejemplo, las publicaciones de solicitud de patente EEUU nº 2005/00373898 y 2005/0037991) . La presente invención da a conocer nuevos métodos para la secuenciación y el genotipado que utilizan nucleótidos terminadores en 2’ en una reacción de secuenciación reversible.
BREVE RESUMEN DE LA INVENCIÓN
La presente invención da a conocer, por ejemplo, un método para determinar una secuencia de ácidos nucleicos utilizando un nucleótido modificado en 2’ que sirve como terminador reversible. En algunas realizaciones, la incorporación del nucleótido terminador reversible modificado en 2’ dentro de una cadena de ácidos nucleicos que está siendo alargada da lugar a una señal detectable. Como métodos ejemplares de detección de señales detectables se incluyen, por ejemplo, la detección del pirofosfato liberado por cascadas enzimáticas acopladas que da lugar a una señal luminosa, la detección de un marcador fluorescente en el nucleótido incorporado (escindible, para múltiples adiciones) , la detección de un nucleósido marcado en el fosfato terminal, la detección a través de una estrategia polifosfato / fosfatasa, entre otros métodos. El tratamiento del nucleótido incorporado con una actividad para eliminar la modificación o el grupo bloqueador da lugar a la extensión adicional de la cadena de ácidos nucleicos. La presente invención incluye de forma adicional, por ejemplo, kits que comprenden los componentes para determinar una secuencia, por ejemplo, para secuenciar, genotipar y similares utilizando el método de terminación reversible de la invención.
En un aspecto, la presente invención da a conocer un método para la secuenciación de al menos una parte de un ácido nucleico molde, comprendiendo dicho método:
a) Incubar al menos un ácido nucleico molde con al menos una polimerasa, al menos un nucleótido
terminador reversible modificado en 2’ que comprende un grupo bloqueador en la posición 2’ de la
fracción azúcar, en el que el grupo bloqueador en la posición 2’ es un fosfato (por ejemplo, un 2’
monofosfato-3’-hidroxil nucleótido, etc.) , y al menos un ácido nucleico cebador complementario de al
menos una subsecuencia del ácido nucleico molde, de manera que la polimerasa extiende el ácido
nucleico cebador produciendo al menos un producto de extensión del cebador que incorpora el
nucleótido terminador reversible modificado en 2’ en el extremo 3’ del producto de extensión del
cebador;
b) Eliminar el grupo bloqueador (por ejemplo un fosfato o similar) de la posición 2’ del nucleótido
terminador reversible modificado en 2’ en el extremo 3’ del producto de extensión del cebador, y,
c) Identificar el nucleótido terminador reversible modificado en 2’ en el producto de extensión del
cebador antes y/o durante
(b) de manera que al menos una parte de la secuencia de bases del ácido nucleico molde es determinable a partir del nucleótido terminador reversible modificado en 2’ identificado, secuenciándose de este modo al menos una parte del ácido nucleico molde. En algunas realizaciones, (b) comprende incubar el producto de extensión del cebador con una actividad que elimina el grupo bloqueador (por ejemplo, un fosfato, etc.) en la posición 2’. El método incluye, típicamente, repetir (a) - (c) una o más veces. En algunas realizaciones, la modificación... [Seguir leyendo]
Reivindicaciones:
1. Método para la secuenciación de al menos una parte de un ácido nucleico molde, que comprende:
(a) Incubar al menos un ácido nucleico molde con al menos una polimerasa, al menos un nucleótido terminador reversible modificado en 2’ que comprende un grupo bloqueador en la posición 2’ de la fracción azúcar, en el que el grupo bloqueador en la posición 2’ es un fosfato, y al menos un ácido nucleico cebador complementario a al menos una subsecuencia del ácido nucleico molde, de manera que la polimerasa extiende el ácido nucleico cebador produciendo al menos un producto de
extensión del cebador que incorpora el nucleótido terminador reversible modificado en 2’ en el extremo 3’ del producto de extensión del cebador;
(b) Eliminar el grupo bloqueador de la posición 2’ del nucleótido terminador reversible modificado en 2’ en el extremo 3’ del producto de extensión del cebador, y,
(c) Identificar el nucleótido terminador reversible modificado en 2’ en el producto de extensión del
cebador antes y/o durante (b) , de manera que al menos una parte de la secuencia de bases del ácido nucleico molde es determinable a partir del nucleótido terminador reversible modificado en 2’ identificado, secuenciándose de este modo al menos una parte del ácido nucleico molde.
2. Método, según la reivindicación 1, en el que (b) comprende incubar el producto de extensión del cebador con una actividad que elimina el fosfato de la posición 2’.
3. Método, según la reivindicación 2, en el que la actividad que elimina el fosfato en posición 2’ es una fosfatasa.
4. Método, según la reivindicación 2, en el que la actividad que elimina el fosfato en posición 2’ es una endonucleasa II.
5. Método, según la reivindicación 2, en el que la actividad que elimina el fosfato en posición 2’ es una 25 endonucleasa IV.
6. Método, según la reivindicación 2, en el que la actividad que elimina el fosfato en posición 2’ es un polinucleótido quinasa.
7. Método, según la reivindicación 2, en el que la actividad que elimina el fosfato en posición 2’ es una fosfodiesterasa o una combinación de una fosfodiesterasa y una fosfatasa.
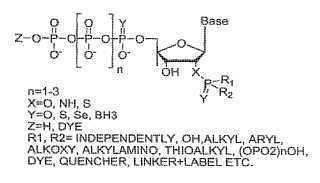
8. Método, según la reivindicación 1, en el que el nucleótido terminador reversible modificado en 2’ se selecciona de grupo formado por:
9. Método, según la reivindicación 1, en el que el nucleótido terminador reversible modificado en 2’ está marcado con al menos una fracción marcadora.
10. Método, según la reivindicación 9, en el que la fracción marcadora comprende un colorante fluorescente, una molécula luminiscente o un radioisótopo.
11. Método, según la reivindicación 10, en el que la fracción marcadora es un colorante fluorescente.
12. Método, según la reivindicación 9, en el que la fracción marcadora está unida a la base del nucleótido
terminador reversible modificado en 2’ a través de un enlazador escindible, y el método comprende además 40 una etapa de escisión del enlazador.
13. Método, según la reivindicación 9, en el que la fracción marcadora está unida a un residuo azúcar del nucleótido terminador reversible modificado en 2’.
14. Método, según la reivindicación 9, en el que la fracción marcadora está unida a un fosfato presente en la posición 2’ del nucleótido terminador reversible modificado en 2’.
15. Método, según la reivindicación 9, en el que el nucleótido terminador reversible modificado en 2’ está unido a dos fracciones marcadoras que comprenden un dador y un aceptor.
16. Método, según la reivindicación 15, en el que el dador y el aceptor son un par informador y desactivador.
17. Método, según la reivindicación 15, en el que las dos fracciones marcadoras son capaces de experimentar transferencia de energía por resonancia de fluorescencia.
18. Método, según la reivindicación 16, en el que la fracción desactivadora está unida a la base del nucleótido terminador reversible modificado en 2’ y la fracción informadora está unida a un fosfato, siempre y cuando el fosfato no sea el fosfato alfa del nucleótido terminador reversible modificado en 2’.
19. Método, según la reivindicación 18, en el que el fosfato es un fosfato gamma.
20. Método, según la reivindicación 18, en el que el fosfato es un fosfato presente en la posición 2’ del nucleótido terminador reversible modificado en 2’.
21. Método, según la reivindicación 15, en el que una fracción marcadora está unida a un fosfato presente en la posición 2’ del nucleótido terminador reversible modificado en 2’ y la segunda fracción marcadora está unida a un segundo fosfato.
22. Método, según la reivindicación 21, en el que el segundo fosfato es un fosfato gamma.
23. Método, según la reivindicación 1, en el que la mezcla de reacción comprende cuatro nucleótidos terminadores reversibles modificados en 2’ diferentes, poseyendo cada uno de ellos una base y un marcado diferentes con una fracción marcadora diferente.
24. Método, según la reivindicación 1, en el que la etapa de detección comprende detectar el pirofosfato generado con la incorporación del nucleótido terminador reversible modificado en 2’.
25. Método, según la reivindicación 1, en el que el nucleótido terminador reversible modificado en 2’ comprende un 2’-monofosfat-3’-hidroxil nucleótido.
26. Método, según la reivindicación 1, que comprende repetir las etapas (a) - (c) una o más veces.
27. Kit para la secuenciación mediante síntesis, que comprende:
- al menos un nucleótido terminador reversible modificado en 2’ que comprende un fosfato como
grupo bloqueador en la posición 2’ de la fracción azúcar, -una polimerasa, y -un reactivo para eliminar la modificación en la posición 2’ del nucleótido terminador reversible
modificado en 2’.
28. Kit, según la reivindicación 27, en el que el reactivo se selecciona del grupo formado por una fosfatasa, una exonucleasa III, una endonucleasa IV y un polinucleótido quinasa.
29. Kit, según la reivindicación 27, en el que el nucleótido terminador reversible modificado en 2’ está unido a un marcador.
30. Kit, según la reivindicación 27, que comprende además un nucleótido extensible.
31. Método de genotipado, que comprende:
a) Incubar un ácido nucleico molde en una mezcla de reacción que comprende un cebador, una polimerasa y un nucleótido terminador reversible modificado en 2’, comprendiendo dicho terminador reversible modificado una de las cuatro bases naturales o análogos de las mismas y comprendiendo además una modificación en la posición 2’ de la fracción azúcar, siendo dicha modificación en la posición 2’ un fosfato que termina la síntesis bajo condiciones en las que el cebador se hibrida al ácido nucleico molde y es extendido por la polimerasa presente en la reacción formando un producto extendido;
b) determinar si está presente una señal indicativa de la incorporación del nucleótido terminador reversible modificado en 2’ en el producto extendido;
c) si dicha señal no está presente, repetir las etapas a) y b) con un nucleótido terminador reversible modificado en 2’ diferente, comprendiendo dicho nucleótido terminador reversible modificado en 2’ diferente una base diferente de las cuatro bases naturales o análogos de las mismas,
d) incubar el producto extendido con una actividad que elimine la modificación presente en la posición 2’ del nucleótido terminador reversible,
e) repetir las etapas a) a c) un número deseado de veces para determinar la secuencia de una parte de dicho ácido nucleico molde, y
f) comparar la secuencia de la parte del ácido nucleico molde con secuencias conocidas de un sitio polimórfico.
32. Método para la secuenciación de al menos una parte de un ácido nucleico molde, que comprende:
a) Incubar el ácido nucleico molde en una mezcla de reacción que comprende un cebador, una polimerasa y un nucleótido terminador reversible modificado en 2’, que comprende un fosfato como grupo bloqueador en la posición 2’ de la fracción azúcar bajo condiciones en las que el cebador se hibrida al ácido nucleico molde y es extendido por la polimerasa presente en la reacción formando un producto extendido.
b) determinar si está presente una señal indicativa de la incorporación del nucleótido terminador reversible modificado en 2’ en el producto extendido;
c) incubar el producto extendido con una actividad que elimine la modificación presente en la posición 2’ del nucleótido terminador reversible,
d) repetir las etapas a) a d) un número deseado de veces para determinar la secuencia de al menos una parte de dicho ácido nucleico molde.
33. Método, según la reivindicación 32, en el que la etapa (a) se lleva a cabo utilizando una serie de nucleótidos terminadores reversibles modificados en 2’ diferentes en una única mezcla, comprendiendo cada nucleótido terminador reversible modificado en 2’ diferente una base diferente.
34. Método, según la reivindicación 32, en el que cada nucleótido terminador reversible modificado en 2’ se añade secuencialmente hasta que se detecta una señal indicativa de la incorporación de un nucleótido terminador reversible modificado en 2’.
35. Método, según la reivindicación 32, en el que cada nucleótido terminador reversible modificado en 2’ se añade simultáneamente y cada nucleótido terminador reversible modificado en 2’ está marcado con una fracción de marcado diferente.
36. Método de secuenciación de al menos una parte de un ácido nucleico molde, que comprende:
a) Incubar un ácido nucleico molde en una mezcla de reacción que comprende un cebador, una polimerasa, y un nucleótido terminador reversible modificado en 2’, comprendiendo dicho terminador reversible modificado una de las cuatro bases naturales o análogos de las mismas y comprendiendo además una modificación en la posición 2’ de la fracción azúcar, siendo dicha modificación en la posición 2’ un fosfato que termina la síntesis bajo condiciones en las que el cebador se hibrida al ácido nucleico molde y es extendido por la polimerasa presente en la reacción formando un producto extendido;
b) determinar si está presente una señal indicativa de la incorporación del nucleótido terminador reversible modificado en 2’ en el producto extendido;
c) si dicha señal no está presente, repetir las etapas a) y b) con un nucleótido terminador reversible modificado en 2’ diferente, comprendiendo dicho nucleótido terminador reversible modificado en 2’ diferente una base diferente de las cuatro bases naturales o análogos de las mismas,
d) incubar el producto extendido con una actividad que elimine la modificación presente en la posición 2’ del nucleótido terminador reversible,
e) repetir las etapas a) a d) un número deseado de veces para determinar la secuencia de al menos una parte de dicho ácido nucleico molde.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]