Métodos y composiciones para la inactivación de genes.
Proteína que comprende un dominio de unión al ADN con dedos de zinc modificada genéticamente,
en la que el dominio de unión al ADN comprende cuatro regiones de reconocimiento de dedo de zinc como sigue:
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2007/012588.
Solicitante: SANGAMO BIOSCIENCES, INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: POINT RICHMOND TECH. CENTER 501 CANAL BOULEVARD SUITE A100 RICHMOND, CA 94804 ESTADOS UNIDOS DE AMERICA.
Inventor/es: ANDO,Dale, HOLMES,Michael Christopher, LEE,Gary Ka Leong.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07K14/47 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › de mamíferos.
PDF original: ES-2378333_T3.pdf
Fragmento de la descripción:
Métodos y composiciones para la inactivación de genes.
ÍMBITO TÉCNICO
La presente descripción pertenece a los ámbitos de la ingeniería polipeptídica y genómica y de la recombinación homóloga.
ANTECEDENTES
Se han descrito varios métodos y composiciones para la escisión dirigida del ADN genómico. Tales escisiones dirigidas se pueden utilizar, por ejemplo, para inducir la mutagénesis dirigida, inducir deleciones dirigidas de las secuencias del ADN celular y facilitar la recombinación dirigida de un locus cromosómico predeterminado. Véanse, por ejemplo, las publicaciones de patente de los Estados Unidos 20030232410; 20050208489; 20050026157; 20050064474; 20060188987; y la publicación de patente internacional WO 07/014275.
CCR5, un receptor de quimiocinas con 7 dominios transmembranarios, es el correceptor principal para la entrada del VIH-1 en los linfocitos T CD4 (Samson et al. (1996) Nature 382: 722-725; Deng et al (1996) Nature 381: 661-666; Alkhatib (1996) Science 272: 1955-1958) . Desde el descubrimiento de que la deleción º32 en el gen del CCR5 confiere resistencia al VIH-1, se ha estudiado intensamente el CCR5 a modo de diana principal para el tratamiento del VIH. Aunque se ha demostrado que algunas moléculas pequeñas inducen la internalización del receptor o bloquean la interacción entre el CCR5 y el VIH (Fatkenheuer et al. (2005) Nat. Med. 11: 1170-1172) , estas estrategias con moléculas pequeñas han dado lugar al desarrollo de la resistencia a través de la selección de mutantes de escape que, interesantemente, continúan utilizando el CCR5 para la entrada del virus (Kuhmann et al. [2004] J. Virol. 78: 2790-2807) . De igual forma, dentro del organismo, las estrategias a base de ARNi y antisentidos sólo han bloqueado parcialmente la expresión del CCR5 hasta la fecha.
Así pues, se siguen necesitando composiciones que supriman completamente el gen de CCR5 para la penetración fenotípica y la resistencia a largo plazo a la infección del VIH.
Mani et al (Biochemical and Biophysical Research Communications, Academic Press Inc. Orlando, FL, US, vol. 335, nº 2, 23 de septiembre de 2005, páginas 447-457) , Urnov et al. (Nature, Nature Publishing Group, Londres, GB, vol. 435, nº 7042, 2 de junio de 2005, páginas 646-651) y Liu Qiang et al (Journal of Biological Chemistr y , vol. 277, n. 6, 8 de febrero de 2002, páginas 3850-3856) describen nucleasas con dedos de zinc que inducen roturas bicatenarias en los sitios elegidos de los genes deseados.
Durai et al. (Nucleic Acids Research, Oxford University Press, Surrey, GB, vol. 33, nº 18, 26 de octubre de 2005, páginas 5678-5990) y Kandavelou Karthikeyan et al. (Nature Biotechnology, Nature Publishing Group, Nueva York, NY, US, vol. 23, nº 6, junio de 2005, páginas 686-687) describen el concepto de utilizar dominios de unión al ADN de tipo dedo de zinc de síntesis artificial para crear nucleasas que se unan a determinadas secuencias diana y las escindan.
COMPENDIO
La presente invención se define mediante el conjunto de reivindicaciones adjuntas.
En la presente memoria se describen composiciones y métodos para la inactivación parcial o completa de un gen diana. También se describen métodos que utilizan estas composiciones (reactivos) , por ejemplo para inactivar un gen en una célula con propósitos terapéuticos y/o para producir líneas celulares en las cuales está inactivado un gen diana.
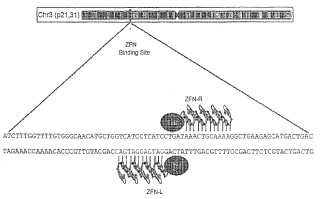
En un aspecto, en la presente memoria se dan a conocer nucleasas con dedos de zinc (ZFN, por su nombre en inglés) que tienen sitios diana en el gen del CCR-5 humano. En algunas realizaciones, la escisión dentro del gen de CCR-5 con estas nucleasas da lugar a la disrupción permanente (p. ej., mutación) del gen de CCR5. En algunas realizaciones, el o los dominios de dedo de zinc se modifican genéticamente para que se unan a un sitio diana cadena arriba de la mutación º32 del CCR5 que se produce de forma natural. Las proteínas con dedos de zinc pueden incluir 1, 2, 3, 4, 5, 6 o más dedos de zinc, teniendo cada dedo de zinc una hélice de reconocimiento que se une a un subsitio diana en el gen diana. En algunas realizaciones, el gen diana es CCR-5 y las proteínas con dedos de zinc comprenden 4 dedos (designados F1, F2, F3 y F4 y ordenados de F1 a F4 desde el extremo amino al extremo carboxilo) y comprenden la secuencia de aminoácidos de las regiones de reconocimiento mostradas en la tabla 1.
Así pues, en algunos aspectos, en la presente memoria se da a conocer una proteína que comprende un dominio de unión al ADN de tipo dedo de zinc modificado genéticamente, en donde el dominio de unión al ADN comprende cuatro regiones de reconocimiento de tipo dedo de zinc ordenadas de F1 a F4 desde el extremo amino al extremo carboxilo, y en donde F1, F3 y F4 comprenden las siguientes secuencias de aminoácidos: F1: DRSNLSR (SEQ ID nº 2) ; F3: RSDNLAR (SEQ ID nº 4) , y F4: TSGNLTR (SEQ ID nº 8) . En algunas realizaciones, F2 comprende la secuencia de aminoácidos ISSNLNS (SEQ ID nº 5) . Alternativamente, F2 comprende la secuencia de aminoácidos VSSNLTS (SEQ ID nº 6) .
Cualquiera de las proteínas descritas en la presente memoria pueden además comprender un dominio de escisión y/o un semidominio de escisión (p. ej., un semidominio de escisión de FokI de tipo silvestre o modificado genéticamente) . Así pues, en cualquiera de las ZFN descritas en la presente memoria, el dominio nucleasa puede comprender un dominio nucleasa o un semidominio nucleasa de tipo silvestre (p. ej., un semidominio de escisión de FokI) . En otras realizaciones, las ZFN comprenden dominios o semidominios nucleasa modificados genéticamente, por ejemplo, semidominios de escisión de FokI modificados genéticamente que forman heterodímeros estrictos. Véase, p. ej., la solicitud de patente provisional de los Estados Unidos nº 60/808 486, registrada el 25 de mayo de 2006.
En otro aspecto, la descripción da a conocer un polinucleótido que codifica cualquiera de las proteínas descritas en la presente memoria. Cualquiera de los polinucleótidos descritos en la presente memoria también puede comprender secuencias (secuencias donadoras o parches) para la inserción dirigida en el gen diana (p. ej., CCR-5) .
En otro aspecto más, se da a conocer un vector para introducir el gen que comprende cualquiera de los polinucleótidos descritos en la presente memoria. En determinadas realizaciones, el vector es un vector adenovírico (p. ej., un vector Ad5/35) . Así pues, en la presente memoria también se dan a conocer vectores de adenovirus (Ad) que comprenden un secuencia que codifica al menos una nucleasa con dedos de zinc (ZFN) y/o una secuencia donadora para la integración dirigida en un gen diana. En algunas realizaciones, el vector Ad es un vector de Ad quimérico, por ejemplo, un vector Ad5/35. En otras realizaciones, el gen diana es el gen del CCR-5 humano. Los vectores descritos en la presente memoria pueden comprender secuencias donadoras. En algunas realizaciones, un único vector comprende secuencias que codifican una o más ZFN y la o las secuencias donadoras. En otras realizaciones, la o las secuencias donadoras están contenidas en un primer vector y las secuencias que codifican la ZFN están presentes en un segundo vector.
Las secuencias de ZFN de los vectores (p. ej., vectores Ad) descritas en la presente memoria típicamente codificarán una fusión de una proteína con dedo de zinc (ZFP, por su nombre en inglés) y un dominio de escisión o semidominio de escisión (a saber, un dominio de nucleasa) . La porción de la proteína ZFN con dedos de zinc se modifica genéticamente para que se una a un sitio diana en el gen diana. Las proteínas con dedos de zinc pueden incluir 1, 2, 3, 4, 5, 6 o más dedos de zinc, teniendo cada dedo de zinc una hélice de reconocimiento que se une a un subsitio diana en el gen diana. En algunas realizaciones, el gen diana es CCR-5 y las proteínas con dedos de zinc comprenden 4 dedos (designados F1, F2, F3 y F4) y comprenden la secuencia de aminoácidos de las regiones de reconocimiento mostradas en la tabla 1.
En cualquiera de los polinucleótidos o proteínas descritos en la presente memoria, el dominio de escisión puede comprender al menos un dominio de escisión o al menos un semidominio de escisión. En algunas realizaciones, el dominio es escisión o el semidominio de escisión es un dominio de escisión de tipo silvestre (p. ej., un semidominio de escisión de tipo silvestre de FokI) . En otras realizaciones, el dominio de escisión o el semidominio... [Seguir leyendo]
Reivindicaciones:
1. Proteína que comprende un dominio de unión al ADN con dedos de zinc modificada genéticamente, en la que el dominio de unión al ADN comprende cuatro regiones de reconocimiento de dedo de zinc como sigue:
2. Proteína que comprende un dominio de unión al ADN con dedos de zinc modificada genéticamente, en la que el dominio de unión al ADN comprende cuatro regiones de reconocimiento de dedo de zinc como sigue:
3. Proteína que comprende un dominio de unión al ADN con dedos de zinc modificada genéticamente, en la que el dominio de unión al ADN comprende cuatro hélices de reconocimiento de dedo de zinc como sigue:
4. Proteína de acuerdo con una cualquiera de las reivindicaciones 1 a 3, que además comprende un dominio de escisión.
5. Proteína de acuerdo con la reivindicación 4, en la que el dominio de escisión es un semidominio de escisión.
6. Proteína de acuerdo con la reivindicación 5, en la que el semidominio de escisión es un semidominio de escisión 15 de FokI de tipo silvestre.
7. Proteína de acuerdo con la reivindicación 5, en la que el semidominio de escisión es un semidominio de escisión de FokI modificado genéticamente.
8. Polinucleótido que codifica la proteína de acuerdo con una cualquiera de las reivindicaciones 1 a 7.
9. Vector para transferencia génica, que comprende un polinucleótido de acuerdo con la reivindicación 8. 20 10. Vector para transferencia génica de acuerdo con la reivindicación 9, en donde el vector es un vector adenovírico.
11. Vector para transferencia génica de acuerdo con la reivindicación 10, en donde el vector adenovírico es un vector Ad5/35.
12. Célula aislada que comprende la proteína de acuerdo con una cualquiera de las reivindicaciones 1 a 7 o el polinucleótido de acuerdo con la reivindicación 8.
13. Célula de acuerdo con la reivindicación 12, en donde la célula se selecciona entre el grupo que consiste en una célula troncal hematopoyética, un linfocito T, un macrófago, una célula dendrítica y una célula presentadora de antígeno.
14. Célula de acuerdo con la reivindicación 13, en donde el linfocito T es un linfocito CD4+.
15. Célula de acuerdo con la reivindicación 13, en donde la célula se selecciona entre el grupo que consiste en una 30 célula K562, una célula HEK293, una PM-1, una célula THP-1 y una línea celular GHOST.
16. Método para inactivar el gen de CCR-5 en una célula humana aislada, comprendiendo el método la introducción de una proteína en la célula de acuerdo con una cualquiera de las reivindicaciones 4 a 7; un polinucleótido de acuerdo con la reivindicación 8 o un vector para transferencia génica de acuerdo con una cualquiera de las reivindicaciones 9 a 11.
17. Método de acuerdo con la reivindicación 16, en el que la célula aislada se selecciona entre el grupo que consiste en una célula troncal hematopoyética, un linfocito T, un macrófago, una célula dendrítica y una célula presentadora de antígeno.
18. Método de acuerdo con la reivindicación 17, en el que el linfocito T es un linfocito CD4+ aislado.
19. Método de acuerdo con la reivindicación 16, en el que la célula aislada se selecciona entre el grupo que consiste en una célula K562, una célula HEK293, una PM-1, una célula THP-1 y una línea celular GHOST.
20. Utilización de un primer ácido nucleico de acuerdo con la reivindicación 8 que codifica un primer polipéptido, en 5 donde el primer polipéptido comprende (i) un dominio de unión al ADN con dedos de zinc que está modificado genéticamente para que se una a un primer sitio diana en el gen de CCR5; y (ii) un dominio de escisión; para preparar un medicamento para tratar o prevenir la infección del VIH en un sujeto.
21. Utilización de acuerdo con la reivindicación 20, en la que el primer ácido nucleico de acuerdo con la reivindicación 7 codifica además un segundo polipéptido, en la que el segundo polipéptido comprende:
(i) un dominio de unión al ADN con dedos de zinc que está modificado genéticamente para que se una a un segundo sitio diana en el gen de CCR5; y
(ii) un dominio de escisión.
22. Utilización de acuerdo con la reivindicación 20, que comprende además un segundo ácido nucleico que contiene dos regiones de homología con el gen de CCR-5 que flanquean una secuencia que no es homóloga a la del gen de CCR-5.
CNT
Patentes similares o relacionadas:
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Polipéptidos de unión específica novedosos y usos de los mismos, del 15 de Julio de 2020, de Pieris Pharmaceuticals GmbH: Muteína de lipocalina lagrimal humana que tiene especificidad de unión para IL-17A, en la que la muteína se une a IL-17A con una KD de aproximadamente 1 nM o menos, en la que […]
Biomarcador de enfermedad autoinmunitaria, del 15 de Julio de 2020, de Tzartos, Socrates: Un método de diagnóstico o pronóstico de una enfermedad autoinmunitaria asociada con la formación de lesiones desmielinizadas del sistema nervioso central (SNC) […]
Composiciones útiles en el tratamiento de la deficiencia de ornitina transcarbamilasa (OTC), del 8 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Un vector vírico recombinante que comprende una secuencia de ácido nucleico que codifica la proteína ornitina transcarbamilasa humana (hOTC) y secuencias […]
Reactivos SIRP-alfa de alta afinidad, del 24 de Junio de 2020, de THE BOARD OF TRUSTEES OF THE LELAND STANFORD JUNIOR UNIVERSITY: Un polipéptido SIRPα de alta afinidad que comprende al menos una y no más de 15 modificaciones de aminoácidos dentro del dominio d1 de una secuencia SIRPα de tipo […]
Procedimientos y composiciones para el tratamiento de una afección genética, del 24 de Junio de 2020, de Sangamo Therapeutics, Inc: Una célula precursora de glóbulos rojos genomanipulada caracterizada por una modificación genómica dentro del exón 2 o el exón 4 de BCL11A o dentro de BCL11A-XL […]
Inmunoterapia WT1 para enfermedad angiogénica intraocular, del 17 de Junio de 2020, de INTERNATIONAL INSTITUTE OF CANCER IMMUNOLOGY, INC.: Una composición farmacéutica que comprende un péptido WT1 o péptido WT1 variante para su uso en el tratamiento y/o prevención de una enfermedad […]
Estructuras artificiales de poliepítopos para uso en inmunoterapia, del 17 de Junio de 2020, de Invectys: Un vector de expresión de ADN o una mezcla de vectores de expresión de ADN que codifica al menos dos epítopos de CD4 de la transcriptasa inversa de la telomerasa […]