Determinación de la variación en el número de copias, métodos y sistemas.
Un método para determinar el número de copias relativo de una secuencia polinucleotídica diana en el genoma de un sujeto,
que comprende:
preamplificar una secuencia polinucleotídica diana y una secuencia polinucleotídica de referencia en una muestra que contiene ADN genómico del sujeto;
ensayar la secuencia polinucleotídica diana y la secuencia polinucleotídica de referencia de la muestra preamplificada mediante una PCR digital;
determinar (a) el número de moléculas polinucleotídicas amplificadas que contienen la secuencia polinucleotídica diana, y (b) el número de moléculas polinucleotídicas amplificadas que contienen la secuencia polinucleotídica de referencia, y determinar la proporción de (a) a (b).
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2008/075636.
Solicitante: FLUIDIGM CORPORATION.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 7000 SHORELINE COURT, SUITE 100 SOUTH SAN FRANCISCO, CA 94080 ESTADOS UNIDOS DE AMERICA.
Inventor/es: RAMAKRISHNAN,Ramesh.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2380844_T3.pdf
Fragmento de la descripción:
Determinación de la variación en el numero de copias, metodos y sistemas Campo de la invención La invención se refiere a un metodo para determinar la variación en el numero de copias dentro de un genoma de 5 poblaciones pequefas o de individuos, y encuentra aplicación en biologfa y medicina.
Antecedentes de la invención Una "PCR digital" se refiere a un metodo en el que moleculas de acidos nucleicos individuales presentes en una muestra se distribuyen en muchos volumenes de reacción distintos (por ejemplo, camaras o partes alfcuotas) antes de la amplificación mediante PCR de una o mas secuencias diana. La concentración de moleculas individuales en la 10 muestra se ajusta para que, despues de la distribución, cada volumen de reacción contenga menos de una molecula polinucleotfdica discreta (o agregado de moleculas polinucleotfdicas enlazadas) , y la mayoría de las camaras contengan una o ninguna molecula. La amplificación de una secuencia diana produce una salida digital binaria en la que cada camara se identifica como que contiene o que no contiene el producto de la PCR indicativo de la presencia de la correspondiente secuencia diana. Un recuento de los volumenes de reacción que contienen niveles 15 detectables de productos finales de la PCR es una medición directa de la cantidad absoluta de acidos nucleicos. En una versión de la PCR digital, las moleculas polinucleotfdicas se distribuyen mediante su reparto en volumenes de reacción separados. Un metodo de reparto emplea la matriz digital BioMark™ 12.765 (Fluidigm Corp., South San Francisco, CA) . Este chip utiliza canales integrados y valvulas que reparten mezclas de muestras y reactivos en camaras de reacción con un volumen de 765 nanolitros. Las moleculas de ADN en cada mezcla se reparten 20 aleatoriamente en las 765 camaras de cada panel. El chip entonces se somete a termociclado y se forma una imagen en un sistema de PCR a tiempo real BioMark de Fluidigm, y las camaras positivas que originariamente contenfan una o mas moleculas pueden ser contadas por el programa informatico de analisis de matrices digitales. Para unos analisis sobre la PCR digital vease, por ejemplo, Vogelstein y Kinzler, 1999, Proc. Natl. Acad. Sci. USA, 96:9236-9241; McBride et al., publicación de la solicitud de patente de EEUU nO 20050252773, en especial el ejemplo 5.
Las variaciones en el numero de copias ("copy number variations", CNV) son las ganancias o las perdidas de regiones genómicas que varfan en tamafo de 500 bases o mas (a menudo entre cinco mil y cinco millones de bases) . Estudios del genoma completo han revelado la presencia de un gran numero de regiones CNV en seres humanos y una amplia gama de diversidad genetica entre la población general. Las CNV han sido el foco de muchos 30 estudios recientes debido a su papel en trastornos geneticos humanos. Vease, por ejemplo, Iafrate et al., 2004, Detection of large-scale variation in the human genome, Nat. Genet., 36:949-951; Sebat et al., 2004, Large-scale copy number polymorphism in the human genome, Science, 305:525-528; Redon et al., 2006, Global variation in copy number in the human genome, Nature, 444:444-454; Wong et al., 2007, A comprehensive analysis of common copy-number variations in the human genome, Am. J. Hum. Genet., 80:91-104; Ropers, 2007, New perspectives for the elucidation of genetic disorders, Am. J. Hum. Genet., 81:199-207; Lupski, 2007, Genomic rearrangements and sporadic disease, Nat. Genet., 39:S43-S47. La aneuploidía, tal como una trisomfa o la delección de un cromosoma completo, es un tipo limitante de variación en el numero de copias asociado con una diversidad de enfermedades humanas.
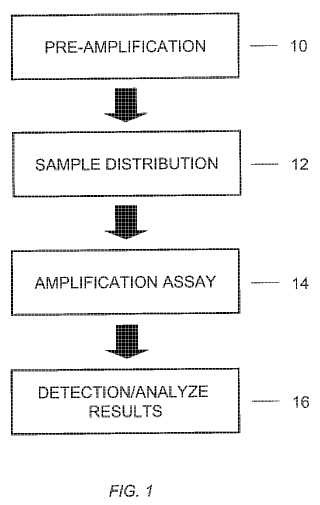
Breve sumario de la invención La invención proporciona un metodo para determinar el numero de copias relativo de una secuencia polinucleotfdica diana en el genoma de un sujeto, que comprende:
preamplificar una secuencia polinucleotfdica diana y una secuencia polinucleotfdica de referencia en una muestra que contiene ADN genómico del sujeto;
ensayar la secuencia polinucleotfdica diana y la secuencia polinucleotfdica de referencia de la muestra 45 preamplificada mediante una PCR digital;
determinar (a) el numero de moleculas polinucleotfdicas amplificadas que contienen la secuencia polinucleotfdica diana, y (b) el numero de moleculas polinucleotfdicas amplificadas que contienen la secuencia polinucleotfdica de referencia, y determinar la proporción de (a) a (b) . La invención se refiere a un metodo para determinar la variación en el numero de copias dentro de un genoma de poblaciones pequefas o de individuos.
50 El metodo proporciona la preamplificación del gen de interes en una muestra antes del analisis mediante PCR digital. La etapa de preamplificación permite la distribución de copias individuales del gen en muestras de reacción de PCR individuales para la detección de una manera que es mas representativa del numero de copias real que cuando se determina mediante PCR digital sin preamplificación.
Los metodos de la invención basados en la PCR digital tienen la capacidad de distinguir diferencias en menos de 2
55 veces en el numero de copias de genes con gran precisión. Por ejemplo, pueden diferenciar entre 1, 2, 3 y 4 copias de genes en diferentes muestras. Para asegurar que la diferencia aparente en el numero de copias de genes en
diferentes muestras es real, y no esta distorsionada por diferencias en las cantidades de muestra, los inventores emplean una expresión, el numero de copias relativo. El numero de copias relativo de un gen (por genoma humano) puede expresarse como la proporción del numero de copias de un gen diana al numero de copias de un gen de referencia de una sola copia en una muestra de ADN, que habitualmente es 1. Por ejemplo, el gen de la ARNasa P es un gen de una sola copia que codifica el resto de ARN de la enzima ARNasa P, y puede utilizarse como gen de referencia en un ensayo de numero de copias.
Un chip de matriz digital disponible en el mercado, tal como el que se ilustra en la figura 3, para realizar una PCR digital se ha utilizado para cuantificar el ADN en una muestra. El chip tiene 12 puertos de entrada de muestras para la introducción de una mezcla de muestra. Cada mezcla de muestra se reparte en 765 camaras de reacción en cada uno de los 12 paneles. Tal como se describe en la bibliograffa (vease, por ejemplo, McBride et al., publicación de la solicitud de patente de EEUU nO 20050252773) , la capacidad para cuantificar el ADN en muestras se basa en el hecho de que, cuando se introduce una cantidad apropiada de ADN, las moleculas de ADN individuales se distribuyen aleatoriamente en las camaras.
Utilizando dos ensayos para dos genes (por ejemplo, ARNasa P y otro gen de interes) con dos tintes fluorescentes en un chip es posible cuantificar simultaneamente la ARNasa P y el otro gen en la misma muestra de ADN y obtener una buena estimación de la proporción de estos dos genes y el numero de copias del gen de interes.
Sin embargo, cuando estan duplicados, multiples copias de un gen pueden estar estrechamente vinculadas en el mismo cromosoma y, por tanto, no pueden separarse entre sf, incluso en la matriz digital. Como resultado, multiples copias se comportarfan como una sola molecula, y el numero total de copias del gen serfa muy subestimado.
La presente invención se ocupa de este problema incluyendo una etapa de preamplificación en el proceso. La preamplificación es una reacción de PCR con cebadores para el gen de interes y para un gen de referencia (por ejemplo, el gen ARNasa P) . Generalmente se realiza durante un numero limitado de ciclos termicos (por ejemplo, 10 ciclos) ; suponiendo unas eficacias de PCR iguales, los numeros de copias de ambos genes aumentan proporcionalmente en la etapa de preamplificación. Utilizando este proceso, aunque multiples copias de un gen esten vinculadas entre sí sobre el genoma, despues de la preamplificación cada copia del gen de interes se amplificara por separado, y se repartira por separado en diferentes camaras de la matriz digital. Puesto que la moleculas recien generadas de ambos genes reflejan la proporción original y ya no estan vinculadas entre sf, un analisis con un chip digital puede cuantificar las moleculas de los dos genes y medir la proporción de los dos genes (y por tanto el numero de copias del gen de interes) de modo preciso.
Por tanto, la presente invención proporciona metodos para realizar analisis basados en genes. De modo mas específico, los metodos de la presente invención en general se refieren a la determinación de la variación en el numero de copias de un polinucleótido de interes... [Seguir leyendo]
Reivindicaciones:
1. Un metodo para determinar el numero de copias relativo de una secuencia polinucleotfdica diana en el genoma de un sujeto, que comprende:
preamplificar una secuencia polinucleotfdica diana y una secuencia polinucleotfdica de referencia en una muestra que contiene ADN genómico del sujeto;
ensayar la secuencia polinucleotfdica diana y la secuencia polinucleotfdica de referencia de la muestra preamplificada mediante una PCR digital;
determinar (a) el numero de moleculas polinucleotfdicas amplificadas que contienen la secuencia polinucleotfdica diana, y (b) el numero de moleculas polinucleotfdicas amplificadas que contienen la secuencia polinucleotfdica de referencia, y determinar la proporción de (a) a (b) .
2. El metodo de la reivindicación 1, en el que la muestra procede de un ser humano.
3. El metodo de la reivindicación 1, en el que: (i) la proporción de (a) a (b) es de aproximadamente 0, 5, y existe una deleción de la secuencia polinucleotfdica diana sobre un cromosoma; o (ii) la proporción de (a) a (b) es de aproximadamente 1, 5, y existe una duplicación de la secuencia polinucleotfdica diana sobre un cromosoma.
4. El metodo de la reivindicación 1, en el que:
la secuencia polinucleotfdica de referencia tiene un numero de copias genómicas predeterminado N;
la determinación del numero de moleculas polinucleotfdicas amplificadas que contienen la secuencia polinucleotfdica diana comprende determinar el numero de volumenes de reacción en los que esta presente la secuencia polinucleotfdica diana o una subsecuencia de esta (A) ;
la determinación del numero de moleculas polinucleotfdicas amplificadas que contienen la secuencia polinucleotfdica de referencia comprende determinar el numero de volumenes de reacción en los que esta presente la secuencia polinucleotfdica de referencia o una subsecuencia de esta (B) ; y el numero de copias relativo del polinucleótido diana en el genoma es aproximadamente igual a (A) / (B) x N.
5. El metodo de la reivindicación 4, en el que la realización de la preamplificación comprende combinar la muestra con una composición que comprende cebadores específicos para la secuencia polinucleotfdica diana y cebadores específicos para la secuencia polinucleotfdica de referencia, y realizar una reacción en cadena de polimerasa (PCR) para amplificar de forma separada el polinucleótido diana y el polinucleótido de referencia en una proporción sustancialmente identica.
6. El metodo de la reivindicación 4, en el que los volumenes de reacción se disponen en un dispositivo de microfluidos, y la preamplificación se realiza en un volumen de reacción separado del dispositivo de microfluidos.
7. El metodo de la reivindicación 4, en el que, antes de ensayar la secuencia polinucleotfdica diana y la secuencia polinucleotfdica de referencia de la muestra preamplificada mediante una PCR digital, toda o una porción de la muestra preamplificada se combina con reactivos seleccionados para la amplificación cuantitativa de la secuencia polinucleotfdica diana y la secuencia polinucleotfdica de referencia.
8. El metodo de la reivindicación 5, en el que los cebadores específicos para la secuencia polinucleotfdica de referencia utilizados en la etapa de preamplificación tambien se utilizan en el ensayo de la secuencia polinucleotfdica de referencia de la muestra preamplificada mediante PCR digital.
9. El metodo de la reivindicación 8, en el que los cebadores específicos para la secuencia polinucleotfdica diana utilizados en la etapa de preamplificación tambien se utilizan en el ensayo de la secuencia polinucleotfdica diana de la muestra preamplificada mediante PCR digital.
10. El metodo de la reivindicación 7, en el que los reactivos comprenden una primera sonda que se hibrida selectivamente con una secuencia polinucleotfdica diana, y una segunda sonda que se hibrida selectivamente con una secuencia polinucleotfdica de referencia bajo condiciones adecuadas para la amplificación de polinucleótidos.
11. El metodo de la reivindicación 10, en el que la primera y la segunda sonda comprenden diferentes marcadores detectables, y en el que la unión de la primera o de la segunda sonda o la degradación de la primera o de la segunda sonda tras una polimerización basada en la reacción en cadena de polimerasa (PCR) produce un cambio en la fluorescencia detectable del respectivo marcador detectable.
12. El metodo de la reivindicación 1, en el que la secuencia polinucleotfdica de referencia comprende una secuencia polinucleotfdica que codifica al menos parcialmente una enzima ARNasa P, beta-actina o GAPDH.
13. El metodo de la reivindicación 1 o de la reivindicación 2, en el que una proporción de secuencia polinucleotfdica diana a secuencia polinucleotfdica de referencia que se desvfe sustancialmente de un valor de 1 indica un numero de copias anómalo de la secuencia polinucleotfdica diana en el genoma del paciente.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]