GENES SENSIBLES A CRH EN EL CNS.
Un método para identificar un compuesto capaz de alterar la respuesta de exposición prolongada de CRH en una célula,
comprendiendo dicho método: a) poner en contacto dicha célula con CRH en presencia y ausencia de dicho compuesto; b) determinar la cantidad de al menos una proteína que modula la señalización de la hormona liberadora de corticotropina (CRH) en dicha célula; y c) comparar la cantidad de dicha proteína en presencia y ausencia de dicho compuesto; en el cual la proteína que modula la señalización de la hormona liberadora de corticotropina (CRH) es SEQ ID NO. 20
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2003/011792.
Solicitante: JANSSEN PHARMACEUTICA N.V..
Nacionalidad solicitante: Países Bajos.
Dirección: TURNHOUTSEWEG 30 2340 BEERSE PAISES BAJOS.
Inventor/es: PEETERS,Pieter Johan,Janssen Pharmaceutica N.V, GÖHLMANN,Hinrich W.H.,Janssen Pharmaceutica N.V, SWAGEMAKERS,Sigrid,Maria,Alice, KASS,Stefan Ulrich,Janssen Pharmaceutica N.V, STECKLER,Thomas H.W.Janssen Pharmaceutica N.V, FIERENS,Frederik,L.P.Janssen Pharmaceutica N.V.
Fecha de Publicación: .
Fecha Solicitud PCT: 23 de Octubre de 2003.
Clasificación Internacional de Patentes:
- C07K14/47 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › de mamíferos.
- C12Q1/68M6
Clasificación PCT:
- C07K14/575 C07K 14/00 […] › Hormonas.
- C12Q1/68 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
PDF original: ES-2357832_T3.pdf




Fragmento de la descripción:
La presente invención se refiere en general a la terapia y diagnosis de la depresión. En particular, esta invención se refiere a los polipéptidos así como a los polinucleótidos que codifican estos polipéptidos, en donde se ha demostrado que dichos polipéptidos juegan un papel fundamental en la mediación de la respuesta celular a la hormona liberadora de corticotropina. Estos polipéptidos y polinucleótidos son útiles en la diagnosis, el tratamiento y/o la prevención de la 5 depresión.
ANTECEDENTES DE LA INVENCIÓN
Análisis socioeconómicos recientes han encontrado que la depresión es una causa principal de discapacidad y un factor de riesgo importante para el desarrollo de otras enfermedades. Además, a escala mundial la depresión está infradiagnosticada e infratratada. Los fármacos antidepresivos actuales han demostrado ser eficaces, pero están 10 gravados con un comienzo de acción lento y efectos secundarios. Por encima de esto, no está todavía claro por qué modo farmacológico de acción ejercen los mismos sus efectos clínicos. La investigación impulsada por hipótesis, basada en la hipótesis de los receptores de corticosteroides de la depresión ha conducido al concepto nuevo que está enfocado en los receptores de neuropéptidos cerebrales, específicamente en el receptor de la hormona liberadora de corticotropina (CRH) como diana del fármaco. 15
La hormona liberadora de corticotropina (CRH), un polipéptido de 41 aminoácidos, juega un papel fundamental en la regulación del eje hipotalámico-pituitario-adrenal, mediando las respuestas endocrinas a diversas causas de estrés. Las neuronas hipotalámicas liberan CRH en el sistema portal hipofisario en respuesta al estrés, estimulando la secreción y biosíntesis de adrenocorticotropina pituitaria (ACTH), conduciendo a una producción incrementada de glucocorticoide adrenal (1). Varios estudios clínicos y preclínicos apuntan hacia un papel causal para las alteraciones en el sistema 20 CRH en el desarrollo de la depresión (2). Los primeros estudios con CRH en humanos demostraron que la respuesta de ACTH a CRH está debilitada en los pacientes deprimidos, reflejando una desensibilización de los receptores de CRH a la secreción de CRH hipotalámica incrementada continuamente (3; 4). En respaldo de la respuesta de ACTH debilitada como consecuencia de la liberación incrementada de CRH está el descubrimiento de niveles elevados de CRH en el fluido cerebroespinal de pacientes con depresión. Otros descubrimientos que refuerzan esta noción de la hipersecreción 25 de CRH en el estado deprimido son un número incrementado de neuronas secretoras del CRH y un número reducido de receptores de CRH en las víctimas de suicidios que sufrían depresión (5: 6).
Para CRH se han descrito dos receptores de afinidad alta, CRH-R1 y CRH-R2, los dos cuales existen en varias formas variantes de remodelación. La activación de estos receptores por CRH da como resultado la estimulación mediada por GS de adenilciclasa que conduce a niveles incrementados de cAMP intracelular. Esto activará por sí mismo la proteína-30 quinasa A dependiente de cAMP (PKA) y dará finalmente como resultado niveles citosólicos incrementados de cAMP y Ca2+. Los niveles incrementados de cAMP y Ca2+ conducen a la activación de varias otras quinasas adicionales tales como la quinasa II dependiente de Ca2+/calmodulina (CAMK II) y las quinasas activadas por mitógenos p42/p44 (MAPK). Como resultado, la proteína de fijación del elemento de respuesta a Ca2+/cAMP (CREB) se fosforila y esto regulará a su vez la transcripción de genes que contienen elementos de respuesta a cAMP (CRE) en su región 35 promotora. Ejemplos de tales genes que se demuestra están implicados en la modulación de la señalización por CRH incluyen c-fos, el gen del factor inhibidor de la migración de los macrófagos Mif, los receptores nucleares huérfanos Nurr77 y Nurr1.
Con objeto de desarrollar un modelo animal para la activación crónica pituitario-adrenal, se han generado ratones que sobreexpresan CRF (CRF-OE) (Stenzel-Poore et al., (1992) Endocrinology 130:3378-3386). Estos ratones transgénicos 40 presentan un fenotipo Cushingoide debido a la superproducción de ACTH y corticosterona a lo largo de su vida como resultado de la sobreexpresión global de CRF en estos animales. En armonía con el papel del CRF cerebral en la mediación de las respuestas endocrina, autonómica y conductual al estrés, los ratones CRF-OE exhiben un estado espontáneo de emotividad acrecentada, son hiper-reactivos a los estresantes y la administración central de un antagonista de los receptores de CRF invierte estos comportamientos de tipo ansiogénico (Stenzel-Poore et al., (1994) 45 J. Neurosci. 14:2579-2584). Estos ratones CRF-OE permiten la investigación de los efectos a largo plazo de CRF sobre el sistema nervioso central y se han utilizado como modelo genético de comportamiento relacionado con la ansiedad y el estrés. A pesar del hecho de que los caminos aguas abajo para los receptores activados por CRH fueron estudiados extensivamente y condujeron a la identificación de varios genes implicados en la cascada de señalización, queda por explorar un área importante. Por ello, ha sido objeto de la presente invención explorar la respuesta transcripcional a la 50 estimulación de CRH al nivel de amplitud del genoma a fin de identificar genes ulteriores implicados en la red génica activada por los receptores de la hormona liberadora de corticotropina. Los polipéptidos así identificados y los polinucleótidos que codifican dichos polipéptidos proporcionan nuevas probabilidades para el desarrollo de fármacos como dianas de fármaco por técnicas de selección, o son útiles en la diagnosis, prevención y/o tratamiento de la depresión. 55
SUMARIO DE LA INVENCIÓN
La presente solicitud describe cierto número de genes que no se habían asociado hasta ahora con señalización de CRH y son útiles por consiguiente en métodos para identificación de compuestos, que modulan la respuesta de señalización de CHR en una célula o en métodos diagnósticos para identificar la depresión inducida por CRH en un individuo.
En una realización, el método para identificar un compuesto capaz de alterar la respuesta de señalización de CRH en una célula, en particular la línea de células de adenoma derivadas del corticotropo de pituitaria de murino AtT-20 comprende poner en contacto dicha célula con CRH en presencia y ausencia de dicho compuesto y determinar el nivel de expresión de un polinucleótido que comprende una secuencia de ácido nucleico seleccionada del grupo constituido por SEQ ID NO.1, SEQ ID NO.2, SEQ ID NO.3, SEQ ID NO.4, SEQ ID NO.5, SEQ ID NO.6, SEQ ID NO.7, SEQ ID 5 NO.8, SEQ ID NO.9, SEQ ID 11, SEQ ID NO.13, SEQ ID NO.15, SEQ ID NO.17, SEQ ID NO.19, SEQ ID NO.21, SEQ ID NO.23, SEQ ID NO.25, SEQ ID NO.26, SEQ ID NO.27, SEQ ID NO.28, SEQ ID NO.29, SEQ ID NO.30, SEQ ID NO.31, SEQ ID NO.32, SEQ ID NO.34, SEQ ID NO.36, SEQ ID NO.38 y SEQ ID NO.40. En este método de selección, los niveles de expresión se evalúan típicamente utilizando una sonda oligonucleotídica que se fija a los polinucleótidos arriba mencionados, utilizando preferiblemente métodos de tecnología de redes. De acuerdo con ello, en una realización 10 particular se describe un método para identificar compuestos que modulan la respuesta de señalización de CRH en una célula, comprendiendo dicho método poner en contacto dicha célula con CRH en presencia y ausencia de dicho compuesto; y determinar el nivel de expresión de los polinucleótidos que tienen las secuencias de ácido nucleico SEQ ID NO.1, SEQ ID NO.2, SEQ ID NO.3, SEQ ID NO.4, SEQ ID NO.5, SEQ ID NO.6, SEQ ID NO.7, SEQ ID NO.8, SEQ ID NO.9, SEQ ID 11, SEQ ID NO.13, SEQ ID NO.15, SEQ ID NO.17, SEQ ID NO.19, SEQ ID NO.21, SEQ ID NO.23, SEQ 15 ID NO.25, SEQ ID NO.26, SEQ ID NO.27, SEQ ID NO.28, SEQ ID NO.29, SEQ ID NO.30, SEQ ID NO.31, SEQ ID NO.32, SEQ ID NO.34, SEQ ID NO.36, SEQ ID NO.38 y SEQ ID NO.40, en donde un cambio en el perfil de expresión de estas secuencias es indicativo de un compuesto capaz de alterar la respuesta de señalización de CRH en dicha célula.
BREVE DESCRIPCIÓN DE LOS DIBUJOS 20
Tabla 1: Tabla de genes que demuestran ser mediadores importantes de los cambios inducidos por CRH en el CNS.
Tabla 2: Secuencias de oligonucleótidos utilizadas para RT-PCR
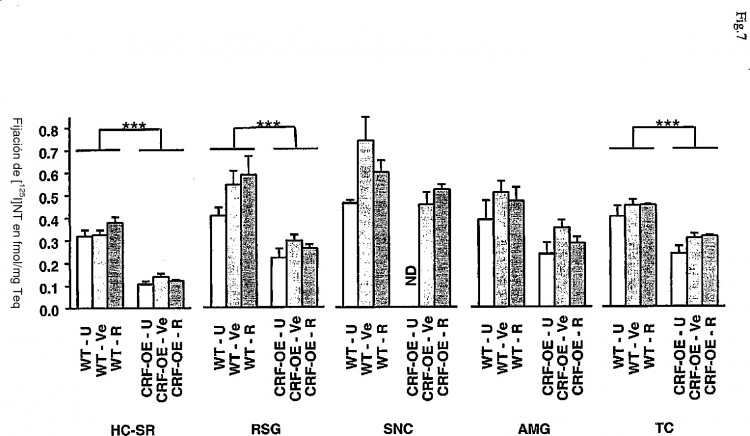
Figura 1: Niveles de expresión de CRH-R1 en diferentes tejidos del ratón. Se aplicó RT-PCR cuantitativa sobre tejidos derivados de 3 animales diferentes para las regiones cerebrales.
Figura 2: Análisis en mapa espectral... [Seguir leyendo]
Reivindicaciones:
1. Un método para identificar un compuesto capaz de alterar la respuesta de exposición prolongada de CRH en una célula, comprendiendo dicho método:
a) poner en contacto dicha célula con CRH en presencia y ausencia de dicho compuesto;
b) determinar la cantidad de al menos una proteína que modula la señalización de la hormona liberadora de 5 corticotropina (CRH) en dicha célula; y
c) comparar la cantidad de dicha proteína en presencia y ausencia de dicho compuesto;
en el cual la proteína que modula la señalización de la hormona liberadora de corticotropina (CRH) es SEQ ID NO. 20.
2. Un método de acuerdo con la reivindicación 1, en el cual la célula es una célula eucariota tal como la 10 línea de células de adenoma derivada del corticotropo de pituitaria murina AtT-20.
3. Un método de acuerdo con las reivindicaciones 1 ó 2 en el cual la cantidad de proteína que modula la señalización de CRH está siendo determinada utilizando un anticuerpo que se fija a un polipéptido que comprende una secuencia de aminoácidos SEQ ID NO. 20.
4. Un método de acuerdo con las reivindicaciones 1 ó 2 en el cual la cantidad de proteína que modula la 15 señalización de CRH está siendo determinada por evaluación del nivel de transcripción génica de un gen que codifica una secuencia de aminoácidos SEQ ID NO. 20.
5. Un método de acuerdo con la reivindicación 4, en el cual el nivel de transcripción génica está siendo evaluado utilizando una sonda que se fija a un polinucleótido que codifica la secuencia de aminoácidos SEQ ID NO. 20. 20
6. Un método de acuerdo con las reivindicaciones 4 ó 5 en el cual el nivel de expresión génica se analiza utilizando tecnología de microrredes.
7. Un método de acuerdo con la reivindicación 6, en el cual el nivel de transcripción génica se analiza utilizando una red de sondas oligonucleotídicas que se fijan a los polinucleótidos que codifican el grupo de polipéptidos que tienen la secuencia de aminoácidos SEQ ID NO. 20. 25
8. Un método de acuerdo con la reivindicación 6, en el cual el nivel de transcripción génica se analiza utilizando una red de sondas oligonucleotídicas que se fijan a los polinucleótidos que tienen la secuencia de ácido nucleico SEQ ID NO. 19.
9. Un método para identificación de un compuesto capaz de alterar la respuesta de exposición prolongada a CRH en una célula, comprendiendo dicho método: 30
a) poner en contacto una célula que expresa al menos una proteína que comprende la secuencia de aminoácidos SEQ ID NO. 20, con dicho compuesto de test; y
b) comparar la respuesta de CRH de dicha célula en presencia y ausencia de dicho compuesto.
10. Un método de acuerdo con la reivindicación 9, en el cual la célula es una célula hospedadora capaz de expresar al menos una proteína que tiene la secuencia de aminoácidos de SEQ ID NO. 20. 35
11. Un método de acuerdo con la reivindicación 10, en el cual la célula hospedadora se transfecta con al menos un vector que comprende una secuencia reguladora.
12. Un método de acuerdo con la reivindicación 10, en el cual la célula hospedadora se transfecta con al menos un vector que comprende una secuencia de polinucleótidos que codifica la secuencia de aminoácidos SEQ ID NO. 20. 40
Patentes similares o relacionadas:
Polipéptidos de unión específica novedosos y usos de los mismos, del 15 de Julio de 2020, de Pieris Pharmaceuticals GmbH: Muteína de lipocalina lagrimal humana que tiene especificidad de unión para IL-17A, en la que la muteína se une a IL-17A con una KD de aproximadamente 1 nM o menos, en la que […]
Biomarcador de enfermedad autoinmunitaria, del 15 de Julio de 2020, de Tzartos, Socrates: Un método de diagnóstico o pronóstico de una enfermedad autoinmunitaria asociada con la formación de lesiones desmielinizadas del sistema nervioso central (SNC) […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Composiciones útiles en el tratamiento de la deficiencia de ornitina transcarbamilasa (OTC), del 8 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Un vector vírico recombinante que comprende una secuencia de ácido nucleico que codifica la proteína ornitina transcarbamilasa humana (hOTC) y secuencias […]
Procedimientos y composiciones para el tratamiento de una afección genética, del 24 de Junio de 2020, de Sangamo Therapeutics, Inc: Una célula precursora de glóbulos rojos genomanipulada caracterizada por una modificación genómica dentro del exón 2 o el exón 4 de BCL11A o dentro de BCL11A-XL […]
Reactivos SIRP-alfa de alta afinidad, del 24 de Junio de 2020, de THE BOARD OF TRUSTEES OF THE LELAND STANFORD JUNIOR UNIVERSITY: Un polipéptido SIRPα de alta afinidad que comprende al menos una y no más de 15 modificaciones de aminoácidos dentro del dominio d1 de una secuencia SIRPα de tipo […]
Inmunoterapia WT1 para enfermedad angiogénica intraocular, del 17 de Junio de 2020, de INTERNATIONAL INSTITUTE OF CANCER IMMUNOLOGY, INC.: Una composición farmacéutica que comprende un péptido WT1 o péptido WT1 variante para su uso en el tratamiento y/o prevención de una enfermedad […]
Estructuras artificiales de poliepítopos para uso en inmunoterapia, del 17 de Junio de 2020, de Invectys: Un vector de expresión de ADN o una mezcla de vectores de expresión de ADN que codifica al menos dos epítopos de CD4 de la transcriptasa inversa de la telomerasa […]