Proteínas de superhélice antiparalela de una sola cadena.
Proteína de una sola cadena aislada que consiste en la fórmula HRS1-L1-HRS2-L2-HRS3,
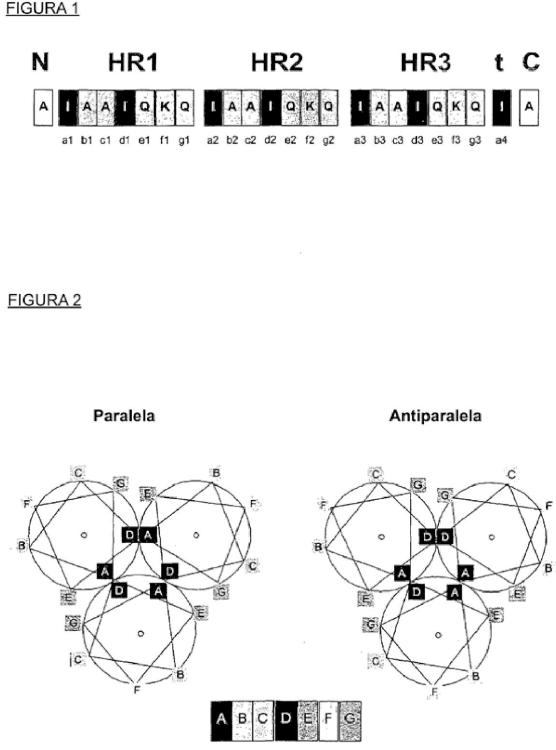
en la que HRS1, L1, HRS2, L2 y HRS3 representan fragmentos de secuencia de aminoácidos que están interconectados covalentemente, plegándose espontáneamente dicha proteína en disolución acuosa por medio de los fragmentos HRS1, HRS2 y HRS3 que forman una estructura de superhélice alfa-helicoidal, antiparalela, de triple hebra, y en la que
cada uno de HRS1, HRS2 y HRS3 es independientemente una secuencia de repetición en héptada que se caracteriza por un patrón de 7 residuos repetido n veces de tipos de aminoácidos, representado como (a-bc- d-e-f-g-)n o (d-e-f-g-a-b-c-)n y una repetición en héptada parcial C-terminal que termina con un residuo de núcleo de héptada ubicado en una posición a o d, en la que los elementos de patrón "a" a "g" indican posiciones de héptada convencionales en las que se ubican dichos tipos de aminoácidos y n es un número igual a o mayor de 2, y
al menos el 50%, el 70%, el 90% o el 100% de las posiciones de héptada convencionales "a" y "d" están ocupadas por aminoácidos seleccionados del grupo que consiste en valina, isoleucina, leucina, metionina, fenilalanina, tirosina, triptófano, histidina, glutamina, treonina, serina, alanina o derivados de los mismos, en la que al menos el 50%, el 70%, el 90% o en la que el 100% de las posiciones de héptada convencionales "b", "c", "e", "f" y "g" están ocupadas por aminoácidos seleccionados del grupo que consiste en glicina, alanina, cisteína, serina, treonina, histidina, asparagina, ácido aspártico, glutamina, ácido glutámico, lisina, arginina o derivados de los mismos, permitiendo la distribución resultante de los tipos de aminoácidos la identificación de dichas secuencias de repetición en héptada, y
cada uno de L1 y L2 es independientemente un ligador que consiste en de 1 a 30 residuos de aminoácido, incluyendo este ligador cualquier residuo de aminoácido que no puede asignarse de manera inequívoca a una secuencia de repetición en héptada, y
L1 y L2 tienen una composición de aminoácidos que comprende al menos el 50% de aminoácidos seleccionados del grupo que consiste en glicina, alanina, serina, treonina, prolina o derivados de los mismos.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2009/066640.
Solicitante: Complix N.V.
Nacionalidad solicitante: Bélgica.
Dirección: Agoralaan Building A-bis 3590 Diepenbeek BELGICA.
Inventor/es: LASTERS, IGNACE, DESMET,JOHAN, LOVERIX,STEFAN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07K1/00 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › Procedimientos generales de preparación de péptidos.
- C07K14/00 C07K […] › Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados.
- C07K7/08 C07K […] › C07K 7/00 Péptidos con 5 a 20 aminoácidos en una secuencia totalmente determinada; Sus derivados. › con 12 a 20 aminoácidos.
PDF original: ES-2547121_T3.pdf




Fragmento de la descripción:
Proteínas de superhélice antiparalela de una sola cadena Campo de la invención
La presente invención se encuentra en el campo de la biología molecular y se refiere a proteínas de una sola cadena, termodinámicamente estables, que consisten esencialmente en una estructura de armazón de superhélice alfa-helicoidal, antiparalela, de triple hebra en disoluciones acuosas. Tales moléculas son muy estables y tolerantes a sustituciones de aminoácidos. Por consiguiente, cumplen los requisitos básicos de un armazón basado en proteínas. Este armazón que muestra capacidades terapéuticas, de diagnóstico y/o de purificación, puede usarse en el campo del descubrimiento de fármacos, la investigación analítica, la tecnología de purificación y como modelo para mejorar el diseño de nuevas estructuras de armazón proteínicas (similares a proteínas). Las moléculas de armazón basadas en proteínas a menudo se consideran la clase de compuestos de "nueva generación" para el reconocimiento molecular, que compiten cada vez más con compuestos basados en inmunoglobulinas. Por consiguiente, los compuestos de la presente invención ofrecen un enfoque alternativo a las inmunoglobulinas, y un tipo adicional de armazón basado en proteínas (proteínico).
Antecedentes de la invención
Los complejos de superhélice alfa-helicoidal de triple hebra (3 hebras) (estructuras de superhélice, superhélices) se forman en disolución por la asociación (reunión) de moléculas peptídicas individuales (separadas, monoméricas, libres) dando lugar a trímeros (complejos de 3 moléculas). Los péptidos individuales comprenden normalmente una o más repeticiones en héptada (unidades de héptada, héptadas) que proporcionan la fuerza motriz termodinámica para tal asociación.
Un problema práctico importante encontrado con la formación de complejos triméricos es el hecho de que tales reacciones son extremadamente dependientes de la concentración. Por tanto, a menos que la fuerza motriz electrodinámica sea extremadamente fuerte (es decir, sólo si las héptadas forman interacciones extremadamente estrechas), tienen que aplicarse concentraciones relativamente altas con el fin de que se forme el complejo trimérico. Concentraciones altas pueden tener múltiples efectos adversos cuando se aplican a (se administran como) compuestos farmacéuticos. A diferencia de los complejos triméricos, la formación de (el plegamiento de) estructuras de superhélice de una sola cadena de la presente invención no es dependiente de su concentración en disolución. La presente invención por tanto pretende proporcionar una solución al problema de la dependencia de la concentración.
Un segundo problema relacionado con el uso de complejos oligoméricos peptídicos (multiméricos) es que los péptidos constituyentes son difíciles de producir (sintetizar) a través de métodos recombinantes (es decir, usando técnicas biológicas moleculares). Esto contrasta con las proteínas de una sola cadena plegadas de manera estable, que son adecuadas idealmente para la síntesis recombinante. Por tanto, la presente invención proporciona una solución a los problemas técnicos relacionados con la síntesis de armazones de superhélice triméricos en forma peptídica.
En tercer lugar, la presente invención tiene como objetivo proporcionar una solución práctica al problema de crear estructuras de superhélice heterotriméricas. La naturaleza oligomérica de las superhélices peptídicas se define en general por el número de péptidos asociados (por ejemplo, 2, 3, 4, para complejos diméricos, triméricos, tetraméricos, respectivamente), su orientación mutua (por ejemplo, paralela o antiparalela) y su similitud química (es decir, su secuencia de aminoácidos con derivatización opcional; por ejemplo, las superhélices homotriméricas se forman mediante tres péptidos idénticos, las superhélices heterotriméricas comprenden al menos un péptido derivatizado o de secuencia diferente). Las superhélices oligoméricas pueden obtenerse en disolución acuosa mezclando péptidos no idénticos. Entonces, tras un tiempo de incubación suficientemente largo, se formará una distribución de superhélices homo y heteroméricas, dependiendo principalmente de la capacidad termodinámica de estas últimas (estabilidad, energía libre, calidad de la asociación). En vista de las interacciones atómicas complicadas que constituyen la base de la capacidad termodinámica y, de ese modo, de las preferencias (distribuciones) oligoméricas, la creación de tipos específicos, deseados de superhélices heteroméricas es técnicamente difícil de controlar. A este respecto, la presente invención proporciona una solución práctica a un problema técnico: puesto que los fragmentos peptídicos que forman la superhélice están unidos covalentemente entre sí dando lugar a una sola cadena (a través de fragmentos de ligador elegidos de manera adecuada), su propensión a formar estructuras de superhélice de naturaleza predefinida (deseada) está considerablemente potenciada en comparación con superhélices equivalentes que consisten en conjuntos de péptidos libres. Por consiguiente, la construcción de superhélices heterodiméricas específicas (por ejemplo, heterotriméricas) se facilita considerablemente. Además, el formato de superhélice de una sola cadena también ofrece la ventaja de evitar (o reducir considerablemente el riesgo de) la formación de tipos de asociación no deseados (por ejemplo, no funcionales). En general, el formato de una sola cadena, que se aplica a todas las realizaciones de la presente invención, proporciona una solución práctica para controlar y conservar la especificidad de plegamiento de una superhélice trimérica en la que los fragmentos peptídicos que forman la superhélice son (opcionalmente) diferentes
en la secuencia de aminoácidos.
Todas las realizaciones de la presente invención se refieren a estructuras de superhélice alfa-helicoidal de "una sola cadena" aunque de "triple hebra". Por motivos de claridad, se explica en el presente documento (y se comenta adicionalmente más adelante en detalle) que la propiedad de "una sola cadena" se refiere a las moléculas completas de la presente invención, mientras que la propiedad de "triple hebra" se refiere a la parte de superhélice alfa- helicoidal dentro de estas moléculas. Siempre que se use la descripción "superhélice de una sola cadena", esto debe interpretarse como una asociación estrecha entre (tres) fragmentos peptídicos que forman superhélice que están interconectados covalentemente mediante (dos) fragmentos de ligador estructuralmente flexibles; dichos fragmentos peptídicos y de ligador forman juntos una molécula de proteína que consiste en una sola cadena de aminoácidos, contigua. Las proteínas de superhélice de una sola cadena de la presente invención también son monómeros (moléculas de proteína monoméricas en disolución), lo que no debe confundirse con la naturaleza trimérica de la estructura de superhélice que está contenida dentro de cada una de tales proteínas. La proteína de una sola cadena aislada se define en las reivindicaciones.
La inmensa mayoría de las estructuras de superhélice de triple hebra en el Protein Data Bank (Banco de Datos de Proteínas) (a continuación en el presente documento denominado PDB) son superhélices paralelas, es decir del tipo de "péptidos alfa-helicoidales paralelos". Esto significa que las superhélices existen como complejos (asociaciones no covalentes) de tres péptidos alfa-helicoidales por estructura y en las que las hélices están orientadas en una configuración (orientación) paralela. Muy raramente, una de las tres alfa-hélices se orienta antiparalela a las otras dos (que entonces son paralelas entre sí). Una disposición antiparalela de este tipo es excepcional en las proteínas naturales y nunca se ha observado en forma de una estructura de superhélice regular que se compone de, y se estabiliza mediante, motivos de repetición en héptada convencionales. La tabla 1 muestra una lista exhaustiva de 179 complejos peptídicos de superhélice de triple hebra a partir del PDB, 175 de los cuales son paralelos y sólo 4 son antiparalelos. Esto sugiere que una orientación paralela es la configuración más estable para las superhélices triméricas peptídicas. Un motivo probable para la abundancia de configuraciones paralelas es la conservación de simetría de 3 pliegues, que permite un número máximo de contactos óptimos. En cambio, todas las realizaciones de la presente invención se refieren a superhélices de una sola cadena que adoptan una orientación antiparalela. En vista de los escasos ejemplos de estructuras de superhélice de triple hebra antiparalela en el PDB, el diseño y la creación de tales estructuras no son obvios... [Seguir leyendo]
Reivindicaciones:
Proteína de una sola cadena aislada que consiste en la fórmula HRS1-L1-HRS2-L2-HRS3, en la que HRS1, L1, HRS2, L2 y HRS3 representan fragmentos de secuencia de aminoácidos que están ¡nterconectados covalentemente, plegándose espontáneamente dicha proteína en disolución acuosa por medio de los fragmentos HRS1, HRS2 y HRS3 que forman una estructura de superhélice alfa-helicoidal, antiparalela, de triple hebra, y en la que
cada uno de HRS1, HRS2 y HRS3 es independientemente una secuencia de repetición en héptada que se caracteriza por un patrón de 7 residuos repetido n veces de tipos de aminoácidos, representado como (a-b- c-d-e-f-g-)n o (d-e-f-g-a-b-c-)n y una repetición en héptada parcial C-terminal que termina con un residuo de núcleo de héptada ubicado en una posición a o d, en la que los elementos de patrón "a" a "g" indican posiciones de héptada convencionales en las que se ubican dichos tipos de aminoácidos y n es un número igual a o mayor de 2, y
al menos el 50%, el 70%, el 90% o el 100% de las posiciones de héptada convencionales "a" y "d" están ocupadas por aminoácidos seleccionados del grupo que consiste en valina, isoleucina, leucina, metionina, fenilalanina, tirosina, triptófano, histidina, glutamina, treonina, serina, alanina o derivados de los mismos, en la que al menos el 50%, el 70%, el 90% o en la que el 100% de las posiciones de héptada convencionales "b", "c", "e", "f y "g" están ocupadas por aminoácidos seleccionados del grupo que consiste en glicina, alanina, cisterna, serina, treonina, histidina, asparagina, ácido aspártico, glutamina, ácido glutámico, lisina, arginina o derivados de los mismos, permitiendo la distribución resultante de los tipos de aminoácidos la identificación de dichas secuencias de repetición en héptada, y
cada uno de L1 y L2 es independientemente un ligador que consiste en de 1 a 30 residuos de aminoácido, incluyendo este ligador cualquier residuo de aminoácido que no puede asignarse de manera inequívoca a una secuencia de repetición en héptada, y
L1 y L2 tienen una composición de aminoácidos que comprende al menos el 50% de aminoácidos seleccionados del grupo que consiste en glicina, alanina, serina, treonina, prolina o derivados de los mismos.
Proteína aislada según la reivindicación 1, en la que al menos el 50%, el 70%, el 90% o en la que el 100% de las posiciones de héptada convencionales "a" y "d" están ocupadas por aminoácidos seleccionados del grupo que consiste en valina, isoleucina, leucina, metionina o derivados de los mismos.
Proteína aislada según cualquiera de las reivindicaciones 1 a 2, en la que al menos el 50%, el 70%, el 90%, o en la que el 100% de las posiciones de héptada convencionales "a" y "d" están ocupadas por isoleucinas.
Proteína aislada según cualquiera de las reivindicaciones 1 a 3, en la que L1 y L2 tienen una composición
de aminoácidos que comprende al menos el 50%, el 70%, el 90% o que comprende el 100% de
aminoácidos seleccionados del grupo que consiste en glicina, alanina, cisteína, prolina, serina, treonina, histidina, asparagina, ácido aspártico, glutamina, ácido glutámico, lisina, arginina o derivados de los
mismos.
Proteína aislada según cualquiera de las reivindicaciones 1 a 4, en la que L1 y L2 tienen una composición de aminoácidos que comprende al menos el 70%, el 90% o que comprende el 100% de aminoácidos seleccionados del grupo que consiste en glicina, alanina, serina, treonina, prolina o derivados de los
mismos.
Proteína aislada según cualquiera de las reivindicaciones 1 a 5, en la que L1 y L2 tienen una composición
de aminoácidos que comprende al menos el 50%, el 70%, el 90% o que comprende el 100% de
aminoácidos glicina y/o serina.
Proteína aislada según cualquiera de las reivindicaciones 1 a 6, en la que el número de residuos de aminoácido de cada uno de L1 y L2 equivale a menos de la mitad del número de residuos de aminoácido de la secuencia de repetición en héptada que precede a los respectivos L1 o L2.
Proteína aislada según cualquiera de las reivindicaciones 1 a 7, en la que residuos de aminoácido cerca de los extremos terminales de L1 y/o L2 estabilizan los extremos alfa-helicoidales de la estructura de superhélice.
Proteína aislada según cualquiera de las reivindicaciones 1 a 8, en la que residuos de aminoácido cerca de los extremos terminales de L1 y/o L2 promueven la formación de un giro local en la estructura.
Proteína aislada según cualquiera de las reivindicaciones 1 a 9, en la que las posiciones de héptada
convencionales "e" y "g" están ocupadas por glutaminas.
11. Proteína aislada según cualquiera de las reivindicaciones 1 a 10, en la que las posiciones de héptada convencionales "b", "c" y "P son aminoácidos polares que promueven la solubilidad.
12. Proteína aislada según cualquiera de las reivindicaciones 1 a 11, que se pliega en disolución acuosa que
tiene un pH de entre 1 y 13, o entre 2 y 12, o entre 3 y 11, o entre 4 y 10, o entre 5 y 9.
13. Proteína aislada según cualquiera de las reivindicaciones 1 a 12, que se pliega en disolución acuosa que
tiene una temperatura de entre 0°C y 100°C, o entre 0°C y 80°C, o entre 0°C y 60°C.
14. Proteína aislada según cualquiera de las reivindicaciones 1 a 13, que se pliega en disolución acuosa que tiene una fuerza iónica de entre 0 y 1,0 molar.
15. Uso de la proteína aislada según cualquiera de las reivindicaciones 1 a 14 como armazón.
16. Ácido nucleico que codifica para una proteína según cualquiera de las reivindicaciones 1 a 14.
17. Vector que comprende un ácido nucleico según la reivindicación 16.
18. Célula huésped que comprende un ácido nucleico o vector según la reivindicación 16 ó 18.
19. Método para la producción de una proteína según cualquiera de las reivindicaciones 1 a 14 que comprende introducir un ácido nucleico o vector en una célula huésped, cultivar dicha célula huésped en un medio en condiciones en las que se expresa el ácido nucleico y se produce la proteína, y aislar la proteína de dicha célula huésped y/o dicho medio.
Patentes similares o relacionadas:
Formulación de inmunoglobulina y procedimiento de preparación de la misma, del 17 de Junio de 2020, de Biogen MA Inc: Una formulación acuosa estable que comprende natalizumab en una concentración de 15 mg/mL a 50 mg/mL, un tampón fosfato, polisorbato 80 en […]
Arginina desiminasa con reactividad cruzada reducida hacia anticuerpos para ADI - PEG 20 para el tratamiento del cáncer, del 6 de Mayo de 2020, de TDW Group: Una composición terapéutica que comprende una arginina desiminasa (ADI) aislada y un vehículo farmacéuticamente aceptable, en donde la ADI aislada comprende la secuencia de […]
Proteínas variantes de empalme her2 y her3 solubles, oligonucleótidos de cambio de empalme y su uso en el tratamiento de enfermedades, del 15 de Abril de 2020, de Sarepta Therapeutics, Inc: Una proteína del receptor 2 del factor de crecimiento epidérmico humano (HER2) aislada y soluble que consiste en la secuencia establecida en la SEQ ID NO: […]
Composiciones y métodos para la distribución de moléculas en células vivas, del 25 de Marzo de 2020, de THE TEXAS A & M UNIVERSITY SYSTEM: Un compuesto que tiene la fórmula: **(Ver fórmula)** En donde X es un resto de unión, Y es un residuo de aminoácido acoplado de forma covalente […]
Nuevas composiciones inmunogénicas para la prevención y tratamiento de enfermedad meningocócica, del 11 de Marzo de 2020, de WYETH HOLDINGS LLC: Una composición que comprende al menos una proteína que comprende una secuencia de aminoácidos que tiene identidad de secuencia mayor de 80 % con la […]
Vacuna peptídica para la prevención e inmunoterapia de demencia del tipo Alzheimer, del 4 de Marzo de 2020, de UNITED BIOMEDICAL, INC.: Una composición que comprende una combinación de construcciones de inmunógenos de péptidos Aβ que consiste en las secuencias de aminoácidos de SEQ […]
Composiciones y métodos para inmunomodulación en un organismo, del 19 de Febrero de 2020, de UNIVERSITY OF CONNECTICUT: Una composicion para su uso en un metodo de (i) tratamiento de cancer en un ser humano; (ii) mejora de la vacunacion en un ser humano; (iii) aumento de las respuestas […]
Uso de moléculas inhibidoras de semaforina-4D en combinación con una terapia inmunomoduladora para inhibir el crecimiento tumoral y la metástasis, del 8 de Enero de 2020, de Vaccinex, Inc: Un anticuerpo aislado o fragmento de unión a un antígeno del mismo que se une específicamente a semaforina-4D (SEMA4D) para su uso en la inhibición, el retraso o la reducción […]