Vectores alfavirales y líneas celulares para la producción de proteínas recombinantes.
Vectores alfavirales y líneas celulares para la producción de proteínas recombinantes.
La presente invención se relaciona con polinucleótidos y vectores alfavirales para la expresión de genes de interés en células de mamífero. Adicionalmente, la invención se relaciona con células que comprenden dichos polinucleótidos y vectores alfavirales, y que son capaces de expresar de forma estable uno o más genes de interés. La invención también se relaciona con métodos para obtener dichas células, con métodos para expresar un gen de interés en dichas células, y con métodos para reemplazar el gen de interés que dichas células expresan de forma estable por otro gen de interés.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201330722.
Solicitante: 3P BIOPHARMACEUTICALS.
Nacionalidad solicitante: España.
Inventor/es: SOLDEVILA FABREGA,ANDREU, SMERDOU PICAZO, CRISTIAN, ARANDA,Alejandro, BEZUNARTEA BEZUNARTEA,Jaione, MOLERO SÁNCHEZ,Dámaso, MISTINIENE,Edita, SERRANO PÉREZ-NIEVAS,Paula.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/86 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Vectores virales.
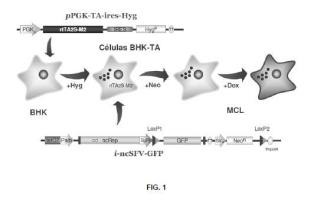
Fragmento de la descripción:
VECTORES ALFAVIRALES Y LÃ?NEAS CELULARES PARA LA PRODUCCIÓN DE PROTEÃ?NAS RECOMBINANTES
CAMPO DE LA INVENCIÓN
La presente invención se encuadra dentro del campo de los vectores para la expresión de genes de interés en células de mamífero, concretamente, en el campo de los vectores alfavirales. El uso de dichos vectores permite la generación de líneas celulares que expresan de forma estable un gen de interés.
ANTECEDENTES DE LA INVENCIÓN
Las células de mamífero son el sistema de expresión más adecuado para la obtención de diferentes tipos de proteínas recombinantes, en especial aquellas proteínas destinadas a aplicaciones terapéuticas. Entre los vectores de expresión más eficaces para incorporar un gen de interés en una célula con el fin de expresar una proteína de interés se encuentran los vectores basados en alfavirus. Se han desarrollado vectores de expresión alfavirales a partir
de diferentes tipos de alfavirus, incluyendo el virus Sindbis (SIN) , el virus del Bosque de Semliki (SFV) y el virus de la encefalitis equina venezolana (VEE) .
El replicón del alfavirus contiene en su extremo 5’ un marco abierto de lectura que codifica la replicasa (Rep) vírica, que se traduce cuando el ARN viral se transfecta en células eucariotas. La Rep se expresa como una poliproteína que se procesa, posteriormente, en cuatro subunidades (nsps 1 a 4) . La Rep sin procesar puede copiar el vector de ARN en una cadena negativa de ARN, un proceso que sólo tiene lugar durante las primeras 3 a 4 horas tras la transfección o infección. Una vez procesada, la Rep utilizará el ARN de cadena negativa como molde para sintetizar más moléculas del replicón. La Rep procesada también puede reconocer una secuencia interna en el ARN de cadena negativa, o promotor subgenómico, a partir del cual sintetizará un ARN de cadena positiva subgenómico que corresponde al extremo 3’ del replicón. Este ARN subgenómico se traducirá para producir la proteína heteróloga en grandes cantidades.
Normalmente, los vectores de alfavirus están basados en replicones de ARN en los que los genes estructurales se han sustituido por un gen heterólogo. Sin embargo, la replicación de la mayoría de los vectores alfavirales es citopática, por lo que dichos vectores no permiten obtener una expresión duradera del gen de interés. Para solventar ese problema, varios grupos han identificado una serie de mutaciones en la replicasa de los alfavirus que pueden convertir esos vectores virales citopáticos en vectores virales no citopáticos, lo que permite una expresión más duradera de los productos recombinantes expresados por el vector viral. Estos estudios han conducido a la generación de diferentes vectores no citopáticos derivados de alfavirus.
Un mutante no citopático aislado de SIN, que contenía un sólo cambio de aminoácido (P por L) en la posición 726 en nsp2 (vector de SIN P726L en nsp2) , mostró un hiperprocesamiento de la Rep (Frolov et al., 1999, J. Virol. 73: 3854-65) . Este mutante fue capaz de establecer replicación continua en células BHK de manera eficaz. Este vector no citopático de SIN se ha utilizado ampliamente in vitro, ya que es capaz de proporcionar una expresión duradera del transgén con buenos niveles de estabilidad y niveles de expresión que eran aproximadamente el 4% de los obtenidos con el vector de SIN original (Agapov et al., 1998, Proc. Natl. Acad. Sci. USA. 95: 12989-94) . No obstante, a pesar de que dicho vector no es citopático, carece de capacidad para generar líneas celulares estables con altos niveles de expresión.
Respecto a los mutantes no citopáticos de SFV descritos por Perri et al. (Perri et al., 2000, J. Virol. 74: 9802-7) , que incluyen los mutantes SF2A (mutación L10T en nsp2) y SF2C (mutación L713P en nsp2) , así como el doble mutante PD (S259P y R650D en nsp2) descrito por Lundström et al. (Lundström et al., 2001. Histochem. Cell. Biol. 115: 83-91) , si bien pueden expresar niveles de proteína similares o incluso superiores a los del virus de tipo silvestre (mutante PD) , en todos los casos esos mutantes continúan siendo citopáticos y no se ha descrito la generación de líneas celulares estables que expresen proteínas heterólogas basadas en dichos vectores virales.
La solicitud de patente WO2008065225 describe un vector SFV no citopático gracias a la presencia de las mutaciones R649H/P718T en la subunidad nsp2 de la replicasa. Dicho vector permite la obtención de líneas celulares capaces de expresar el gen de interés de forma constitutiva y estable mediante el cultivo en presencia de un antibiótico cuyo gen de resistencia está incorporado en el vector alfaviral (Casales et al. 2008. Virology. 376:24251) .
La utilización de vectores virales basados en replicones no citopáticos tiene asociado el inconveniente de que se pueden acumular mutaciones en el gen de interés, debido a que la replicasa viral, que carece de actividad correctora de errores, está constantemente copiando el ARN viral. Un segundo inconveniente de estos sistemas es el hecho de que no permiten la expresión de proteínas tóxicas para la célula ya que la expresión se produce de forma constitutiva. Finalmente en estos sistemas con frecuencia se observa una disminución en los niveles de expresión del gen de interés lo que puede deberse, en parte, a que la adaptación de las células a crecer en presencia del antibiótico, necesario para mantener la expresión del replicón alfaviral puede hacer que las células necesiten menos cantidad de replicón para sobrevivir. Estos problemas podrían solucionarse teóricamente mediante el diseño de un vector viral integrado en el genoma celular y en el que la expresión del replicón alfaviral pueda ser regulable mediante promotores inducibles u otros sistemas (WO9738087, WO2000006205 y WO2001081553) . Sin embargo, no existen evidencias de que este tipo de vectores hayan permitido obtener una elevada expresión de las proteínas de interés de una forma sostenida en el tiempo. El uso de secuencias de ADN complementarias a las secuencias de ARN que constituyen el replicón alfaviral ha permitido diseñar vectores alfavirales que permitan la integración del gen de interés en el genoma celular, favoreciendo de este modo la obtención de una línea celular capaz de expresar de forma estable el gen de interés sin necesidad de añadir un antibiótico. Sin embargo, las líneas celulares obtenidas mediante estos vectores con frecuencia muestran una gran variabilidad en los niveles de expresión de las proteínas de interés, así como, en su caso, en la inducibilidad de la expresión de estas proteínas, probablemente debido a diferencias en el sitio de integración y el número de integraciones en el genoma celular, por lo que es necesario el aislamiento y selección de aquellos clones con características óptimas.
Por lo tanto, existe la necesidad de proporcionar un sistema de expresión de genes de interés en células de mamífero que permita la expresión de dichos genes de forma estable y en cantidades elevadas, de forma que sea adecuado para la producción a gran escala de proteínas recombinantes.
COMPENDIO DE LA INVENCIÓN
En un primer aspecto, la invención se relaciona con un nucleótido que comprende (i) una secuencia reguladora de la transcripción,
(ii) una secuencia de ADN complementaria a un replicón de un alfavirus que se encuentra operativamente unida a dicha secuencia reguladora de la transcripción y en donde dicha secuencia complementaria del replicón del alfavirus comprende una primera secuencia de reconocimiento para una recombinasa específica de sitio situada entre el promotor subgenómico del replicón del alfavirus y la secuencia 3´ no traducida del replicón del alfavirus,
(iii) una secuencia de terminación de la transcripción en posición 3’ con respecto al extremo 3’ de la secuencia complementaria a un replicón de un alfavirus y
(iv) una segunda secuencia de reconocimiento para una recombinasa específica de sitio situada en posición 3’ respecto a la secuencia de terminación de la transcripción.
En un segundo aspecto, la invención se relaciona con un vector de expresión que comprende el polinucleótido del primer aspecto.
En un tercer aspecto, la invención se relaciona con una célula eucariota que comprende el polinucleótido del primer aspecto o el vector de expresión del segundo aspecto.
En un cuarto aspecto, la invención se relaciona con una célula eucarita que comprende el
polinucleótido del primer aspecto en donde dicho polinucleótido se encuentra integrado en su genoma.
En un quinto aspecto, la invención se relaciona con un método in vitro para generar una línea...
Reivindicaciones:
1. Un polinucleótido que comprende (i) una secuencia reguladora de la transcripción,
(ii) una secuencia de ADN complementaria a un replicón de un alfavirus que se encuentra operativamente unida a dicha secuencia reguladora de la transcripción y en donde dicha secuencia complementaria del replicón del alfavirus comprende una primera secuencia de reconocimiento para una recombinasa específica de sitio situada entre el promotor subgenómico del replicón del alfavirus y la secuencia 3´ no traducida del replicón del alfavirus,
(iii) una secuencia de terminación de la transcripción en posición 3’ con respecto al extremo 3’ de la secuencia complementaria a un replicón de un alfavirus y
(iv) una segunda secuencia de reconocimiento para una recombinasa específica de sitio situada en posición 3’ respecto a la secuencia de terminación de la 15 transcripción.
2. El polinucleótido según la reivindicación 1 en donde las secuencias de reconocimiento para una recombinasa específica de sitio son secuencias heteroespecíficas.
3. El polinucleótido según las reivindicaciones 1 o 2 en donde las secuencias de reconocimiento para una recombinasa específica de sitio son secuencias LoxP.
4. El polinucleótido según la reivindicación 3 en donde la primera y la segunda secuencia LoxP se seleccionan del grupo formado por las parejas 25 (a) SEQIDNO:9ySEQIDNO:10
(b) SEQIDNO:11ySEQIDNO:12
(c) SEQIDNO:9ySEQIDNO:12
(d) SEQIDNO:11ySEQIDNO:10
(e) SEQIDNO:10ySEQIDNO:9 30 (f) SEQIDNO:12ySEQIDNO:11
(g) SEQIDNO:10ySEQIDNO:11 y
(h) SEQIDNO:12ySEQIDNO:9
5. El polinucleótido según la reivindicación 4 en donde la secuencia reguladora de la 35 transcripción comprende un promotor específico de hígado.
6. El polinucleótido según la reivindicación 5 en donde el promotor específico de hígado es el promotor del gen de albúmina o una variante funcionalmente equivalente del mismo.
7. El polinucleótido según cualquiera de las reivindicaciones 1 a 6 en donde la secuencia 5 reguladora de la transcripción tiene una actividad regulable.
8. El polinucleótido según la reivindicación 7 en donde la secuencia reguladora de la transcripción que tiene una actividad regulable comprende adicionalmente un elemento de respuesta a un agente inductor.
9. El polinucleótido según la reivindicación 8 en donde el elemento de respuesta a un agente inductor es un elemento de respuesta a un ligando.
10. El polinucleótido según la reivindicación 9 en donde el elemento de respuesta a un 15 ligando es un elemento de respuesta a tetraciclina o a un análogo de tetraciclina.
11. El polinucleótido según cualquiera de las reivindicaciones 5 a 10 en donde la secuencia reguladora de la transcripción comprende la secuencia de nucleótidos SEQ ID NO: 5.
12. El polinucleótido según la reivindicación 11 en donde la secuencia reguladora de la transcripción comprende la secuencia de nucleótidos SEQ ID NO: 8 y en donde la secuencia reguladora de la transcripción no comprende la secuencia SEQ ID NO: 4.
13. El polinucleótido según cualquiera de las reivindicaciones 1 a 12 en donde la secuencia de ADN complementaria a un replicón de un alfavirus además comprende una secuencia heteróloga en posición 3’ respecto al promotor subgenómico del replicón y respecto a la primera secuencia de reconocimiento para una recombinasa específica de sitio.
14. El polinucleótido según la reivindicación 13 en donde dicha secuencia heteróloga se selecciona del grupo formado por
(i) una secuencia de relleno,
(ii) una secuencia que comprende un sitio múltiple de clonaje,
(iii) una secuencia de un gen de interés unida operativamente al promotor 35 subgenómico del replicón del alfavirus y
(iv) cualquier combinación de las mismas.
15. El polinucleótido según la reivindicación 14 en donde dicha secuencia heteróloga es una secuencia de un gen de interés unida operativamente al promotor subgenómico del replicón del alfavirus y en donde el gen de interés codifica una proteína de interés o un precursor de la misma.
16. El polinucleótido según la reivindicación 15 en donde la proteína de interés adicionalmente comprende al menos una secuencia etiqueta.
17. El polinucleótido según cualquiera de las reivindicaciones 14 a 16 en donde el replicón del alfavirus comprende al menos una secuencia de un segundo gen de interés, en donde dicha secuencia de un segundo gen de interés se encuentra en posición 3’ respecto a la primera secuencia de reconocimiento para una recombinasa específica de sitio y en donde dicha secuencia de un segundo gen de interés está operativamente unida a un promotor subgenómico adicional o se encuentra operativamente asociada a la secuencia del primer gen de interés por medio de un IRES o de una secuencia que codifica un sitio de corte proteolítico post-traduccional.
18. El polinucleótido según la reivindicación 17 en donde la secuencia del segundo gen de interés está operativamente asociada a la secuencia del primer gen de interés por medio de una secuencia que codifica un sitio de corte proteolítico post-traduccional y en donde dicha secuencia que codifica un sitio de corte proteolítico post-traduccional es una secuencia que codifica una autoproteasa.
19. El polinucleótido según la reivindicación 18 en donde la autoproteasa es la autoproteasa 2A del virus de la fiebre aftosa.
20. El polinucleótido según cualquiera de las reivindicaciones 17 a 19 en donde el primer gen de interés y el segundo gen de interés codifican, respectivamente, una primera y una segunda cadena de un anticuerpo.
21. El polinucleótido según cualquiera de las reivindicaciones 1 a 20 en donde el replicón de un alfavirus es un replicón del Semliki Forest Virus.
22. El polinucleótido según la reivindicación 21 en donde el replicón del Semliki Forest Virus comprende al menos una mutación que confiere a dicho replicón un fenotipo no citopático.
23. El polinucleótido según la reivindicación 22 en donde dicha mutación se localiza en la región que codifica la replicasa.
24. El polinucleótido según la reivindicación 23 en donde dicha mutación se localiza en la región que codifica la subunidad nsp2 de dicha replicasa. 10
25. El polinucleótido según la reivindicación 24 en donde dicha mutación se selecciona del grupo formado por las mutaciones P718T, R649H y una combinación de ambas.
26. El polinucleótido según cualquiera de las reivindicaciones 1 a 25 que comprende
adicionalmente un gen de selección, en donde dicho gen de selección se localiza en la región comprendida entre el primer y el segundo sitio de reconocimiento de la recombinasa y en donde (i) si la secuencia reguladora de la transcripción que está unida operativamente a la secuencia complementaria del replicón del alfavirus es una secuencia regulable,
entonces el gen de selección se encuentra fuera de dicho replicón y unido a un promotor constitutivo o (ii) si la secuencia reguladora de la transcripción que está unida operativamente a la secuencia complementaria del replicón del alfavirus comprende un promotor constitutivo, entonces dicho gen de selección está operativamente asociado al
gen de interés por medio de un IRES o de una secuencia que codifica un sitio de corte proteolítico post-traduccional, o dicho gen de selección está bajo el control de un segundo promotor constitutivo.
27. El polinucleótido según la reivindicación 26 en donde el gen de selección está
operativamente asociado al gen de interés por medio de una secuencia que codifica un sitio de corte proteolítico post-traduccional y en donde dicha secuencia que codifica un sitio de corte proteolítico post-traduccional es una secuencia que codifica una autoproteasa.
28. El polinucleótido según la reivindicación 27 en donde la autoproteasa es la autoproteasa 2A del virus de la fiebre aftosa.
29. El polinucleótido según cualquiera de las reivindicaciones 26 a 28 en donde el gen de selección se encuentra operativamente unido a un promotor constitutivo.
30. El polinucleótido según la reivindicación 29 en donde el promotor constitutivo al que se encuentra unido el gen de selección comprende la secuencia de nucleótidos que se muestra en SEQ ID NO: 14.
31. El polinucleótido según cualquiera de las reivindicaciones 26 a 30 en donde el gen de 10 selección es un gen que confiere resistencia a un antibiótico.
32. El polinucleótido según la reivindicación 31 en donde el gen que confiere resistencia a un antibiótico se selecciona del grupo formado por el gen de resistencia a neomicina y el gen de resistencia a puromicina.
33. El polinucleótido según cualquiera de las reivindicaciones 1 a 32 en donde la secuencia de terminación de la transcripción comprende la secuencia SEQ ID NO: 13.
34. El polinucleótido según cualquiera de las reivindicaciones 1 a 33 que adicionalmente
comprende una señal de poliadenilación o una señal de terminación de la transcripción y poliadenilación en posición 3’ respecto al segundo sitio de reconocimiento de la recombinasa.
35. El polinucleótido según la reivindicación 34 en donde la señal de terminación de la 25 transcripción y poliadenilación comprende la secuencia SEQ ID NO: 70.
36. Un vector de expresión que comprende el polinucléotido según cualquiera de las reivindicaciones 1 a 35.
37. Una célula eucariota que comprende un polinucleótido según cualquiera de las reivindicaciones 1 a 35 o un vector de expresión según la reivindicación 36.
38. Una célula eucariota que comprende un polinucleótido según cualquiera de las reivindicaciones 1 a 35 en donde el polinucleótido se encuentra integrado en su 35 genoma.
39. La célula según cualquiera de las reivindicaciones 37 o 38 en donde la secuencia reguladora de la transcripción que está unida operativamente a la secuencia complementaria del replicón del alfavirus es una secuencia regulable y en donde dicha célula comprende adicionalmente una secuencia de ADN que codifica un activador transcripcional unido operativamente a un promotor constitutivo, en donde dicho activador transcripcional es capaz de regular la transcripción a partir de dicha secuencia reguladora de la transcripción mediante su unión al sitio de respuesta a un ligando.
40. La célula según la reivindicación 39 en donde la secuencia de ADN que codifica un activador transcripcional está integrada en el genoma.
41. La célula según cualquiera de las reivindicaciones 39 o 40 en donde el ligando es tetraciclina o un análogo de tetraciclina. 15
42. La célula según la reivindicación 41 en donde el análogo de tetraciclina es doxiciclina.
43. La célula según la reivindicación 42 en donde el activador transcripcional regulable por
doxiciclina comprende la secuencia SEQ ID NO: 16. 20
44. La célula según cualquiera de las reivindicaciones 37 a 43 en donde la secuencia de ADN que codifica un activador transcripcional comprende adicionalmente un gen de selección operativamente asociado a la secuencia que codifica dicho activador transcripcional por medio de un IRES o de una secuencia que codifica un sitio de corte proteolítico post-traduccional.
45. La célula según la reivindicación 44 en donde el gen de selección está operativamente asociado a la secuencia que codifica el activador transcripcional por medio de una secuencia que codifica un sitio de corte proteolítico post-traduccional y en donde dicha secuencia que codifica un sitio de corte proteolítico post-traduccional es una secuencia que codifica una autoproteasa.
46. La célula según la reivindicación 45 en donde la autoproteasa es la autoproteasa 2A del
virus de la fiebre aftosa. 35
47. La célula según cualquiera de las reivindicaciones 44 a 46 en donde el gen de selección codifica una proteína que confiere resistencia a higromicina.
48. La célula según cualquiera de las reivindicaciones 44 a 47 en donde dicha célula 5 eucariota es una célula de mamífero.
49. La célula según cualquiera de las reivindicaciones 44 a 48 en donde dicha célula eucariota es una célula que puede crecer en suspensión.
50. Un método in vitro para generar una línea celular capaz de expresar un gen de interés que comprende (i) poner en contacto una célula con un polinucléotido según cualquiera de las reivindicaciones 1 a 35, en donde dicho polinucléotido comprende adicionalmente dicho gen de interés unido operativamente al promotor subgenómico del replicón,
o con un vector de expresión que comprende dicho polinucleótido y
(ii) seleccionar las células que han incorporado dicho polinucléotido o un vector de expresión que comprende dicho polinucleótido.
51. El método según la reivindicación 50 en donde la secuencia reguladora de la transcripción que está operativamente unida a la secuencia complementaria del replicón del alfavirus es una secencia regulable y en donde dicho método comprende adicionalmente las etapas de (i) introducir en dicha célula un polinucléotido que codifica un activador transcripcional específico para dicha secuencia reguladora de la transcripción y
(ii) seleccionar las células que hayan incorporado dicho polinucléotido que codifica un activador transcripcional específico para dicha secuencia reguladora de la transcripción.
52. El método según la reivindicación 51 en donde el activador transcripcional es un 30 activador transcripcional regulable por un ligando.
53. El método según la reivindicación 52 en donde el ligando es tetraciclina o un análogo de tetraciclina.
54. El método según la reivindicación 53 en donde el análogo de tetraciclina es doxiciclina.
55. El método según la reivindicación 54 en donde el activador transcripcional regulable por doxiciclina comprende la secuencia SEQ ID NO: 16.
56. El método según cualquiera de las reivindicaciones 50 a 55 en donde el polinucléotido
de acuerdo a las reivindicaciones 1 a 35 comprende adicionalmente un primer gen de selección y/o el polinucléotido que codifica un activador transcripcional específico para dicho promotor regulable comprende adicionalmente un segundo gen de selección, y en donde la etapa de selección de las células que han incorporado dicho polinucleótido de acuerdo a las reivindicaciones 1 a 35 que comprende un primer gen de selección y/o
dicho polinucléotido que codifica un activador transcripcional de forma estable se lleva a cabo en condiciones selectivas para las células que expresen dicho primer y/o dicho segundo gen de selección.
57. El método según la reivindicación 56 en donde el primer y el segundo gen de selección 15 son diferentes.
58. El método según la reivindicación 57 en donde el primer gen de selección se selecciona del gen que confiere resistencia a neomicina y del gen que confiere resistencia a puromicina, y el segundo gen de selección es el gen que confiere resistencia a higromicina.
59. Un método para expresar un gen de interés que comprende poner una célula de acuerdo con las reivindicaciones 37 a 49 en condiciones adecuadas para la activación de la transcripción de la secuencia complementaria al replicón alfaviral, en donde dicha célula comprende un polinucléotido según cualquiera de las reivindicaciones o un vector de expresión que comprende dicho polinucleótido 1 a 35, en donde dicho polinucleótido comprende adicionalmente la secuencia del gen de interés unida operativamente al promotor subgenómico.
60. El método según la reivindicación 59 en donde la secuencia reguladora de la transcripción que se encuentra unida operativamente a la secuencia complementaria al replicón del alfavirus es una secuencia regulable y en donde dichas condiciones adecuadas para la activación de la transcripción del replicón alfaviral comprenden condiciones adecuadas para la activación de dicha secuencia regulable.
61. El método según la reivindicación 60 en donde el promotor regulable se activa en presencia de un ligando y en donde dichas condiciones adecuadas para la activación de la transcripción del replicón alfaviral comprenden la puesta en contacto de la célula con dicho ligando o un precursor del mismo.
62. Un vector que comprende una secuencia de ADN que comprende, ordenados en la dirección 5’ a 3’,
(vii) una primera secuencia de reconocimiento para una recombinasa específica de sitio, 10 (viii) una secuencia de un gen de interés,
(ix) una secuencia 3’ necesaria para la replicación de un alfavirus,
(x) una secuencia de terminación de la transcripción,
(xi) una secuencia de un gen de selección unida operativamente a un promotor y
(xii) una segunda secuencia de reconocimiento para una recombinasa específica de sitio.
63. El vector según la reivindicación 61 que adicionalmente comprende al menos una secuencia de un gen de interés adicional en donde dicha secuencia está separada de la secuencia del primer gen de interés por un IRES, o por una secuencia que codifica un sitio de corte proteolítico post-traduccional, o dicha secuencia está unida operativamente a un promotor subgenómico situado entre la secuencia del gen de interés (ii) y la secuencia 3’ necesaria para la replicación del alfavirus (iii) .
64. El vector según la reivindicación 62 en donde la secuencia del gen de interés adicional está separada de la secuencia del primer gen de interés por una secuencia que codifica un sitio de corte proteolítico post-traduccional, y en donde dicha secuencia que codifica un sitio de corte proteolítico post-traduccional es una secuencia que codifica una autoproteasa.
65. El vector según la reivindicación 63 en donde la autoproteasa es la autoproteasa 2A del virus de la fiebre aftosa.
66. El vector según cualquiera de las reivindicación 61 a 64en donde la secuencia 3’ necesaria para la replicación del alfavirus procede del Semliki Forest Virus.
67. El vector según cualquiera de las reivindicaciones 61 a 65 en donde las secuencias de reconocimiento para una recombinasa específica de sitio son secuencias heteroespecíficas.
68. El vector según cualquiera de las reivindicaciones 61 a 66 en donde las secuencias de reconocimiento para una recombinasa específica de sitio son secuencias LoxP.
69. El vector según la reivindicación 67 en donde la primera y la segunda secuencia LoxP
se seleccionan del grupo formado por las parejas 10 (a) SEQIDNO:9ySEQIDNO:10,
(b) SEQ ID NO: 11 y SEQ ID NO: 12,
(c) SEQIDNO:9ySEQIDNO:12,
(d) SEQ ID NO: 11 y SEQ ID NO: 10,
(e) SEQIDNO:10ySEQIDNO:9, 15 (f) SEQ ID NO: 12 y SEQ ID NO: 11,
(g) SEQIDNO:10ySEQIDNO:11y
(h) SEQIDNO:12ySEQIDNO:9.
70. El vector según cualquiera de las reivindicaciones 61 a 68 en donde el gen de interés 20 codifica una proteína de interés o un precursor de la misma.
71. El vector según la reivindicación 70 en donde la proteína de interés adicionalmente comprende una secuencia etiqueta.
72. El vector según cualquiera de las reivindicaciones 62 a 71 en donde la secuencia de terminación de la transcripción comprende la secuencia SEQ ID NO: 13.
73. El vector según cualquiera de las reivindicaciones 62 a 72 en donde el gen de selección carece de señal de poliadenilación. 30
74. Un método in vitro para generar una línea celular capaz de expresar un gen de interés que comprende las etapas de:
(i) introducir en una célula según cualquiera de las reivindicaciones 37 a 49 un vector de acuerdo a cualquiera de las reivindicaciones 62 a 73, en donde: 35 (f) dicha célula comprende una secuencia heteróloga en posición 3’
respecto al promotor subgenómico del replicón, en donde dicha 85
secuencia heteróloga es una secuencia de un gen de interés unida operativamente al promotor subgenómico, o un vector de expresión que comprende dicho polinucleótido,
(g) la primera secuencia de reconocimiento del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es compatible con la primera secuencia de reconocimiento heteroespecífica del vector,
(h) la segunda secuencia de reconocimiento del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es compatible con la segunda secuencia de reconocimiento heteroespecífica del vector
(i) la célula expresa una recombinasa específica de dichas primeras y segundas secuencias de reconocimiento y,
(j) la secuencia necesaria para la replicación del alfavirus presente en el
vector coincide con la secuencia necesaria para la replicación del alfavirus que forma parte del replicón alfaviral,
(ii) mantener la célula en condiciones adecuadas para permitir la sustitución mediante recombinación específica del gen de interés que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus por el gen de interés que forma parte del vector de acuerdo a las reivindicaciones 61 a 71 y
(iii) seleccionar las células en las que se ha producido la sustitución del primer gen de interés por el segundo gen de interés.
75. El método según la reivindicación 74 en donde la etapa de selección de las células en las que se ha producido la sustitución del primer gen de interés por el segundo gen de interés se lleva a cabo mediante selección por medio del gen de selección que forma parte del vector, en donde dicho gen de selección es distinto al que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus.
76. El método según las reivindicaciones 74 o 75 en donde si el gen de selección que forma parte del vector carece de la señal de poliadenilación, entonces la célula comprende una señal de poliadenilación en posición 3’ respecto al segundo sitio de reconocimiento de la recombinasa, dando lugar a la reconstitución de un sitio de poliadenilación en proximidad al gen de selección tras la recombinación.
77. El método según cualquiera de las reivindicaciones 74 a 76 en donde las secuencias de reconocimiento para una recombinasa específica de sitio son secuencias heteroespecíficas.
78. El método según cualquiera de las reivindicaciones 74 a 77 en donde las secuencias de reconocimiento para una recombinasa específica de sitio son secuencias LoxP y la recombinasa específica de sitio es la recombinasa Cre.
79. El método según la reivindicación 78 en donde -si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la secuencia SEQ ID NO: 9, entonces la segunda secuencia de reconocimiento que forma parte del vector es la secuencia SEQ ID NO: 10 y si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la secuencia SEQ ID NO: 11, entonces la segunda secuencia de reconocimiento que forma parte del vector es la secuencia SEQ ID NO: 12; -si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la secuencia SEQ ID NO: 9, entonces la segunda secuencia de reconocimiento que forma parte del vector es la secuencia SEQ ID NO: 12 y si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la secuencia SEQ ID
NO: 11, entonces la segunda secuencia de reconocimiento que forma parte del es la secuencia SEQ ID NO: 10 ; -si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la secuencia SEQ ID NO: 10, entonces la segunda secuencia de reconocimiento que forma parte del vector es la secuencia SEQ ID NO: 9 y si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la secuencia SEQ ID NO: 12, entonces la segunda secuencia de reconocimiento que forma parte del vector es la secuencia SEQ ID NO: 11 y
- si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la 87
secuencia SEQ ID NO: 10, entonces la segunda secuencia de reconocimiento que forma parte del vector es la secuencia SEQ ID NO: 11 y si la primera secuencia de reconocimiento que forma parte del polinucleótido que comprende la secuencia complementaria de un replicón de un alfavirus es la secuencia SEQ ID NO: 12, entonces la segunda secuencia de reconocimiento que forma parte del vector es la secuencia SEQ ID NO: 9.
DIBUJOS
FIG. 1
FIG. 2
FIG. 3
FIG. 3 (CONT.)
FIG. 4
FIG. 5
FIG. 6
FIG. 7
FIG. 8
FIG. 8 (CONT.)
FIG. 9
D
FIG. 9 (CONT.)
FIG. 10
100
101
FIG. 11
102
FIG. 12
103
FIG. 13
104
SEQUENCE LISTING
<110> 3P BIOPHARMACEUTICALS
<120> VECTORES ALFAVIRALES Y LÃ?NEAS CELULARES PARA LA PRODUCCIÓN DE PROTEÃ?NAS RECOMBINANTES
<130> P8332ES00
<160> 70
<170> PatentIn version 3.5
<210> 1
<211> 86
<212> DNA
<213> Semliki Forest virus
<400> 1 gatggcggat gtgtgacata cacgacgcca aaagattttg ttccagctcc tgccacctcc 60
gctacgcgag agattaacca cccacg 86
<210> 2
<211> 7296
<212> DNA
<213> Artificial Sequence
<220>
<223> secuencia que codifica la poliproteina no estructural del SFV con las mutaciones P718T y R649H
<400> 2 atggccgcca aagtgcatgt tgatattgag gctgacagcc cattcatcaa gtctttgcag 60 aaggcatttc cgtcgttcga ggtggagtca ttgcaggtca caccaaatga ccatgcaaat 120 gccagagcat tttcgcacct ggctaccaaa ttgatcgagc aggagactga caaagacaca 180 ctcatcttgg atatcggcag tgcgccttcc aggagaatga tgtctacgca caaataccac 240 tgcgtatgcc ctatgcgcag cgcagaagac cccgaaaggc tcgatagcta cgcaaagaaa 300 ctggcagcgg cctccgggaa ggtgctggat agagagatcg caggaaaaat caccgacctg 360 cagaccgtca tggctacgcc agacgctgaa tctcctacct tttgcctgca tacagacgtc 420 acgtgtcgta cggcagccga agtggccgta taccaggacg tgtatgctgt acatgcacca 480 acatcgctgt accatcaggc gatgaaaggt gtcagaacgg cgtattggat tgggtttgac 540 accaccccgt ttatgtttga cgcgctagca ggcgcgtatc caacctacgc cacaaactgg 600 gccgacgagc aggtgttaca ggccaggaac ataggactgt gtgcagcatc cttgactgag 660 ggaagactcg gcaaactgtc cattctccgc aagaagcaat tgaaaccttg cgacacagtc 720 atgttctcgg taggatctac attgtacact gagagcagaa agctactgag gagctggcac 780 ttaccctccg tattccacct gaaaggtaaa caatccttta cctgtaggtg cgataccatc 840
gtatcatgtg aagggtacgt agttaagaaa atcactatgt gccccggcct gtacggtaaa 900 acggtagggt acgccgtgac gtatcacgcg gagggattcc tagtgtgcaa gaccacagac 960 actgtcaaag gagaaagagt ctcattccct gtatgcacct acgtcccctc aaccatctgt 1020 gatcaaatga ctggcatact agcgaccgac gtcacaccgg aggacgcaca gaagttgtta 1080 gtgggattga atcagaggat agttgtgaac ggaagaacac agcgaaacac taacacgatg 1140 aagaactatc tgcttccgat tgtggccgtc gcatttagca agtgggcgag ggaatacaag 1200 gcagaccttg atgatgaaaa acctctgggt gtccgagaga ggtcacttac ttgctgctgc 1260 ttgtgggcat ttaaaacgag gaagatgcac accatgtaca agaaaccaga cacccagaca 1320 atagtgaagg tgccttcaga gtttaactcg ttcgtcatcc cgagcctatg gtctacaggc 1380 ctcgcaatcc cagtcagatc acgcattaag atgcttttgg ccaagaagac caagcgagag 1440 ttaatacctg ttctcgacgc gtcgtcagcc agggatgctg aacaagagga gaaggagagg 1500 ttggaggccg agctgactag agaagcctta ccacccctcg tccccatcgc gccggcggag 1560 acgggagtcg tcgacgtcga cgttgaagaa ctagagtatc acgcaggtgc aggggtcgtg 1620 gaaacacctc gcagcgcgtt gaaagtcacc gcacagccga acgacgtact actaggaaat 1680 tacgtagttc tgtccccgca gaccgtgctc aagagctcca agttggcccc cgtgcaccct 1740 ctagcagagc aggtgaaaat aataacacat aacgggaggg ccggcggtta ccaggtcgac 1800 ggatatgacg gcagggtcct actaccatgt ggatcggcca ttccggtccc tgagtttcaa 1860 gctttgagcg agagcgccac tatggtgtac aacgaaaggg agttcgtcaa caggaaacta 1920 taccatattg ccgttcacgg accgtcgctg aacaccgacg aggagaacta cgagaaagtc 1980 agagctgaaa gaactgacgc cgagtacgtg ttcgacgtag ataaaaaatg ctgcgtcaag 2040 agagaggaag cgtcgggttt ggtgttggtg ggagagctaa ccaacccccc gttccatgaa 2100 ttcgcctacg aagggctgaa gatcaggccg tcggcaccat ataagactac agtagtagga 2160 gtctttgggg ttccgggatc aggcaagtct gctattatta agagcctcgt gaccaaacac 2220 gatctggtca ccagcggcaa gaaggagaac tgccaggaaa tagttaacga cgtgaagaag 2280 caccgcggga aggggacaag tagggaaaac agtgactcca tcctgctaaa cgggtgtcgt 2340 cgtgccgtgg acatcctata tgtggacgag gctttcgctt gccattccgg tactctgctg 2400 gccctaattg ctcttgttaa acctcggagc aaagtggtgt tatgcggaga ccccaagcaa 2460 tgcggattct tcaatatgat gcagcttaag gtgaacttca accacaacat ctgcactgaa 2520 gtatgtcata aaagtatatc cagacgttgc acgcgtccag tcacggccat cgtgtctacg 2580 ttgcactacg gaggcaagat gcgcacgacc aacccgtgca acaaacccat aatcatagac 2640
accacaggac agaccaagcc caagccagga gacatcgtgt taacatgctt ccgaggctgg 2700 gcaaagcagc tgcagttgga ctaccgtgga cacgaagtca tgacagcagc agcatctcag 2760 ggcctcaccc gcaaaggggt atacgccgta aggcagaagg tgaatgaaaa tcccttgtat 2820 gcccctgcgt cggagcacgt gaatgtactg ctgacgcgca ctgaggatag gctggtgtgg 2880 aaaacgctgg ccggcgatcc ctggattaag gtcctatcaa acattccaca gggtaacttt 2940 acggccacat tggaagaatg gcaagaagaa cacgacaaaa taatgaaggt gattgaagga 3000 ccggctgcgc ctgtggacgc gttccagaac aaagcgaacg tgtgttgggc gaaaagcctg 3060 gtgcctgtcc tggacactgc cggaatcaga ttgacagcag aggagtggag caccataatt 3120 acagcattta aggaggacag agcttactct ccagtggtgg ccttgaatga aatttgcacc 3180 aagtactatg gagttgacct ggacagtggc ctgttttctg ccccgaaggt gtccctgtat 3240 tacgagaaca accactggga taacagacct ggtggaagga tgtatggatt caatgccgca 3300 acagctgcca ggctggaagc tagacatacc ttcctgaagg ggcagtggca tacgggcaag 3360 caggcagtta tcgcagaaag aaaaatccaa ccgctttctg tgctggacaa tgtaattcct 3420 atcaaccgca ggctgccgca cgccctggtg gctgagtaca agacggttaa aggcagtagg 3480 gttgagtggc tggtcaataa agtaagaggg taccacgtcc tgctggtgag tgagtacaac 3540 ctggctttgc ctcgacacag ggtcacttgg ttgtcaccgc tgaatgtcac aggcgccgat 3600 aggtgctacg acctaagttt aggactgccg gctgacgccg gcaggttcga cttggtcttt 3660 gtgaacattc acacggaatt cagaatccac cactaccagc agtgtgtcga ccacgccatg 3720 aagctgcaga tgcttggggg agatgcgcta cgactgctaa aaacgggcgg catcttgatg 3780 agagcttacg gatacgccga taaaatcagc gaagccgttg tttcctcctt aagcagaaag 3840 ttctcgtctg caagagtgtt gcgcccggat tgtgtcacca gcaatacaga agtgttcttg 3900 ctgttctcca actttgacaa cggaaagaga ccctctacgc tacaccagat gaataccaag 3960 ctgagtgccg tgtatgccgg agaagccatg cacacggccg ggtgtgcacc atcctacaga 4020 gttaagagag cagacatagc cacgtgcaca gaagcggctg tggttaacgc agctaacgcc 4080 cgtggaactg taggggatgg cgtatgcagg gccgtggcga agaaatggcc gtcagccttt 4140 aagggagcag caacaccagt gggcacaatt aaaacagtca tgtgcggctc gtaccccgtc 4200 atccacgctg tagcgcctaa tttctctgcc acgactgaag cggaagggga ccgcgaattg 4260 gccgctgtct accgggcagt ggccgccgaa gtaaacagac tgtcactgag cagcgtagcc 4320 atcccgctgc tgtccacagg agtgttcagc ggcggaagag ataggctgca gcaatccctc 4380 aaccatctat tcacagcaat ggacgccacg gacgctgacg tgaccatcta ctgcagagac 4440 aaaagttggg agaagaaaat ccaggaagcc attgacatga ggacggctgt ggagttgctc 4500
aatgatgacg tggagctgac cacagacttg gtgagagtgc acccggacag cagcctggtg 4560 ggtcgtaagg gctacagtac cactgacggg tcgctgtact cgtactttga aggtacgaaa 4620 ttcaaccagg ctgctattga tatggcagag atactgacgt tgtggcccag actgcaagag 4680 gcaaacgaac agatatgcct atacgcgctg ggcgaaacaa tggacaacat cagatccaaa 4740 tgtccggtga acgattccga ttcatcaaca cctcccagga cagtgccctg cctgtgccgc 4800 tacgcaatga cagcagaacg gatcgcccgc cttaggtcac accaagttaa aagcatggtg 4860 gtttgctcat cttttcccct cccgaaatac catgtagatg gggtgcagaa ggtaaagtgc 4920 gagaaggttc tcctgttcga cccgacggta ccttcagtgg ttagtccgcg gaagtatgcc 4980 gcatctacga cggaccactc agatcggtcg ttacgagggt ttgacttgga ctggaccacc 5040 gactcgtctt ccactgccag cgataccatg tcgctaccca gtttgcagtc gtgtgacatc 5100 gactcgatct acgagccaat ggctcccata gtagtgacgg ctgacgtaca ccctgaaccc 5160 gcaggcatcg cggacctggc ggcagatgtg caccctgaac ccgcagacca tgtggacctc 5220 gagaacccga ttcctccacc gcgcccgaag agagctgcat accttgcctc ccgcgcggcg 5280 gagcgaccgg tgccggcgcc gagaaagccg acgcctgccc caaggactgc gtttaggaac 5340 aagctgcctt tgacgttcgg cgactttgac gagcacgagg tcgatgcgtt ggcctccggg 5400 attactttcg gagacttcga cgacgtcctg cgactaggcc gcgcgggtgc atatattttc 5460 tcctcggaca ctggcagcgg acatttacaa caaaaatccg ttaggcagca caatctccag 5520 tgcgcacaac tggatgcggt ccaggaggag aaaatgtacc cgccaaaatt ggatactgag 5580 agggagaagc tgttgctgct gaaaatgcag atgcacccat cggaggctaa taagagtcga 5640 taccagtctc gcaaagtgga gaacatgaaa gccacggtgg tggacaggct cacatcgggg 5700 gccagattgt acacgggagc ggacgtaggc cgcataccaa catacgcggt tcggtacccc 5760 cgccccgtgt actcccctac cgtgatcgaa agattctcaa gccccgatgt agcaatcgca 5820 gcgtgcaacg aatacctatc cagaaattac ccaacagtgg cgtcgtacca gataacagat 5880 gaatacgacg catacttgga catggttgac gggtcggata gttgcttgga cagagcgaca 5940 ttctgcccgg cgaagctccg gtgctacccg aaacatcatg cgtaccacca gccgactgta 6000 cgcagtgccg tcccgtcacc ctttcagaac acactacaga acgtgctagc ggccgccacc 6060 aagagaaact gcaacgtcac gcaaatgcga gaactaccca ccatggactc ggcagtgttc 6120 aacgtggagt gcttcaagcg ctatgcctgc tccggagaat attgggaaga atatgctaaa 6180 caacctatcc ggataaccac tgagaacatc actacctatg tgaccaaatt gaaaggcccg 6240 aaagctgctg ccttgttcgc taagacccac aacttggttc cgctgcagga ggttcccatg 6300
gacagattca cggtcgacat gaaacgagat gtcaaagtca ctccagggac gaaacacaca 6360 gaggaaagac ccaaagtcca ggtaattcaa gcagcggagc cattggcgac cgcttacctg 6420 tgcggcatcc acagggaatt agtaaggaga ctaaatgctg tgttacgccc taacgtgcac 6480 acattgtttg atatgtcggc cgaagacttt gacgcgatca tcgcctctca cttccaccca 6540 ggagacccgg ttctagagac ggacattgca tcattcgaca aaagccagga cgactccttg 6600 gctcttacag gtttaatgat cctcgaagat ctaggggtgg atcagtacct gctggacttg 6660 atcgaggcag cctttgggga aatatccagc tgtcacctac caactggcac gcgcttcaag 6720 ttcggagcta tgatgaaatc gggcatgttt ctgactttgt ttattaacac tgttttgaac 6780 atcaccatag caagcagggt actggagcag agactcactg actccgcctg tgcggccttc 6840 atcggcgacg acaacatcgt tcacggagtg atctccgaca agctgatggc ggagaggtgc 6900 gcgtcgtggg tcaacatgga ggtgaagatc attgacgctg tcatgggcga aaaaccccca 6960 tatttttgtg ggggattcat agtttttgac agcgtcacac agaccgcctg ccgtgtttca 7020 gacccactta agcgcctgtt caagttgggt aagccgctaa cagctgaaga caagcaggac 7080 gaagacaggc gacgagcact gagtgacgag gttagcaagt ggttccggac aggcttgggg 7140 gccgaactgg aggtggcact aacatctagg tatgaggtag agggctgcaa aagtatcctc 7200 atagccatgg ccaccttggc gagggacatt aaggcgttta agaaattgag aggacctgtt 7260 atacacctct acggcggtcc tagattggtg cgttaa 7296
<210> 3
<211> 840
<212> DNA
<213> Semliki Forest virus
<400> 3 attacatccc tacgcaaacg ttttacggcc gccggtggcg cccgcgcccg gcggcccgtc 60 cttggccgtt gcaggccact ccggtggctc ccgtcgtccc cgacttccag gcccagcaga 120 tgcagcaact catcagcgcc gtaaatgcgc tgacaatgag acagaacgca attgctcctg 180 ctaggcctcc caaaccaaag aagaagaaga caaccaaacc aaagccgaaa acgcagccca 240 agaagatcaa cggaaaaacg cagcagcaaa agaagaaaga caagcaagcc gacaagaaga 300 agaagaaacc cggaaaaaga gaaagaatgt gcatgaagat tgaaaatgac tgtatcttcg 360 tatgcggcta gccacagtaa cgtagtgttt ccagacatgt cgggcaccgc actatcatgg 420 gtgcagaaaa tctcgggtgg tctgggggcc ttcgcaatcg gcgctatcct ggtgctggtt 480 gtggtcactt gcattgggct ccgcagataa gttagggtag gcaatggcat tgatatagca 540 agaaaattga aaacagaaaa agttagggta agcaatggca tataaccata actgtataac 600
ttgtaacaaa gcgcaacaag acctgcgcaa ttggccccgt ggtccgcctc acggaaactc 660 ggggcaactc atattgacac attaattggc aataattgga agcttacata agcttaattc 720 gacgaataat tggattttta ttttattttg caattggttt ttaatatttc caaaaaaaaa 780 aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 840
<210> 4
<211> 503
<212> DNA
<213> Artificial Sequence
<220>
<223> Secuencia reguladora de la transcripción pO7Palb
<400> 4 tcgagtttac cactccctat cagtgataga gaaaagtgaa agtcgagttt accactccct 60 atcagtgata gagaaaagtg aaagtcgagt ttaccactcc ctatcagtga tagagaaaag 120 tgaaagtcga gtttaccact ccctatcagt gatagagaaa agtgaaagtc gagtttacca 180 ctccctatca gtgatagaga aaagtgaaag tcgagtttac cactccctat cagtgataga 240 gaaaagtgaa agtcgagttt accactccct atcagtgata gagaaaagtg aaagtcgagc 300 tcggtacaca gctccagatg gcaaacatac gcaagggatt tagtcaaaca actttttggc 360 aaagatggta tgattttgta atggggtagg aaccaatgaa atgcgaggta agtatggtta 420 atgatctaca gttattggtt aaagaagtat attagagcga gtctttctgc acacacgatc 480 acctttccta tcaaccccac tat 503
<210> 5
<211> 467
<212> DNA
<213> Artificial Sequence
<220>
<223> Variante de la secuencia reguladora de la transcripción pO7Palb
<400> 5 tcgagtttac cactccctat cagtgataga gaaaagtgaa agtcgagttt accactccct 60 atcagtgata gagaaaagtg aaagtcgagt ttaccactcc ctatcagtga tagagaaaag 120 tgaaagtcga gtttaccact ccctatcagt gatagagaaa agtgaaagtc gagtttacca 180 ctccctatca gtgatagaga aaagtgaaag tcgagtttac cactccctat cagtgataga 240 gaaaagtgaa agtcgagttt accactccct atcagtgata gagaaaagtg aaagtcgagc 300 tcggtacaca gctccagatg gcaaacatac gcaagggatt tagtcaaaca actttttggc 360 aaagatggta tgattttgta atggggtagg aaccaatgaa atgcgaggta agtatggtta 420 atgatctaca gttattggtt aaagaagtat attagagcga gtctttc
467
<210> 6
<211> 479
<212> DNA
<213> Artificial Sequence
<220>
<223> Variante de la secuencia reguladora de la transcripción pO7Palb
<400> 6 tcgagtttac cactccctat cagtgataga gaaaagtgaa agtcgagttt accactccct
atcagtgata gagaaaagtg aaagtcgagt ttaccactcc ctatcagtga tagagaaaag 120 tgaaagtcga gtttaccact ccctatcagt gatagagaaa agtgaaagtc gagtttacca 180 ctccctatca gtgatagaga aaagtgaaag tcgagtttac cactccctat cagtgataga 240 gaaaagtgaa agtcgagttt accactccct atcagtgata gagaaaagtg aaagtcgagc 300 tcggtacaca gctccagatg gcaaacatac gcaagggatt tagtcaaaca actttttggc 360 aaagatggta tgattttgta atggggtagg aaccaatgaa atgcgaggta agtatggtta 420 atgatctaca gttattggtt aaagaagtat attagagcga gtctttctgc acacacgat 479
<210> 7
<211> 480
<212> DNA
<213> Artificial Sequence
<220>
<223> Variante de la secuencia reguladora de la transcripción pO7Palb
<400> 7 tcgagtttac cactccctat cagtgataga gaaaagtgaa agtcgagttt accactccct 60 atcagtgata gagaaaagtg aaagtcgagt ttaccactcc ctatcagtga tagagaaaag 120 tgaaagtcga gtttaccact ccctatcagt gatagagaaa agtgaaagtc gagtttacca 180 ctccctatca gtgatagaga aaagtgaaag tcgagtttac cactccctat cagtgataga 240 gaaaagtgaa agtcgagttt accactccct atcagtgata gagaaaagtg aaagtcgagc 300 tcggtacaca gctccagatg gcaaacatac gcaagggatt tagtcaaaca actttttggc 360 aaagatggta tgattttgta atggggtagg aaccaatgaa atgcgaggta agtatggtta 420 atgatctaca gttattggtt aaagaagtat attagagcga gtctttctgc acacacgatc 480
<210> 8
<211> 481
<212> DNA
<213> Artificial Sequence
<220>
<223> Variante de la secuencia reguladora de la transcripción pO7Palb
<400> 8 tcgagtttac cactccctat cagtgataga gaaaagtgaa agtcgagttt accactccct
atcagtgata gagaaaagtg aaagtcgagt ttaccactcc ctatcagtga tagagaaaag 120
tgaaagtcga gtttaccact ccctatcagt gatagagaaa agtgaaagtc gagtttacca 180
ctccctatca gtgatagaga aaagtgaaag tcgagtttac cactccctat cagtgataga 240
gaaaagtgaa agtcgagttt accactccct atcagtgata gagaaaagtg aaagtcgagc 300
tcggtacaca gctccagatg gcaaacatac gcaagggatt tagtcaaaca actttttggc 360
aaagatggta tgattttgta atggggtagg aaccaatgaa atgcgaggta agtatggtta 420
atgatctaca gttattggtt aaagaagtat attagagcga gtctttctgc acacacgatc 480
a 481
<210> 9
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Secuencia LoxP1
<400> 9 taccgttcgt ataaagtatc ctatacgaag ttat 34
<210> 10
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Secuencia LoxP2
<400> 10 taccgttcgt ataatgtgta ctatacgaag ttat 34
<210> 11
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Secuencia LoxP3
<400> 11 ataacttcgt ataaagtatc ctatacgaac ggta
<210> 12
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Secuencia LoxP4
<400> 12 ataacttcgt ataatgtgta ctatacgaac ggta
<210> 13
<211> 381
<212> DNA
<213> Simian virus 40
<400> 13 cgcccttccc aacagttgcg cagcctgaat ggcgaatgga gatccaattt ttaagtgtat 60 aatgtgttaa actactgatt ctaattgttt gtgtatttta gattcacagt cccaaggctc 120 atttcaggcc cctcagtcct cacagtctgt tcatgatcat aatcagccat accacatttg 180 tagaggtttt acttgcttta aaaaacctcc cacacctccc cctgaacctg aaacataaaa 240 tgaatgcaat tgttgttgtt aacttgttta ttgcagctta taatggttac aaataaagca 300 atagcatcac aaatttcaca aataaagcat ttttttcact gcattctagt tgtggtttgt 360 ccaaactcat caatgtatct t 381
<210> 14
<211> 317
<212> DNA
<213> Simian virus 40
<400> 14 ggtgtggaaa gtccccaggc tccccagcag gcagaagtat gcaaagcatg catctcaatt 60 agtcagcaac caggtgtgga aagtccccag gctccccagc aggcagaagt atgcaaagca 120 tgcatctcaa ttagtcagca accatagtcc cgcccctaac tccgcccatc ccgcccctaa 180 ctccgcccag ttccgcccat tctccgcccc atggctgact aatttttttt atttatgcag 240 aggccgaggc cgcctcggcc tctgagctat tccagaagta gtgaggaggc ttttttggag 300 gcctaggctt ttgcaaa 317
<210> 15
<211> 13
<212> PRT
<213> herpes simplex virus 7
<400> 15
Pro Ala Asp Ala Leu Asp Asp Phe Asp Leu Asp Met Leu 1 5 10
<210> 16
<211> 747
<212> DNA
<213> Artificial Sequence
<220>
<223> Secuencia que codifica el activador transcripcional rtTA2sM2
<400> 16 atgtctagac tggacaagag caaagtcata aacggcgctc tggaattact caatggagtc
ggtatcgaag gcctgacgac aaggaaactc gctcaaaagc tgggagttga gcagcctacc 120 ctgtactggc acgtgaagaa caagcgggcc ctgctcgatg ccctgccaat cgagatgctg 180 gacaggcatc atacccactt ctgccccctg gaaggcgagt catggcaaga ctttctgcgg 240 aacaacgcca agtcattccg ctgtgctctc ctctcacatc gcgacggggc taaagtgcat 300 ctcggcaccc gcccaacaga gaaacagtac gaaaccctgg aaaatcagct cgcgttcctg 360 tgtcagcaag gcttctccct ggagaacgca ctgtacgctc tgtccgccgt gggccacttt 420 acactgggct gcgtattgga ggaacaggag catcaagtag caaaagagga aagagagaca 480 cctaccaccg attctatgcc cccacttctg agacaagcaa ttgagctgtt cgaccggcag 540 ggagccgaac ctgccttcct tttcggcctg gaactaatca tatgtggcct ggagaaacag 600 ctaaagtgcg aaagcggcgg gccggccgac gcccttgacg attttgactt agacatgctc 660 ccagccgatg cccttgacga ctttgacctt gatatgctgc ctgctgacgc tcttgacgat 720 tttgaccttg acatgctccc cgggtaa 747
<210> 17
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el gen que confiere resistencia a higromicina
<400> 17 cccgggatga aaaagcctga actca 25
<210> 18
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el gen que confiere resistencia a higromicina
<400> 18 gcggccgcct attcctttgc cctcggac
<210> 19
<211> <212> <213> 23 DNA Artificial Sequence
<220> <223> Oligonucleótido sentido para amplificar el vector pSFV-1
<400> 19 atggcggatg tgtgacatac acg 23
<210> <211> <212> <213> 20 47 DNA Artificial Sequence
<220> <223> Oligonucleótido antisentido para amplificar el vector pSFV-1
<400> 20 gctcctcgcc cttgctcacc atcgtgggtg gttaatctct cgcgtag 47
<210> <211> <212> <213> 21 20 DNA Artificial Sequence
<220> <223> Oligonucleótido sentido para amplificar el vector pCMV-GFP
<400> 21 atggtgagca agggcgagga 20
<210> <211> <212> <213> 22 30 DNA Artificial Sequence
<220> <223> Oligonucleótido antisentido para amplificar el vector pCMV-GFP
<400> 22 actagtctta agatacattg atgagtttgg 30
<210> <211> <212> <213> 23 39 DNA Artificial Sequence
<220> <223> Oligonucleótido sentido para amplificar el vectorAAV8-Tetbidir-Alb-CMVm-luc
<400> 23 atgcatcgcg atagcggtac cgagctctta cgctcgagt 39
<210> 24
<211> 56
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el vector AAV8-Tetbidir-Alb-CMVm-luc
<400> 24 ggtggcagga gctggaacaa aatcttttgg cgtcgtgtat gtcacacatc cgccat
<210> 25
<211> 82
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el vector AAV8-Tetbidir-Alb-CMVm-luc
<400> 25 ggtggcagga gctggaacaa aatcttttgg cgtcgtgtat gtcacacatc cgccatgatc 60
gtgtgtgcag aaagactcgc tc 82
<210> 26
<211> 83
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el vector AAV8-Tetbidir-Alb-CMVm-luc
<400> 26 ggtggcagga gctggaacaa aatcttttgg cgtcgtgtat gtcacacatc cgccatatgt 60
cgtgtgtgca gaaagactcg ctc 83
<210> 27
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el vector pSFV-1
<400> 27 agagctcggc cgcctcggcc tctga
<210> 28
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el vector pSFV-1
<400> 28 atgcatggcg gtaatacggt ta
<210> 29
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el vector pSFV-1
<400> 29 atggcggatg tgtgacatac acg 23
<210> 30
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el vector pSFV-1
<400> 30 gggcccgata tccaagatga gtgtgtc 27
<210> 31
<211> 62
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el vector pSFV-1
<400> 31 actagtataa cttcgtatag tacacattat acgaacggta gtgggcgaag aactccagca 60
tg 62
<210> 32
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el gen de GFP
<400> 32 ggatccatgg tgagcaaggg cgaggagc
<210> 33
<211> 43
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el gen de GFP
<400> 33 cccgggatct taattaatta cttgtacagc tcgtccatgc cga
<210> 34
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para generar el sitio LoxP1
<400> 34 gatctaccgt tcgtataaag tatcctatac gaagttatc 39
<210> 35
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para generar el sitio LoxP1
<400> 35 gatcgataac ttcgtatagg atactttata cgaacggta 39
<210> 36
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para generar un fragmento con las secuencias LoxP1-GFP-SV40PolyA-SV40Prom-neo-LoxP2-TK-PolyA
<400> 36 catatgtacc gttcgtataa agtatcct 28
<210> 37
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para generar un fragmento con las secuencias LoxP1-GFP-SV40PolyA-SV40Prom-neo-LoxP2-TK-PolyA
<400> 37 actagtgcta tggcagggcc tgccgccccg
<210> 38
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el promotor EF1a
<400> 38 gatatcgtga ggctccggtg cccgtcag
<210> 39
<211> 44
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el promotor EF1a
<400> 39 gcggccgctt cacgacacct gaaatggaag aaaaaaactt tgaa 44
<210> 40
<211> 62
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para generar la secuencia LoxP3 seguida de un sitio de clonación múltiple
<400> 40 tataacttcg tataaagtat cctatacgaa cggtatctag atctcgcgag ctcagccata 60
tg 62
<210> 41
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para generar la secuencia LoxP3 seguida de un sitio de clonación múltiple
<400> 41 tatggctgag ctcgcgagat ctagataccg ttcgtatagg atactttata cgaagttat 59
<210> 42
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar la señal de poliadenilación de SV40
<400> 42 catatgttag ggtaggcaat ggcattga
<210> 43
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar la señal depoliadenilación de SV40
<400> 43
cagctgctgg cttaactatg cggcatc 27
<210> 44
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para generar el sitio LoxP4
<400> 44
catgggatca taacttcgta taatgtgtac tatacgaacg gccatg 46
<210> 45
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para generar el sitio LoxP4
<400> 45
gatccatggc cgttcgtata gtacacatta tacgaagtta tgatcc 46
<210> 46
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el gen Tomato
<400> 46
agatctgcac catggtgagc aagggcgagg a 31
<210> 47
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el gen Tomato
<400> 47
catatgttac ttgtacagct cgtccatg 28
<210> 48
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el gen humano de cardiotrofina-1
<400> 48 agatctgcac catgtcggcc ctgctgatcc tgg
<210> 49
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el gen humano de cardiotrofina-1
<400> 49 catatgtcag gccgagcccc cgggcag 27
<210> 50
<211> 101
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para amplificar el gen humano de oncostatina M
<400> 50 ggatcctcgc gagcaccatg tcggccctgc tgatcctggc cctggtcgga gccgccgtcg 60
cccaccacca ccaccaccac gcggctatag gcagctgctc g 101
<210> 51
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el gen humano de oncostatina M
<400> 51 ggatccatat gctatctccg gctccggttc gggc
<210> 52
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para detectar ARN de SFV que codifican GFP
<400> 52 cctaggggga cattaaggcg tttaag
<210> 53
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para detectar ARN de SFV que codifican GFP
<400> 53 gtcctccttg aagtcgatgc 20
<210> 54
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el gen humano de cardiotrofina 1
<400> 54 cgacatggtg cagatctaga 20
<210> 55
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para detectar la la secuencia de TK-PolyA
<400> 55 ccgtgtttca gttagcctcc ccc 23
<210> 56
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para detectar el intercambio de neomicina y purimicina
<400> 56 tgctcctgcc gagaaagtat
<210> 57
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para detectar el intercambio de neomicina y purimicina
<400> 57 aagcttccgc cacgaccggt gccg
<210> 58
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para detectar la replicasa
<400> 58 ctgttctcga cgcgtcgtc 19
<210> 59
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para detectar la replicasa
<400> 59 gaggtgtttc cacgaccc 18
<210> 60
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido sentido para detectar actina
<400> 60 gggatgtttg ctccaaccaa 20
<210> 61
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para detectar actina
<400> 61 gcgcttttga ctcaaggatt taa
<210> 62
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el ADNc en el análisis del sitio de iniciación de la transcripción a partir del promotor mínimo de albúmina
<400> 62 tgcttgtcgg ccatgatata
<210> 63
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el ADNc en el análisis del sitio de iniciación de la transcripción a partir del promotor mínimo de albúmina
<400> 63 gaacttcagg gtcagcttgc 20
<210> 64
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Oligonucleótido antisentido para amplificar el ADNc en el análisis del sitio de iniciación de la transcripción a partir del promotor mínimo de albúmina
<400> 64 aacttgtggc cgtttacgtc 20
<210> 65
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Olignucleótido sentido para insertar un promotor subgenómico entre los genes de las cadena pesada y ligera del anticuerpo A20
<400> 65 agatctcacc atgaagttgc ctgttaggct g 31
<210> 66
<211> 48
<212> DNA
<213> Artificial Sequence
<220>
<223> Olignucleótido antisentido para insertar un promotor subgenómico entre los genes de las cadena pesada y ligera del anticuerpo A20
<400> 66
ccaatctagg accgccgtag aggtttaaca ctcattcctg ttgaagct
<210> 67
<211> 78
<212> DNA
<213> Artificial Sequence
<220>
<223> Olignucleótido sentido para insertar un promotor subgenómico entre los genes de las cadena pesada y ligera del anticuerpo A20
<400> 67 acctctacgg cggtcctaga ttggtgcgtt aatacacaga attctgattg caccatggca 60
tggaacttca tcatggtc 78
<210> 68
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Olignucleótido antisentido para insertar un promotor subgenómico entre los genes de las cadena pesada y ligera del anticuerpo A20
<400> 68 catatgctat ttacccggag tccgggag 28
<210> 69
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Secuencia LoxP salvaje
<400> 69 ataacttcgt ataatgtatg ctatacgaag ttat 34
<210> 70
<211> 271
<212> DNA
<213> herpes simplex virus 7
<400> 70 gggggaggct aactgaaaca cggaaggaga caataccgga aggaacccgc gctatgacgg
caataaaaag acagaataaa acgcacggtg ttgggtcgtt tgttcataaa cgcggggttc 120
ggtcccaggg ctggcactct gtcgataccc caccgagacc ccattggggc caatacgccc 180
gcgtttcttc cttttcccca ccccaccccc caagttcggg tgaaggccca gggctcgcag 240
ccaacgtcgg ggcggcaggc cctgccatag c
271
Patentes similares o relacionadas:
Método y medios para purificar vectores retrovíricos, del 29 de Julio de 2020, de Autolus Limited: Una célula productora de retrovirus que expresa una proteína de marcaje en la superficie celular, de tal manera que los vectores retrovíricos producidos por la célula se […]
 CD52 soluble para su uso en el tratamiento o la prevención de la esclerosis múltiple o de la artritis reumatoide, del 29 de Julio de 2020, de THE WALTER AND ELIZA HALL INSTITUTE OF MEDICAL RESEARCH: Uno cualquiera o más de:
i) glucoproteína CD52 soluble, ii) una proteína de fusión que comprende la glucoproteína CD52 soluble como una primera proteína, y una segunda proteína;
[…]
CD52 soluble para su uso en el tratamiento o la prevención de la esclerosis múltiple o de la artritis reumatoide, del 29 de Julio de 2020, de THE WALTER AND ELIZA HALL INSTITUTE OF MEDICAL RESEARCH: Uno cualquiera o más de:
i) glucoproteína CD52 soluble, ii) una proteína de fusión que comprende la glucoproteína CD52 soluble como una primera proteína, y una segunda proteína;
[…]
Arenavirus trisegmentados como vectores de vacunas, del 22 de Julio de 2020, de UNIVERSITE DE GENEVE: Una partícula de arenavirus trisegmentada infecciosa y competente para la replicación que comprende un segmento L y dos segmentos S, en donde uno de los dos segmentos […]
Promotor híbrido y usos del mismo, del 15 de Julio de 2020, de Just Biotherapeutics, Inc: Un promotor híbrido, que comprende: (i) una secuencia potenciadora de citomegalovirus murino (mCMV), que comprende un elemento potenciador de mCMV (mCMV-E) y una […]
Señal para el empaquetamiento de vectores del virus de la gripe, del 24 de Junio de 2020, de WISCONSIN ALUMNI RESEARCH FOUNDATION: Un vector del virus de la gripe para la expresión y empaquetamiento de ARNv recombinante, en el que el vector comprende: secuencias correspondientes […]
Métodos y composiciones para la producción de virus vaccina, del 10 de Junio de 2020, de SillaJen Biotherapeutics, Inc: Un metodo para producir un virus vaccinia de la cepa Wyeth o Western Reserve que comprende: (a) infectar las celulas HeLa adheridas a una superficie […]
Vectores de AAV dirigidos a oligodendrocitos, del 10 de Junio de 2020, de THE UNIVERSITY OF NORTH CAROLINA AT CHAPEL HILL: Un ácido nucleico que codifica una cápside de AAV, comprendiendo el ácido nucleico una secuencia codificante de la cápside de AAV que es al menos el 96 % idéntica […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]