Composiciones y métodos para potenciar respuestas inmunitarias.
Un vector de vacuna bacteriana que comprende una secuencia polinucleotídica que codifica un polipéptido antigénico y una secuencia polinucleotídica de CD154 que codifica un polipéptido de CD154 capaz de unirse con CD40,
en donde el polipéptido de CD154 tiene menos de aproximadamente 50 aminoácidos y comprende los aminoácidos 140-149 de SEC ID Nº: 26 o un homólogo de los mismos que es capaz de unirse con CD40, en donde el polipéptido antigénico es un polipéptido de la gripe, en donde la secuencia polinucleotídica que codifica el polipéptido antigénico y la secuencia polinucleotídica de CD154 se insertan dentro de una secuencia que codifica una parte externa de una proteína transmembrana y en donde el vector de vacuna potencia la respuesta inmunitaria de un sujeto.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2007/078785.
Solicitante: THE BOARD OF TRUSTEES OF THE UNIVERSITY OF ARKANSAS.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 2404 N. University Avenue Little Rock, AR 72207 ESTADOS UNIDOS DE AMERICA.
Inventor/es: KWON YOUNG-MIN, BOTTJE,WALTER, HARGIS,BILLY, BERGHMAN,LUC, COLE,KIMBERLY, COX,MANDY, LAYTON,SHERRYLL.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N1/21 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 1/00 Microorganismos, p.ej. protozoos; Composiciones que los contienen (preparaciones de uso médico que contienen material de protozoos, bacterias o virus A61K 35/66, de algas A61K 36/02, de hongos A61K 36/06; preparación de composiciones de uso médico que contienen antígenos o anticuerpos bacterianos, p. ej. vacunas bacterianas, A61K 39/00 ); Procesos de cultivo o conservación de microorganismos, o de composiciones que los contienen; Procesos de preparación o aislamiento de una composición que contiene un microorganismo; Sus medios de cultivo. › modificados por la introducción de material genético extraño.
PDF original: ES-2520026_T3.pdf




Fragmento de la descripción:
Composiciones y métodos para potenciar respuestas inmunitarias Solicitudes relacionadas La presente solicitud reivindica el beneficio de prioridad de la Solicitud Provisional de Estados Unidos Nº 60/825.983, presentada el 18 de septiembre de 2006.
Declaración con respecto a la investigación patrocinada por el gobierno federal
La presente invención se realizó con el apoyo del gobierno de los Estados Unidos otorgado por la subvención de los Institutos Nacionales de la Salud R21 AI063137. Los Estados Unidos pueden tener ciertos derechos en la presente invención.
Introducción La infección por virus de la gripe, particularmente gripe aviar H5N1, constituye una preocupación sanitaria y económica creciente. Las pruebas indican claramente que H5N1 continua circulando entre aves y cerdos susceptibles en regiones crecientes del mundo. Muchos científicos creen que si permanece incontrolada, la gripe aviar H5N1 actual mutará hasta permitir la transmisión entre seres humanos y provocar una pandemia global. Con una tasa de mortalidad de más del 50 %, dicho brote sería devastador. Independientemente de la capacidad del virus para provocar enfermedad humana, la gripe aviar H5N1 ya amenaza tener una repercusión económica enorme debido a la erradicación de poblaciones de aves de corral en áreas afectadas. Por lo tanto, es necesario el desarrollo de una vacuna para proteger a seres humanos, aves de corral, cerdos y otros animales domesticados de la gripe H5N1. Una vacuna de la gripe que es capaz de proteger contra H5N1 así como otros virus de la gripe sería óptima.
VEGA MARIO I ET AL., IMMUNOLOGY, vol. 110, nº 2, octubre de 2003, páginas 206-216 describe una proteína de fusión OmpC de Salmonella typhi que expresa la cadena de aminoácidos de CD154 Trp140-Ser149. Los autores indican que la proteína de fusión se une con CD40 y activa una línea de linfocitos B de linfoma.
El documento WO2006/1105972 describe un organismo transgénico que expresa un ácido nucleico que codifica CD40L, y usos del mismo como medicamento o como una vacuna. También se desvelan una composición farmacéutica o vacuna que comprende dicho organismo transgénico, métodos para tratar a un sujeto infectado con dicho organismo, y métodos para estimular una respuesta inmunitaria contra una enfermedad o enfermedades provocadas por dicho organismo en un sujeto que lo necesite.
Sumario Se desvelan cepas 13A de Salmonella enteritidis que tienen los números de depósito de ATCC PTA-7871, PTA7872 o PTA-7873. También se desvela una composición que comprende una cepa de Salmonella atenuada y un vehículo farmacéuticamente aceptable.
En otro aspecto, se proporcionan métodos para potenciar una respuesta inmunitaria en un sujeto administrando un vector de vacuna al sujeto. Un polinucleótido que codifica un polipéptido de CD 154 capaz de unirse con CD40, teniendo el polipéptido menos de 50 aminoácidos y comprendiendo los aminoácidos 140-149 de SEC ID Nº : 26 o un homólogo del mismo. El vector de vacuna se administra al sujeto en una cantidad eficaz para potenciar la respuesta inmunitaria del sujeto a la vacuna.
En un aspecto adicional, se proporcionan métodos para potenciar la respuesta inmunitaria contra la gripe A en un sujeto administrando al sujeto una bacteria que comprende un polinucleótido que codifica un polipéptido de proteína M2e de gripe A en una cantidad eficaz para potenciar la respuesta inmunitaria del sujeto a la gripe A.
En otro aspecto más, se proporcionan métodos para reducir la morbilidad asociada con la infección por gripe A en un sujeto administrando al sujeto una bacteria que comprende un polinucleótido que codifica un polipéptido de proteína M2e de gripe A en una cantidad eficaz para reducir la morbilidad asociada con una infección posterior con gripe A.
En otro aspecto más, se proporcionan métodos para generar mutaciones específicas de sitio en una bacteria. Se genera un primer polinucleótido que comprende un marcador de contraselección y un marcador de resistencia a antibióticos flanqueado por polinucleótidos homólogos de las secuencias que flanquean un sitio de mutación en el cromosoma de la bacteria. El primer polinucleótido se introduce después en la bacteria y después de recombinación homóloga y selección de antibióticos se aísla un intermedio. Se genera un segundo polinucleótido que comprende la mutación flanqueada por polinucleótidos homólogos de secuencias que flanquean el sitio de mutación. El segundo polinucleótido se introduce después en el intermedio y el mutante específico de sitio se aísla por contraselección con
respecto a pérdida del marcador de contraselección.
En un aspecto adicional más, se proporcionan métodos para desarrollar vectores de vacuna bacteriana. Se selecciona una bacteria capaz de colonizar un sujeto. La bacteria se atenúa y se incorpora un polinucleótido que comprende una secuencia que codifica un polipéptido de CD 154 capaz de unirse con CD40 en la bacteria.
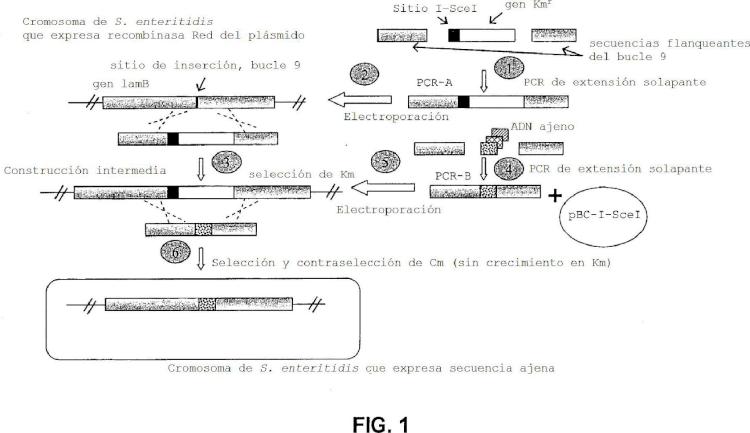
Breve descripción de los dibujos La Figura 1 representa el esquema para realizar mutaciones dirigidas en Salmonella enteritidis. La Figura 2 representa el esquema de diseño del método de PCR de extensión solapante usado para generar las inserciones de M2e y M2e-CD154 en el bucle 9 del polinucleótido lamB. La Figura 3 es un gráfico de barras que muestra la cantidad relativa de anticuerpo en suero generado en los puntos temporales indicados en respuesta a la administración del tratamiento indicado. La Figura 4 es un gráfico de líneas que muestra la cantidad de anticuerpo en suero a lo largo del tiempo después de la administración de los tratamientos indicados. La Figura 5 es un gráfico que muestra la morbilidad de pollos después de vacunación con SE HM el día de la eclosión, refuerzo el día 21 e infección de exposición con una gripe A de baja patogenicidad a los 32 días después de la eclosión. La Figura 6 es un gráfico que muestra difusión viral los días 2 y 4 después de la exposición a una gripe A de baja patogenicidad después de la vacunación con SE HM el día de la eclosión, refuerzo el día 21 e infección de exposición el día 32 después de la eclosión. La Figura 7 es un gráfico que muestra la morbilidad de pollos después de vacunación con SE HM el día de la eclosión, refuerzo el día 21 e infección de exposición con una gripe A de alta patogenicidad el día 32 después de la eclosión. La Figura 8 es un gráfico que muestra difusión viral los días 2 y 4 después de la exposición a una gripe A de alta patogenicidad después de la vacunación con SE HM el día de la eclosión, refuerzo el día 21 e infección de exposición el día 32 después de la eclosión.
Descripción detallada Las tecnologías de ADN recombinante permiten una manipulación relativamente fácil de muchas especies bacterianas y virales. Algunas bacterias y virus son levemente patógenos o no patógenos, pero son capaces de generar una respuesta inmunitaria robusta. Estas bacterias y virus hacen atractivos los vectores de vacunas para inducir una respuesta inmunitaria a un antígeno heterólogo o ajeno. Los vectores de vacuna bacteriana o viral pueden imitar la infección natural y producen inmunidad robusta y de larga duración. Los vectores de vacunas son con frecuencia relativamente económicos de producir y administrar. Además, dichos vectores pueden con frecuencia portar más de un antígeno y pueden proporcionar protección contra múltiples agentes infecciosos.
En un aspecto, la presente divulgación se refiere al uso de vectores de Salmonela en la vacunación y generación de respuestas inmunitarias contra Salmonela y otros agentes patógenos. Las cepas de Salmonela son vectores de vacuna adecuados debido a la capacidad de hacer a las bacterias capaces de expresar polipéptidos heterólogos. Además, los genes bacterianos pueden mutarse o atenuarse para crear bacterias con de baja a ninguna patogénesis para el sujeto infectado o inmunizado, manteniendo al mismo tiempo la inmunogenicidad.
La capacidad de la Salmonela para sobrevivir al tracto gastrointestinal del hospedador y dar lugar a una respuesta inmunitaria mucosa está documentada. Las vacunas orales que usan un vector de Salmonela producen una respuesta inmunitaria mucosa robusta y son relativamente fáciles de administrar tanto a animales como a seres humanos. Muchas de las cepas de vacuna de Salmonela actuales no son tan eficaces en la generación de una respuesta inmunitaria protectora fuerte en comparación con sus homólogos más virulentos. Una cepa de Salmonela que pudiera usarse para vacunación mucosa, por ejemplo, oral, eficaz proporcionaría un vector que podría usarse para vacunar fácilmente a un sujeto contra uno o más patógenos, tales como gripe... [Seguir leyendo]
Reivindicaciones:
1. Un vector de vacuna bacteriana que comprende una secuencia polinucleotídica que codifica un polipéptido antigénico y una secuencia polinucleotídica de CD154 que codifica un polipéptido de CD154 capaz de unirse con 5 CD40, en donde el polipéptido de CD154 tiene menos de aproximadamente 50 aminoácidos y comprende los aminoácido.
14. 149 de SEC ID Nº : 26 o un homólogo de los mismos que es capaz de unirse con CD40, en donde el polipéptido antigénico es un polipéptido de la gripe, en donde la secuencia polinucleotídica que codifica el polipéptido antigénico y la secuencia polinucleotídica de CD154 se insertan dentro de una secuencia que codifica una parte externa de una proteína transmembrana y en donde el vector de vacuna potencia la respuesta inmunitaria de un sujeto.
2. El vector de vacuna de la reivindicación 1, en el que la secuencia polinucleotídica que codifica un polipéptido antigénico es una polinucleótido M2e de la gripe que codifica un polipéptido M2e de la gripe.
3. El vector de vacuna de las reivindicaciones 1 o 2, en el que el polipéptido antigénico y el polipéptido de CD154 están codificados por la misma secuencia polinucleotídica.
4. Uso de una secuencia polinucleotídica de CD154 que codifica un polipéptido de CD154 capaz de unirse con un CD40 para la preparación de un vector de vacuna bacteriano para potenciar la respuesta inmunitaria de un sujeto a un polipéptido de la gripe, en donde el polipéptido de CD154 tiene menos de aproximadamente 50 aminoácidos y comprende los aminoácido.
14. 149 de SEC ID Nº : 26 o un homólogo de los mismos capaz de unirse con CD40, en donde la secuencia polinucleotídica que codifica el polipéptido de la gripe y la secuencia polinucleotídica de CD154 se insertan dentro de una secuencia que codifica una parte externa de una proteína transmembrana.
6. El uso de cualquiera de las reivindicaciones 4-5, en el que el polipéptido de CD154 se expresa en la superficie del vector de vacuna.
8. El uso de una cualquiera de las reivindicaciones 4-7, en el que el polipéptido de la gripe y el polipéptido de CD154 35 están codificados por la misma secuencia polinucleotídica.
9. Uso de una bacteria que comprende al menos una secuencia polinucleotídica de M2e de la Gripe A que codifica una polipéptido M2e de la Gripe A para la preparación de una vacuna eficaz para potenciar la respuesta inmunitaria de un sujeto a la Gripe A, en donde la bacteria comprende además una secuencia polinucleotídica de CD154 que codifica un polipéptido de CD154 capaz de unirse con CD40 y que tiene menos de 50 aminoácidos y comprende los aminoácido.
14. 149 de SEC ID Nº : 26 o un homólogo de los mismos capaz de unirse con CD40, y en donde la secuencia polinucleotídica de M2e de Gripe A y la secuencia polinucleotídica de CD154 se insertan en una secuencia polinucleotídica que codifica una parte externa de una proteína transmembrana.
10. Uso de una bacteria que comprende al menos una secuencia polinucleotídica de M2e de la Gripe A que codifica un polipéptido M2e de la Gripe A para la preparación de una vacuna eficaz para reducir la morbilidad relacionada con la Gripe A, en donde la bacteria comprende además una secuencia polinucleotídica de CD154 que codifica un polipéptido de CD154 capaz de unirse con CD40 y que tiene menos de 50 aminoácidos y comprende los aminoácido.
14. 149 de SEC ID Nº : 26 o un homólogo de los mismos capaz de unirse con CD40 y en donde la 50 secuencia polinucleotídica de M2e de la Gripe A y la secuencia polinucleotídica de CD154 se insertan en una secuencia polinucleotídica que codifica una parte externa de una proteína transmembrana.
11. El uso de cualquiera de las reivindicaciones 8-10, en el que el polipéptido M2e de la Gripe A se selecciona del grupo que consiste en SEC ID Nº : 1; SEC ID Nº : 2; SEC ID Nº : 3; SEC ID Nº : 4; SEC ID Nº : 5; SEC ID Nº : 20 y un 55 fragmento inmunogénico de SEC ID Nº : 1, un fragmento inmunogénico de SEC ID Nº : 2, un fragmento inmunogénico de SEC ID Nº : 3 y un fragmento inmunogénico de SEC ID Nº : 4.
12. El uso de cualquiera de las reivindicaciones 7-11, en el que la bacteria es un miembro de la familia Enterobacteriaceae. 60
13. El uso de la reivindicación 12, en el que la bacteria es una cepa de Salmonela de cualquiera de (a) Salmonella enteritidis que tiene el número de depósito de la ATCC PTA-7871, (b) una cepa de Salmonela capaz de colonizar un sujeto, (c) una cepa de Salmonela que comprende una mutación en una ruta de aromatización, (d) una cepa de Salmonela que comprende una mutación dentro de aroA, (e) una cepa de Salmonela que comprende una mutación 65 en una ruta de respuesta a estrés y (f) una cepa de Salmonela que comprende una mutación en htrA.
14. El uso de cualquiera de las reivindicaciones 9-13, en el que el polipéptido de CD154 y el polipéptido M2e de la Gripe están codificados por la misma secuencia polinucleotídica.
15. Un método para desarrollar un vector de vacuna bacteriana que comprende:
a) seleccionar una bacteria capaz de colonizar un sujeto; b) atenuar la bacteria para generar una bacteria atenuada; c) incorporar una secuencia polinucleotídica de CD154 que codifica un polipéptido de CD154 capaz de unirse con CD40 en la bacteria atenuada para generar un vector de vacuna, en donde el polipéptido de CD154 tiene menos de 50 aminoácidos y comprende los aminoácido.
14. 149 de SEC ID Nº : 26 o un homólogo de los mismos capaz de unirse con CD40; y d) incorporar una segunda secuencia polinucleotídica que codifica un polipéptido de la Gripe en el cromosoma del vector de vacuna, en donde las secuencias polinucleotídicas que codifican el polipéptido de la Gripe y la secuencia polinucleotídica de CD154 se insertan dentro de una secuencia que codifica una parte externa de una proteína transmembrana.
16. El método de la reivindicación 15, en el que el polipéptido de CD154 y el polipéptido antigénico están codificados por la misma secuencia polinucleotídica.
Patentes similares o relacionadas:
Xilanasa mutante, método de fabricación y uso de la misma, y método para fabricar lignocelulosa sacarificada, del 29 de Julio de 2020, de MITSUI CHEMICALS, INC.: Una xilanasa mutante que comprende la secuencia de aminoácidos de la SEQ ID NO: 2 con una sustitución del resto de aminoácido en la posición 154 en la cual el resto de lisina […]
Anticuerpo anti-FGF23 y composición farmacéutica que comprende el mismo, del 15 de Julio de 2020, de Kyowa Kirin Co., Ltd: Anticuerpo o fragmento funcional del mismo que se une a la totalidad o a una parte del epítopo de FGF23 humano, al que se une un anticuerpo producido […]
Biblioteca de péptidos y su uso, del 8 de Julio de 2020, de DAIICHI SANKYO COMPANY, LIMITED: Una biblioteca de péptidos que comprende una pluralidad de péptidos diferentes en la que los péptidos comprenden cada uno una secuencia de aminoácidos […]
Microorganismo del género Corynebacterium de producción de lisina y procedimiento de producción de lisina usando el mismo, del 10 de Junio de 2020, de CJ CHEILJEDANG CORPORATION: Un microorganismo del género Corynebacterium productor de L-lisina, en el que la vía de biosíntesis de L-lisina está potenciada y la oxalacetato-descarboxilasa […]
Métodos y organismos con mayores eficiencias del flujo de carbono, del 13 de Mayo de 2020, de Genomatica, Inc: Un organismo microbiano no natural que comprende una modificación genética de la atenuación de cydA o cydB, y una o más de una modificación genética que aumenta la expresión […]
Métodos para la expresión recombinante del gen de la beta-glucosidasa, del 29 de Abril de 2020, de Wilmar (shanghai) Biotechnology Research & Development Center Co., Ltd: Una proteína de fusión, en donde dicha proteína de fusión comprende: (a) una proteasa aspártica o un fragmento soluble de la misma, en donde dicho fragmento soluble […]
Células huéspedes modificadas y usos de las mismas, del 22 de Abril de 2020, de GLAXOSMITHKLINE BIOLOGICALS S.A.: Una célula huésped que comprende: i. un ácido nucleico que codifica una glicosiltransferasa derivada de un racimo rfb de Pseudomonas; ii. un ácido nucleico […]
Fábrica de células bacterianas modificadas genéticamente para la producción de tiamina, del 22 de Abril de 2020, de Biosyntia ApS: Bacteria modificada genéticamente para la producción de tiamina no fosforilada; en la que dicha bacteria se caracteriza por tener transgenes […]