Método de determinación de tropismo del VIH-1 y kit asociado.
Método de determinación de tropismo del VIH-1 y kit asociado.
La presente invención se refiere a un método de determinación del tropismo del virus de la inmunodeficiencia humana 1 (VIH-1) que comprende la amplificación por PCR del gen env. Además,
la presente invención se refiere un kit para llevar a cabo dicho método y al uso de dicho kit para la determinación del tropismo del VIH-1.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201331182.
Solicitante: UNIVERSIDAD DE JAEN.
Nacionalidad solicitante: España.
Inventor/es: CARUZ ARCOS,Antonio José.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/04 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › Determinación de la presencia o del tipo de microorganismo; Empleo de medios selectivos para la investigación o análisis de antibióticos o bactericidas; Composiciones para este fin que contienen un indicador químico.
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
MÉTODO DE DETERMINACIÓN DE TROPISMO DEL VIH-1 Y KIT ASOCIADO
La presente invención se refiere a un método de determinación del tropismo del virus de la inmunodeficiencia humana 1 (VIH-1) que comprende la amplificación por PCR del gen env. Además, la presente invención se refiere un kit para llevar a cabo dicho método y al uso de dicho kit para la determinación del tropismo del VIH-1.
ESTADO DE LA TECNICA ANTERIOR
El virus de inmunodeficiencia humana 1 (VIH-1) es un lentivirus de la familia Retroviridae que infecta células del sistema immune. El virus interacciona con la célula diana a través del producto del gen de la envuelta (env) , que corresponde a la proteína de envuelta gp120/gp41. Este proceso requiere la unión al receptor CD4 seguido de un cambio conformacional que permite la interacción de un segundo correceptor que puede ser CCR5, o CXCR4. Según cuál sea el correceptor implicado en la interacción, el virus presentará un tropismo viral u otro.
Inicialmente el tropismo viral se clasificaba en cepas inductoras de sincitios (SI) y cepas no inductoras de sincitios (NSI) , en función de si el virus causaba dicho efecto citopático. En el caso del VIH, un sincitio es una célula multinucleada consecuencia de la fusión de células individuales. Se ha demostrado que la diferenciación entre cepas SI y NSI es debida al uso diferencial de correceptores: las cepas SI presentan CXCR4 como correceptor y las cepas NSI tienen CCR5. Se considera que hay tres tipos de tropismo viral: las cepas R5, cuyos virus usan el correceptor CCR5, las cepas X4 que utilizan CXCR4 como correceptor, y las cepas X4R5 capaces de usar ambos correceptores.
Según el tropismo viral, el tratamiento médico más adecuado para el paciente variará. Actualmente el único fármaco comercializado como antagonista del receptor CCR5 con capacidad de bloquear la entrada viral es el Maraviroc (Pfeizer) , pero existen otros fármacos de la misma familia en diferentes estados de desarrollo clínico como el Vicriviroc (Schering-Plough) , SPRO 140 (Progenics Pharmaceuticals) y PF-232798 (ViiV Healthcare) . El uso de estos fármacos está limitado a pacientes infectados con cepas virales dependientes de CCR5. La próxima aparición de nuevos tratamientos augura un incremento significativo en el número de determinaciones que se tendrán que realizar para seleccionar los pacientes que se pueden beneficiar del tratamiento.
Según el informe anual de la Comisión de Seguridad y Cambio de los Estados Unidos, sobre las empresas Monogram y Quest, las tecnologías para identificar resistencias genéticas del VIH-1 a los nuevos tratamientos con inhibidores de la entrada viral son esenciales para un mejor manejo del tratamiento de la infección y proveen de información crítica que permite la prescripción personalizada. Este sistema de diagnóstico también ayuda al desarrollo y evaluación clínica de nuevos fármacos en desarrollo por las compañías farmacéuticas, especialmente en la selección de los pacientes que más se pueden beneficiar de la terapia.
La determinación del tropismo viral del VIH-1 se puede realizar mediante métodos fenotípicos y métodos genotípicos (Rose et al., 2009 Curr Opin HIV AIDS 4: 136-142) . Existen varios métodos fenotípicos de determinación del tropismo viral. El más antiguo es el conocido como MT2 y consiste en la detección de la presencia o ausencia de sincitios en células que expresen el receptor CD4. De entre los métodos más actuales se destaca el Trofile (Monogram Biosciences) . El método Trofile se basa en la generación de pseudovirus recombinantes para infectar líneas celulares que expresen en su superficie CD4, uno de los correceptores y que además contienen un gen marcador. La presencia del gen marcador permite la medida del nivel de infección de células diana, así como la inhibición de la infección mediante antagonistas, en caso que los pseudovirus recombinantes sean capaces de infectar las células. Para la generación de pseudovirus recombinantes se amplifica mediante PCR el gen de la envuelta del paciente y se clona en un vector de expresión que se cotransfecta en células que contienen el virus defectivo con una deleción en el propio gen de la envuelta. De esta manera se consigue un fenómeno de complementación en trans por la proteína de la envuelta clonada y se generan pseudovirus recombinantes que sólo tienen un ciclo infectivo (Rose et al., 2009 Curr Opin HIV AIDS 4: 136-142) .
Una aproximación para la generación de pseudovirus recombinantes sin necesidad de clonación consiste en la construcción de un ADN recombinante que contiene un promotor CMV y el gen de la envuelta que se transfecta a una línea celular para generar los pseudovirus recombinantes (Kirchherr et al., 2007 J Virol Methods 143 (1) : 104-111; Lin et al. Journal of Virological Methods 169 (2010) 39–46) .
Los métodos genotípicos son la principal alternativa gracias a la aplicación de metodologías in silico. El uso de tecnologías disruptivas como la secuenciación directa del genoma viral y la predicción del tipo de cepa usando algoritmos específicos actualmente presentan importantes problemas de sensibilidad y especificidad (Rose et al., 2009 Curr Opin HIV AIDS 4: 136-142) .
Otros métodos son los descritos en González et al., 2010 J Antimicrob Chemother. 65 (12) :2493-501; Raymond et al., 2010 J Clin Virol. 47 (2) :126-30; o Saliou et al., 2011 Antimicrob Agents Chemother. 55 (6) :2831-6.
Actualmente el diagnóstico del tipo de cepa viral se realiza principalmente en la empresa Monogram Biosciences (San Francisco, EEUU) con un coste por muestra elevado. En Europa la oferta es limitada, destacando Lab21 (Reino Unido) . Quest Diagnostics ha desarrollado un sistema de diagnóstico basado en la amplificación y la secuenciación de ADN viral (US7566532) . Los hospitales y centros de investigación realizan numerosos diagnósticos al año, y es altamente probable que el volumen siga creciendo en los próximos años por la incorporación de nuevos fármacos de la misma familia, lo cual hace necesario un método más sencillo de menos coste que puede realizarse en los propios hospitales y centros de investigación.
DESCRIPCION DE LA INVENCION
Los autores de la presente invención han desarrollado un método de expresión de productos de PCR directamente en el citoplasma de células de mamífero transfectadas, sin necesidad de clonar dicho producto en un plásmido de expresión eucariota. El método se basa en las siguientes premisas técnicas:
1. El extremo 5´ de cualquier cebador de PCR puede aceptar un fragmento de ADN de secuencia no complementaria al fragmento a amplificar sin pérdida significativa de rendimiento en la amplificación. Esto puede incluir un promotor, dianas de enzimas de restricción o colas de poliadenina, etc…
2. Dos productos de PCR con una pequeña región solapante pueden ser mezclados y amplificados nuevamente con dos cebadores externos que se unen al extremo 5´del primer producto y extremo 3´del segundo. Generando un producto de fusión de ambos fragmentos de ADN.
3. Los denominados Internal Ribosome Entries (IRES) de varios virus con replicación citoplásmica permiten la traducción de ARNm en ausencia de un capping estándar en el extremo 5´ del ARNm.
4. La ARN polimerasa del bacteriófago T7 puede ser eficientemente expresada funcionalmente en el citoplasma de células de mamífero.
Esta técnica permite la determinación del tropismo viral del VIH-1 en el que no es necesario el uso de vectores de clonaje y, por tanto, simplifica dicho método. El método de la presente invención permite el aislamiento y la amplificación del gen env a partir de una muestra aislada de un paciente infectado con el VIH-1, y lo hace de manera más directa y sencilla y en cantidades suficientes como para la determinación del tropismo viral en estudios de fusión célula-célula (formación de sincitios) .
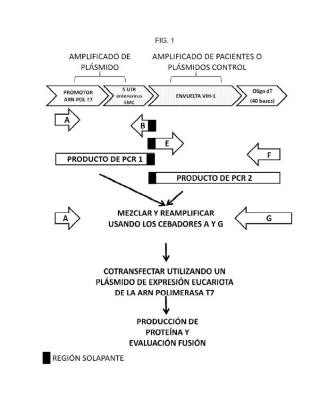
El método de la presente invención es sencillo y se basa en 7 cebadores que son empleados en sucesivas reacciones de PCR. Las secuencias de dichos cebadores permiten la generación de un amplicón que comprende un promotor citoplásmico fuerte, una secuencia IRES, el gen env del VIH-1 y un poliA.
El método de diagnóstico de la presente invención requiere un tiempo de procesamiento inferior que el de los métodos conocidos en el estado de la técnica anterior y su coste es menor. Además, los métodos genotípicos usados hasta la fecha, como por ejemplo la secuenciación, son poco específicos.
El método de determinación del tropismo viral del VIH-1 de la presente invención, brevemente, consiste en obtener un ADN copia del genoma del virus que infecta a un paciente,...
Reivindicaciones:
1. Método para la determinación del tropismo viral del virus de la inmunodeficiencia humana 1 que comprende las siguientes etapas:
(i) amplificar mediante PCR un promotor y una secuencia IRES a partir de un ácido nucleico molde que comprende dicho promotor y dicha secuencia IRES, mediante un cebador directo A, donde dicho cebador A hibrida con el extremo 5’ del promotor, y un cebador inverso B, donde dicho cebador B hibrida por su extremo 3’ con el extremo 3’ de la secuencia IRES y por su extremo 5’ es complementario al extremo 5’ del gen env del virus de la inmunodeficiencia humana 1;
(ii) amplificar mediante PCR una secuencia que comprende un fragmento del virus de la inmunodeficiencia humana 1 que incluye el gen env, a partir de ADN copia o ADN genómico mediante un cebador directo C y un cebador inverso D, donde dichos cebadores C y D hibridan con las secuencias que flanquean dicho gen env ;
(iii) amplificar mediante PCR la secuencia del gen env a partir del producto obtenido en la etapa (ii) , mediante un cebador directo E, donde el extremo 5’ de dicho cebador E es complementario con la secuencia IRES de la etapa (i) y donde el extremo 3’ de dicho cebador E hibrida con el extremo 5’ del gen env, y un cebador inverso F, que hibrida con el extremo 3’ del gen env;
(iv) ensamblar los productos de las etapas (i) y (iii) mediante PCR utilizando un cebador directo A que hibrida con el extremo 5´del producto de la etapa (i) y un cebador inverso G cuyo extremo 3´ hibrida con el extremo 3´ del producto de la etapa (iii) y cuyo extremo 5´comprende un poli-T;
(v) transfectar el amplicón obtenido en la etapa (iv) en células que expresan la proteína transactivadora de la actividad viral;
(vi) co-cultivar las células de la etapa (v) con células que contienen un gen marcador flanqueado por secuencias LTR y expresan:
a. El receptor CD4 y el correceptor CXCR4,
b. El receptor CD4 y el correceptor CCR5
c. El receptor CD4 y conjuntamente los correceptores CXCR4 y CCR5.
(vii) detectar y/o cuantificar la expresión del gen marcador, y
(viii) asociar los niveles de expresión del gen marcador detectados y/o cuantificados en la etapa (vii) al tropismo viral CXCR4, CCR5 o tropismo doble.
2. Método según la reivindicación 1, donde el promotor es el promotor de la ARN polimerasa del bacteriófago T7.
3. Método según cualquiera de las reivindicaciones 1 o 2, donde la secuencia IRES es la región 5’ UTR del enterovirus de la encefalomiocarditis.
4. Método según cualquiera de las reivindicaciones 1 a 3, donde la secuencia del cebador A es SEQ ID NO: 1, la secuencia del cebador B es SEQ ID NO: 2, la secuencia del cebador C es SEQ ID NO: 3, la secuencia del cebador D es SEQ ID NO: 4, la secuencia del cebador E es SEQ ID NO: 5 , la secuencia del cebador F es SEQ ID NO: 6 y la secuencia del cebador G es SEQ ID NO:7.
5. Método según cualquiera de las reivindicaciones 1 a 4, donde las células que expresan proteína transactivador de la actividad viral son la línea celular U251 MG.
6. Método según cualquiera de las reivindicaciones 1 a 5, donde las células que expresan proteína transactivador de la actividad viral comprenden la ARN polimerasa del bacteriófago T7.
7. Método según la reivindicación 6, donde las células se obtienen transfectando un plásmido de expresión de la ARN polimerasa del bacteriófago T7.
8. Método según cualquiera de las reivindicaciones 1 a 7, donde las células que contienen un gen marcador flanqueado por secuencias LTR, expresan el receptor CD4 y el correceptor CXCR4 son la línea celular HeLa.
9. Método según cualquiera de las reivindicaciones 1 a 8, donde las células que contienen un gen marcador flanqueado por secuencias LTR, expresan el receptor CD4 y el correceptor CCR5 son la línea celular P4-R5 MAGI.
10. Método según cualquiera de las reivindicaciones 1 a 9, donde las células contienen un gen marcador que es el de la β-galactosidasa, luciferasa, amilasa o proteína verde fluorescente.
11. Método según la reivindicación 10, donde el gen marcador es el de la βgalactosidasa.
12. Método según cualquiera de las reivindicaciones 10 o 11, donde la detección y/o cuantificación de la expresión del gen marcador se realiza mediante fluorescencia, marcaje colorimétrico, métodos inmunológicos, métodos espectroscópicos y/o PCR.
13. Método según cualquiera de las reivindicaciones 10 a 12, donde la detección y/o cuantificación de la expresión del gen marcador se realiza mediante tinción de β-galactosidasa y/o actividad β-galactosidasa luminiscente.
14. Cebadores SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 5 y SEQ ID NO: 7.
15. Kit que comprende:
(i) un fragmento de ADN artificial producido por PCR que incluye en el extremo 5’ el promotor de la ARN polimerasa del bacteriófago T7 y en el extremo 3´ una secuencia IRES y un fragmento del gen env del virus de la inmunodeficiencia humana 1;
(ii) un cebador directo A, SEQ ID NO: 1, que hibrida con la secuencia flanqueante extremo 5’ el promotor de la ARN polimerasa del bacteriófago T7
(iii) un cebador inverso B, SEQ ID NO: 2, que hibrida en el extremo 3´ una secuencia IRES y en el extremo 5´con un fragmento del gen env del virus de la inmunodeficiencia humana 1;
(iv) un cebador directo C, SEQ ID NO: 3, que hibrida con la secuencia flanqueante 5’ del gen env;
(v) un cebador inverso D, SEQ ID NO: 4, que hibrida con la secuencia flanqueante 3’ del gen env;
(vi) un cebador directo E, SEQ ID NO: 5, que por su extremo 5’ es complementario con la secuencia IRES y por su extremo 3’ hibrida con el extremo 5’ del gen env; y
(vii) un cebador inverso F, SEQ ID NO: 6, que hibrida por su zona 3’ con el extremo 3’ del gen env.
(viii) un cebador inverso G, SEQ ID NO: 7, que hibrida por su zona 3’ con el extremo 3’ del gen env y cuyo extremo 5’ comprende un poli-T.
16. Kit según la reivindicación 15, que además comprende un plásmido de expresión de la proteína transactivadora de la actividad viral TAT, un plásmido de expresión de la ARN polimerasa del bacteriófago T7, un plásmido de 5 expresión la proteína rev, un plásmido de expresión de la proteína de la envuelta gp160 y/o un plásmido de expresión de la proteína verde fluorescente.
17. Uso de los cebadores SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 5, SEQ ID NO: 6 y SEQ ID NO: 7 o del kit según cualquiera de las reivindicaciones 15 a 10 16 para determinar el tropismo viral del virus de la inmunodeficiencia humana 1.
FIG. 1
FIG. 2
FIG. 3
LISTA DE SECUENCIAS
<110> Universidad de Jaen <120> METODO DE DETERMINACION DE TROPISMO DEL VIH-1 Y KIT ASOCIADO
<130> ES1881.10
<160> 6
<170> PatentIn version 3.5
<210> 1
<211> 32
<212> DNA
<213> Secuencia artificial
<220>
<223> T7-IRES-FORWARD
<400> 1 taatacgact cactataggc tagcctcgag ga <210> 2
<211> 48
<212> DNA
<213> Secuencia artificial
<220>
<223> T7-IRES-REVERSE
<400> 2 ttcctgccat aggagatgcc taagtcgact ctagaggatc ccgggttg
<210> 3
<211> 33
<212> DNA
<213> Oligonucleótido Env-1
<400> 3 tacagtgcag gggaaagaat aatagacata ata <210> 4
<211> 22
<212> DNA
<213> oligonucleótido Env-2
<400> 4 agacccagta caggcraraa gc
<210> 5
<211> 52
<212> DNA
<213> Secuencia artificial
<220>
<223> Oligonule6tido Env-3
<400> 5 caacccggga tcctctagag tcgacttagg catctcctat ggcaggaaga ag
<210> 6
<211> 26
<212> DNA 1
<213> Secuencia artificial
<220>
<223> Oligonucleótido Env-4
<400> 6 ccagtccccc cttttctttt aaaaag 26
<210> 7
<211> 66
<212> DNA
<213> Secuencia artificial
<220>
<223> Oligonucleótido Env-4-POLYT
<400> 7 tttttttttt tttttttttt tttttttttt tttttttttt ccagtccccc cttttctttt 60
aaaaag 66
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]