Meganucleasas monocatenarias diseñadas racionalmente con secuencias de reconocimiento no palindrómicas.
Una meganucleasa monocatenaria recombinante que comprende:
una primera subunidad LAGLIDADG que comprende una secuencia polipeptídica que tiene al menos un 85% de identidad de secuencia con los restos 9-151 de una meganucleasa I-CreI de tipo salvaje de SEC IDNº:
1 y que tiene un primer semisitio de reconocimiento;
una segunda subunidad LAGLIDADG que comprende un polipéptido que tiene al menos un 85 % deidentidad de secuencia con los restos 9-151 de una meganucleasa I-CreI de tipo salvaje de SEC ID Nº: 1 yque tiene un segundo semisitio de reconocimiento;
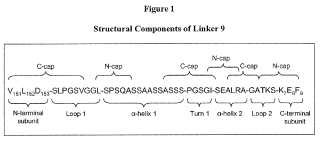
en la que dichas primera y segunda subunidades LAGLIDADG están unidas covalentemente con unengarce que consiste en cualquiera de las SEC ID Nº: 77-93 o con un engarce que comprende del extremoN terminal al C terminal un primer bucle, una primera hélice α, una primera vuelta, una segunda hélice α yun segundo bucle,
y en la que dichas primera y segunda subunidades LAGLIDADG son capaces de actuar juntas parareconocer y escindir una secuencia de ADN no palindrómica que es un híbrido de dicho primer semisitio dereconocimiento y dicho segundo semisitio de reconocimiento.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2008/082072.
Solicitante: Precision Biosciences, Inc.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 302 East Pettigrew Street, Dibrell Building, Suite A-100 Durham, NC 27701 ESTADOS UNIDOS DE AMERICA.
Inventor/es: Smith,James Jefferson, Jantz,Derek.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N9/22 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › Ribonucleasas.
PDF original: ES-2422291_T3.pdf
Fragmento de la descripción:
Meganucleasas monocatenarias diseñadas racionalmente con secuencias de reconocimiento no palindrómicas.
Campo de la invención La invención se refiere al campo de la biología molecular y tecnología de ácidos nucleicos recombinantes. En particular, la invención se refiere a meganucleasas de origen no natural diseñadas racionalmente en las que un par de subunidades enzimáticas que tienen especificidad para semisitios de secuencia de reconocimiento diferentes se unen en un polipéptido individual para formar un heterodímero funcional con una secuencia de reconocimiento no palindrómica. La invención también se refiere a procedimientos para producir dichas meganucleasas y procedimientos para producir ácidos nucleicos recombinantes y organismos que usan dichas meganucleasas.
Antecedentes de la invención La ingeniería genómica requiere la capacidad para insertar, suprimir, sustituir y manipular de otro modo secuencias genéticas específicas dentro de un genoma, y tiene numerosas aplicaciones terapéuticas y biotecnológicas. El desarrollo de medios eficaces para modificación del genoma sigue siendo un objetivo principal en la terapia génica, agrotecnología, y biología sintética (Porteus y col. (2005) , Nat. Biotechnol. 23: 967-73; Tzfira y col. (2005) , Trends Biotechnol. 23: 567-9; McDaniel y col. (2005) , Curr. Opin. Biotechnol. 16: 476-83) . Un procedimiento habitual para insertar o modificar una secuencia de ADN implica introducir una secuencia de ADN transgénico flanqueada por secuencias homólogas de la diana genómica y seleccionar o explorar con respecto a un acontecimiento recombinante homólogo exitoso. La recombinación con el ADN transgénico se produce pocas veces pero puede estimularse por una rotura bicatenaria en el ADN genómico en el sitio diana. Se ha empleado numerosos procedimientos para crear roturas bicatenarias de ADN, incluyendo irradiación y tratamientos químicos. Aunque estos procedimientos estimulan eficazmente la recombinación, las roturas bicatenarias se dispersan aleatoriamente en el genoma, lo que puede ser altamente mutagénico y tóxico. En la actualidad, la incapacidad para dirigir modificaciones génicas a sitios únicos dentro de un fondo cromosómico es un impedimento importante para la ingeniería genómica exitosa.
Un enfoque para conseguir este objetivo es estimular la recombinación homóloga en una rotura bicatenaria en un locus diana usando una nucleasa con especificidad por una secuencia que es suficientemente grande para estar presente solamente en un único sitio dentro del genoma (véase, por ejemplo, Porteus y col. (2005) , Nat. Biotechnol.
23: 967-73) . La eficacia de esta estrategia se ha demostrado en una diversidad de organismos usando fusiones quiméricas entre un dominio de unión a ADN de dedo de cinc obtenido por ingeniería genética y el dominio de nucleasa no específico de la enzima de restricción FokI (Porteus (2006) , Mol. Ther. 13: 438-46; Wright y col. (2005) , Plant J. 44: 693-705; Urnov y col. (2005) , Nature 435: 646-51) . Aunque estas nucleasas de dedos de cinc artificiales estimulan la recombinación específica de sitio, conservan actividad de escisión no específica residual que resulta de la infrarregulación del dominio nucleasa y frecuentemente se escinden en sitios no pretendidos (Smith y col. (2000) , Nucleic Acids Res. 28: 3361-9) . Dicha escisión no pretendida puede provocar mutaciones y toxicidad en el organismo tratado (Porteus y col. (2005) , Nat. Biotechnol. 23: 967-73) .
Un grupo de nucleasas de origen natural que reconocen sitios de escisión de 15-40 pares de bases habitualmente hallados en los genomas de plantas y hongos pueden proporcionar una alternativa de ingeniería genómica menos tóxica. Dichas “meganucleasas” o “endonucleasas buscadoras” se asocian con frecuencia con elementos de ADN parasitarios, tales como intrones de autocorte y empalme de grupo I e inteínas. Promueven de forma natural la recombinación homóloga o inserción génica en localizaciones específicas en el genoma huésped produciendo una rotura bicatenaria en el cromosoma, que recluta la maquinaria de reparación de ADN celular (Stoddard (2006) , Q. Rev. Biophys. 38: 49-95) . Las meganucleasas se agrupan habitualmente en cuatro familias: la familia LAGLIDADG, la familia GIY-YIG, la familia de caja His-Cys y la familia HNH. Estas familias se caracterizan por motivos estructurales que afectan a la actividad catalítica y secuencia de reconocimiento. Por ejemplo, los miembros de la familia LAGLIDADG se caracterizan por tener una o dos copias del motivo LAGLIDADG conservado (véase Chevalier y col. (2001) , Nucleic Acids Res. 29 (18) : 3757-3774) . Las meganucleasas LAGLIDADG con una única copia del motivo LAGLIDADG (“meganucleasas mono-LAGLIDADG”) forman homodímeros, mientras que los miembros con dos copias del motivo LAGLIDADG (“meganucleasas di-LAGLIDADG”) se encuentran como monómeros. Las meganucleasas mono-LAGLIDADG tales como 1-CreI, I-CeuI e I-MsoI reconocen y escinden sitios de ADN que son palindrómicos o seudopalindrómicos, mientras que las meganucleasas di-LAGLIDADG tales como I-SceI, I-AniI e I-DmoI reconocen generalmente sitios de ADN que no son palindrómicos (Stoddard (2006) , Q. Rev. Biophys. 38: 49-95) .
Se han usado meganucleasas naturales de la familia de LAGLIDADG para promover eficazmente la modificación genómica específica de sitio en plantas, levadura, Drosophila, células de mamífero y ratones, pero este enfoque se ha limitado a la modificación de genes homólogos que conservan la secuencia de reconocimiento de meganucleasas (Monnat y col. (1999) , Biochem. Biophys. Res. Commun. 255: 88-93) o a genomas previamente modificados por ingeniería genética en los que se ha introducido una secuencia de reconocimiento (Rouet y col. (1994) , Mol. Cell. Biol. 14: 8096-106; Chilton y col. (2003) , Plant Physiol. 133: 956-65; Puchta y col. (1996) , Proc. Natl. Acad. Sci. USA
93: 5055-60; Rong y col. (2002) , Genes Dev. 16: 1568-81; Gouble y col. (2006) , J. Gene Med. 8 (5) :616-622) .
La implementación sistemática de modificación génica estimulada por nucleasa requiere el uso de enzimas modificadas por ingeniería genética con especificidades adaptadas para dirigir roturas de ADN a sitios existentes en un genoma y, por lo tanto, ha habido gran interés en adaptar meganucleasas para estimular modificaciones génicas en sitios médica o biotecnológicamente relevantes (Porteus y col. (2005) , Nat. Biotechnol. 23: 967-73; Sussman y col. (2004) , J. Mol. Biol. 342: 31-41; Epinat y col. (2003) , Nucleic Acids Res. 31: 2952-62) .
I-CreI es un miembro de la familia LAGLIDADG que reconoce y corta una secuencia de reconocimiento de 22 pares de bases en el cromosoma del cloroplasto y que presenta una diana atractiva para rediseño de meganucleasas. La enzima de tipo salvaje es un homodímero en el que cada monómero entra en contacto directo con 9 pares de bases en la secuencia de reconocimiento de longitud completa. Se han usado técnicas de selección genética para modificar la preferencia de sitio de escisión de I-CreI de tipo salvaje (Sussman y col. (2004) , J. Mol. Biol. 342: 31-41; Chames y col. (2005) , Nucleic Acids Res. 33: e178; Seligman y col. (2002) , Nucleic Acids Res. 30: 3870-9, Arnould y col. (2006) , J. Mol. Biol. 355: 443-58, Rosen y col. (2006) , Nucleic Acids Res. 34: 4791-4800, Arnould y col. (2007) .
J. Mol. Biol. 371: 49-65, documentos WO 2008/010009, WO 2007/093918, WO 2007/093836, WO 2006/097784, WO 2008/059317, WO 2008/059382, WO 2008/102198, WO 2007/060495, WO 2007/049156, WO 2006/097853, WO 2004/067736) . Más recientemente, se describió un procedimiento de diseño racional de meganucleasas mono-LAGLIDADG que es capaz de rediseñar de forma exhaustiva I-CreI y otras de tales meganucleasas para dirigir a sitios de ADN ampliamente divergentes, incluyendo sitios en genomas de mamífero, levadura, planta, bacterianos y virales (documento WO 2007/047859) .
Una limitación importante para usar meganucleasas mono-LAGLIDADG tales como I-CreI para la mayoría de aplicaciones de ingeniería genética es el hecho de que estas enzimas se dirigen de forma natural a sitio de reconocimiento de ADN palindrómicos. Dichos sitios de ADN palindrómicos largos (10-40 pb) son escasos en la naturaleza y es improbable que aparezcan por casualidad en un sitio de ADN de interés. Para dirigirse a un sitio de ADN no palindrómico con una meganucleasa mono-LAGLIDADG, se puede producir un par de monómeros que reconocen los dos semisitios diferentes y que heterodimerizan para formar una meganucleasa que escinde el sitio no palindrómico deseado. Puede conseguirse heterodimerización mediante coexpresión de un par de monómeros de meganucleasa en una célula huésped o mezclando... [Seguir leyendo]
Reivindicaciones:
1. Una meganucleasa monocatenaria recombinante que comprende:
una primera subunidad LAGLIDADG que comprende una secuencia polipeptídica que tiene al menos un 85
% de identidad de secuencia con los restos 9-151 de una meganucleasa I-CreI de tipo salvaje de SEC ID
Nº: 1 y que tiene un primer semisitio de reconocimiento;
una segunda subunidad LAGLIDADG que comprende un polipéptido que tiene al menos un 85 % de identidad de secuencia con los restos 9-151 de una meganucleasa I-CreI de tipo salvaje de SEC ID Nº: 1 y
que tiene un segundo semisitio de reconocimiento;
en la que dichas primera y segunda subunidades LAGLIDADG están unidas covalentemente con un engarce que consiste en cualquiera de las SEC ID Nº.
7. 93 o con un engarce que comprende del extremo N terminal al C terminal un primer bucle, una primera hélice a, una primera vuelta, una segunda hélice a y un segundo bucle,
y en la que dichas primera y segunda subunidades LAGLIDADG son capaces de actuar juntas para reconocer y escindir una secuencia de ADN no palindrómica que es un híbrido de dicho primer semisitio de reconocimiento y dicho segundo semisitio de reconocimiento.
2. La meganucleasa monocatenaria recombinante de la reivindicación 1 en la que dichas primera y segunda subunidades LAGLIDADG se unen covalentemente con un engarce que consiste en cualquiera de las SEC ID Nº.
7. 93.
3. La meganucleasa monocatenaria recombinante de la reivindicación 1 en la que dichas primera y segunda subunidades LAGLIDADG se unen covalentemente con un engarce que comprende del extremo N terminal al C terminal, un primer bucle, una primera hélice a, una primera vuelta, una segunda hélice a y un segundo bucle.
4. La meganucleasa monocatenaria recombinante de las reivindicaciones 1-3 en la que al menos uno de dichos dominios LAGLIDADG comprende al menos una modificación de aminoácido seleccionada del grupo que consiste en: Y75, L75, C75, Y139, C46, A46, H75, R75, H46, K46, R46, K70, E70, E75, E46, D46, Q70, C70, L70, Q75, H139, Q46, G70, A70, S70, G46, T44, A44, V44, I44, L44, N44, D70, K44, R44, H70, D44, E44, C44, Q68, C24, E68, F68, K24, R24, M68, C68, L68, H68, Y68, K68, A26, Q77, E77, K26, R77, E26, S77, Q26, S26, E42, R42, K28, C28, Q42, M66, K66, Q40, E40, R28, R40, C40, I40, V40, C79, I79, V79, Q28, A40, A79, A28, H28, S40, S28, E38, K30, R30, K38, R38, E30, I38, L38, C38, H38, N38, Q30, F33, E33, D33, H33, L33, V33, I33, C33, R32, R33, E32, K32, L32, V32, A32, C32, D32, I32, N32, H32, Q32 y T32.
5. La meganucleasa monocatenaria recombinante de las reivindicaciones 1-4 en la que cada una de dichas subunidades LAGLIDADG tiene un semisitio de reconocimiento seleccionado del grupo que consiste en las SEC ID Nº: 7-30.
6. La meganucleasa monocatenaria recombinante de la reivindicación 5 en la que al menos una de dichas subunidades LAGLIDADG tiene un semisitio de reconocimiento seleccionado del grupo que consiste en las SEC ID Nº: 7-30; y la otra de dichas subunidades LAGLIDADG tiene un semisitio de reconocimiento que difiere en una modificación de al menos un par de bases de un semisitio de reconocimiento seleccionado del grupo que consiste en las SEC ID Nº: 7-30.
7. La meganucleasa monocatenaria recombinante de las reivindicaciones 1-6 en la que dicho engarce comprend.
2. 56 restos.
8. Un procedimiento ex vivo para producir una célula eucariota modificada genéticamente que incluye una secuencia exógena de interés insertada en un cromosoma de dicha célula eucariota, que comprende:
(a) transfectar una célula eucariota con uno o más ácidos nucleicos que incluyen
(i) una primera secuencia de ácido nucleico que codifica una meganucleasa y
(ii) una segunda secuencia de ácido nucleico que incluye dicha secuencia de interés;
en el que dicha meganucleasa produce un sitio de escisión en dicho cromosoma y dicha secuencia de interés se inserta en dicho cromosoma en dicho sitio de escisión; y en el que dicha meganucleasa es una meganucleasa monocatenaria recombinante de una cualquiera de las reivindicaciones 1-7 o
(b) introducir una proteína meganucleasa en una célula eucariota; y transfectar dicha célula eucariota con un ácido nucleico que incluye dicha secuencia de interés; en el que dicha meganucleasa produce un sitio de escisión en dicho cromosoma y dicha secuencia de interés se inserta en dicho cromosoma en dicho sitios de escisión; y en el que dicha meganucleasa es una meganucleasa monocatenaria recombinante de una cualquiera de las reivindicaciones 1-7.
9. Un procedimiento ex vivo para producir una célula eucariota modificada genéticamente alterando una secuencia diana en un cromosoma de dicha célula eucariota, que comprende: transfectar una célula eucariota con un ácido nucleico que modifica una meganucleasa; en el que dicha meganucleasa produce un sitio de escisión en dicho cromosoma y dicha secuencia diana se altera por unión de extremos no homólogos en dicho sitio de escisión; y en el que dicha meganucleasa es una meganucleasa monocatenaria recombinante de una cualquiera de las reivindicaciones 1-7.
10. Un procedimiento para producir un organismo modificado genéticamente no humano que comprende: producir una célula eucariota modificada genéticamente de acuerdo con el procedimiento de una cualquiera de las reivindicaciones 8-9 en el que la célula eucariota es un protoplasto, un gameto no humano, un cigoto no humano, un blastocisto no humano o una célula madre embrionaria no humana; y cultivar dicha célula eucariota modificada genéticamente para producir dicho organismo modificado genéticamente.
11. El procedimiento de la reivindicación 10 en el que el organismo modificado genéticamente no humano es una planta y la célula eucariota modificada genéticamente es un protoplasto.
12. El procedimiento de la reivindicación 10 en el que el organismo modificado genéticamente no humano es un
animal y la célula eucariota modificada genéticamente es un cigoto no humano o una célula madre embrionaria no humana.
13. Una meganucleasa monocatenaria recombinante de una cualquiera de las reivindicaciones 1-7 para su uso en terapia génica.
14. Una meganucleasa monocatenaria recombinante de una cualquiera de las reivindicaciones 1-7 para su uso en el
tratamiento de una infección por patógeno viral en un huésped eucariota o una infección por patógeno procariota en un huésped eucariota.
Patentes similares o relacionadas:
Procedimientos y composiciones para el tratamiento de una afección genética, del 24 de Junio de 2020, de Sangamo Therapeutics, Inc: Una célula precursora de glóbulos rojos genomanipulada caracterizada por una modificación genómica dentro del exón 2 o el exón 4 de BCL11A o dentro de BCL11A-XL […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para el tratamiento de enfermedades por almacenamiento lisosomal, del 27 de Mayo de 2020, de Sangamo Therapeutics, Inc: Uno o más transgenes y una o más nucleasas con dedos de zinc (ZFN) para su uso en un método de tratamiento de una enfermedad por almacenamiento lisosomal, el método […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Métodos y composiciones para modular PD1, del 13 de Mayo de 2020, de Sangamo Therapeutics, Inc: Célula aislada que comprende una inserción o una deleción en un gen de PD1 endógeno dentro de, o entre, las secuencias mostradas en SEQ ID NO: 56 y SEQ ID NO: 60 del gen de PD1 […]
Cepas bacterianas que expresan genes de metilasa y sus usos, del 22 de Abril de 2020, de LOMA LINDA UNIVERSITY: Una bacteria aislada para usar en la producción de ADN plasmídico metilado, en donde la bacteria comprende un polinucleótido exógeno que codifica una CpG metilasa […]
Método y composiciones para reducir productos de amplificación no específicos, del 25 de Marzo de 2020, de Paragon Genomics, Inc: Un método para reducir productos de amplificación no específicos de una reacción de extensión de cebadores dependiente de plantilla, comprendiendo el método: […]
Proteínas que tienen actividad nucleasa, proteínas de fusión y usos de estas, del 18 de Marzo de 2020, de HELMHOLTZ ZENTRUM MUNCHEN DEUTSCHES FORSCHUNGSZENTRUM FUR GESUNDHEIT UND UMWELT (GMBH): Una molécula de ácido nucleico que codifica (I) un polipéptido que tiene la actividad de una endonucleasa, que es (a) una molécula de ácido nucleico que […]