Detección rápida de SNP basada en secuencias utilizando ensayos de ligación.
Procedimiento para la detección rápida de por lo menos 100 secuencias de nucleótidos elegidas como objetivo en una pluralidad de muestras,
comprendiendo el procedimiento las etapas de:
(a) proporcionar para cada secuencia de nucleótidos elegida como objetivo en cada una de las muestras:
I. una primera sonda y una segunda sonda, comprendiendo la primera sonda una sección específica elegida como objetivo en su extremo 3' y una primera sección etiquetada que no es complementaria a la secuencia de nucleótidos elegida como objetivo y que comprende una primera secuencia de enlace con un cebador, comprendiendo la segunda sonda una segunda sección específica elegida como objetivo en su extremo 5' y una segunda sección etiquetada que no es complementaria a la secuencia de nucleótidos elegida como objetivo y que comprende una segunda secuencia de enlace con el cebador, comprendiendo la primera o la segunda sección etiquetada(s), o ambas, una secuencia identificadora que se encuentra entre la secuencia primera o segunda correspondiente de enlace con el cebador y la sección específica elegida como objetivo primera o segunda correspondiente, o
II. una sonda circularizable, en la que las sondas primera y segunda tal como se definen en la alternativa I se combinan en una sonda; o
III. una primera y una segunda sonda tal como se definen en la alternativa I, comprendiendo además la primera sonda una sección de sujeción que puede hibridarse con una sección de sujeción complementaria que se encuentra en la segunda sonda, por lo que las secciones de sujeción son sustancialmente no complementarias a la secuencia elegida como objetivo. o
IV. una primera y una segunda sonda tal como se definen en la alternativa I, excepto en que la primera sonda no comprende una primera secuencia de enlace con el cebador y en que se proporciona una sonda del compuesto que comprende una sección que puede hibridarse con una parte de la primera sección específica elegida como objetivo la primera sonda y comprende además una sección de enlace con el cebador, y en el que el alargamiento de la sonda del compuesto proporciona una sonda del compuesto alargada amplificable,
(b) permitir que la primera y la segunda sección específica elegidas como objetivo de las sondas primera y segunda correspondientes o la sonda circularizable para hibridarse con la secuencia elegida como objetivo,
(c) ligar las sondas primera y segunda o la sonda circularizable cuando las secciones específicas elegidas como objetivo correspondientes de las sondas o la sonda circularizable se hibridan con secciones sustancialmente adyacentes en la secuencia elegida como objetivo para proporcionar sondas ligadas,
(d) opcionalmente, amplificar las sondas ligadas con un primer y un segundo cebador para proporcionar amplicones,
(e) someter las sondas ligadas o amplicones a la tecnología de secuenciación rápida para determinar por lo menos una parte de la secuencia de nucleótidos, comprendiendo por lo menos la(s) secuencia(s) identificadora(s), de las sondas ligadas o los amplicones,
(f) identificar la presencia de la secuencia de nucleótidos elegida como objetivo en la muestra determinando la presencia o ausencia de la secuencia identificadora en la secuencia de nucleótidos de la etapa (e).
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/NL2007/000055.
Solicitante: KEYGENE N.V..
Nacionalidad solicitante: Países Bajos.
Dirección: AGRO BUSINESS PARK 90 6708 PW WAGENINGEN PAISES BAJOS.
Inventor/es: VAN EIJK,MICHAEL,JOSEPHUS,THERESIA, HOGERS,RENE,CORNELIS,JOSEPHUS.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2446927_T3.pdf
Fragmento de la descripción:
Detección rápida de SNP basada en secuencias utilizando ensayos de ligación.
CAMPO DE LA INVENCIÓN
La presente invención se refiere al campo de la biología molecular y de la biotecnología. En particular, la presente invención se refiere al campo de la detección de ácidos nucleicos, más particularmente, al diseño y a la composición de (colecciones) de sondas que se pueden utilizar para la detección rápida de ácidos nucleicos. La presente invención se refiere asimismo a procedimientos para la detección de ácidos nucleicos utilizando las sondas y las composiciones. La presente invención proporciona además sondas que pueden hibridarse con una secuencia elegida como objetivo de interés, cebadores para la amplificación de sondas ligadas, la utilización de dichas sondas y cebadores en la identificación y/o detección de secuencias de nucleótidos que están relacionadas con una amplia variedad de características genéticas y genes y equipos de reactivos de cebadores y/o sondas aptos para utilizar en el procedimiento según la presente invención. La presente invención encuentra aplicabilidad en el campo de la detección rápida de secuencias de nucleótidos elegidas como objetivo, de origen sintético, vegetal, animal o humano o combinaciones de los mismos. La presente invención encuentra aplicación particular en el campo de la determinación rápida del genotipo.
ANTECEDENTES DE LA INVENCIÓN
Cada vez existe más interés en la detección de secuencias específicas de ácidos nucleicos. Este interés no ha surgido únicamente a partir de la secuencia preliminar de nucleótidos descrita recientemente del genoma humano y muchos otros genomas y la presencia en el mismo, así como en los genomas de muchos otros organismos, de una cantidad abundante de polimorfismos de nucleótido único (SNP) y polimorfismos de pequeñas inserciones / deleciones (indel) , sino asimismo de tecnologías de marcadores (tales como AFLP) , SNPWave y el reconocimiento general de la importancia de la detección de secuencias específicas de ácido nucleico como indicación de, por ejemplo, enfermedades genéticas hereditarias. La detección de los distintos alelos del gen del cáncer de mama BRCA 1 para realizar una selección con respecto a la susceptibilidad hacia el cáncer de mama es únicamente uno de muchos ejemplos. El reconocimiento de que la presencia de sustituciones e indeles de nucleótidos únicos y en los genes proporciona una amplia variedad de información se ha atribuido asimismo a dicho aumento del interés. En la actualidad se reconoce generalmente que dichas sustituciones de nucleótido único constituyen una de las principales causas de un número significativo de enfermedades heredadas monogenética y multigenéticamente, por ejemplo, en seres humanos, o de algún otro modo se encuentran implicadas en el desarrollo de fenotipos complejos, tales como las características de rendimiento de especies vegetales y de ganado. Por lo tanto, las sustituciones de un nucleótido único en muchos casos están asimismo relacionadas con, o por lo menos indican, características importantes de seres humanos, y especies vegetales y animales.
El análisis de dichas sustituciones e indeles de nucleótido único proporcionarán una gran cantidad de información valiosa, que tendrá amplias implicaciones en medicina y agricultura en el sentido más amplio posible. En general se prevé, por ejemplo, que dichos desarrollos proporcionen una la medicación específica para cada paciente. Para analizar dichos polimorfismos genéticos, existe una necesidad cada vez mayor de procedimientos adecuados, fiables y rápidos que permitan la manipulación de grandes cantidades de muestras y grandes cantidades de (predominantemente) SNP de un modo rápido, sin comprometer significativamente la calidad de los datos obtenidos. Uno de los procedimientos principales utilizados en el análisis de los ácidos nucleicos de una secuencia conocida se basa en la hibridación de dos sondas con una secuencia elegida como objetivo y, cuando las sondas se hibridan de un modo adyacente a la secuencia elegida como objetivo, ligando las sondas.
Se ha descrito el principio del OLA (ensayo de ligación de oligonucleótidos) , entre otros, en la patente US n.º
4.988.617 (Landegren et al.) . Esta publicación describe un procedimiento para determinar la secuencia de ácido nucleico en una región de una secuencia de ácido nucleico conocida que presenta una mutación posible conocida. Para detectar la mutación, los oligonucleótidos se seleccionan para que se hibriden con los segmentos adyacentes de la secuencia a determinar de inmediato. Una de las sondas de oligonucleótidos seleccionadas presenta una región terminal en la que uno de los nucleótidos de la región terminal es complementario al nucleótido normal o al mutado de la posición correspondiente en la secuencia de ácido nucleico conocida. Se proporciona una ligasa que conecta covalentemente las dos sondas cuando se han emparejado correctamente y se encuentran inmediatamente adyacentes entre sí. La presencia, ausencia o cantidad de sondas enlazadas es una indicación de la presencia de la secuencia y/o mutación conocida.
Se han descrito otras variantes de técnicas basadas en el OLA, entre otros, en Nilsson et al. Human mutation, 2002, 19, 410-415; Science 1994, 265: 2085-2088; los documentos US n.º 5.876.924; WO 98/04745; WO 98/04746; US n.º 6.221.603; US n.º 5.521.065; US n.º 5.962.223; EP 185494B1; US n.º 6.027.889; US n.º 4.988.617; EP 246864B1; US n.º 6.156.178; EP 745140 B1; EP 964704 B1; WO 03/054511; US n.º 2003/0119004; US n.º 2003/190646; EP 1313880; US n.º 2003/0032016; EP 912761; EP 956359; US n.º 2003/108913; EP 1255871; EP 1194770; EP 1252334; WO96/15271; WO7/45559; US n.º 2003/0119004A1; US n.º 5.470.705.
Se han publicado avances particulares en las técnicas del OLA en Keygene, Wageningen, Países Bajos. En los documentos WO 2004/111271, WO2005/021794, WO2005/118847 y WO03/052142, se han descrito diversos procedimientos y diseños de la sonda que mejoran la fiabilidad de los ensayos de ligación de oligonucleótidos. Dichas solicitudes dan a conocer además la mejora significativa en los niveles múltiplex que se pueden alcanzar. Sin embargo, todas las publicaciones anteriores adolecen del inconveniente de que se basan en procedimientos de detección electroforéticos o basados en matrices. Un inconveniente adicional es la amplia variación de la longitud de las sondas utilizadas, lo que puede provocar que la amplificación sea menos constante.
Es evidente que existe una necesidad continua de sondas de oligonucleótidos que combinen las ventajas y eviten los inconvenientes específicos de los diversos tipos de sonda de ligación y de los procedimientos de detección conocidos en la técnica. Existe asimismo una necesidad de continuar mejorando la tecnología proporcionando sondas que presenten ventajas adicionales. Constituye uno de los objetivos de la presente invención proporcionar dichas sondas. Constituye un objetivo adicional de la presente invención evitar los inconvenientes de las sondas conocidas comúnmente tal como se ha mencionado anteriormente en la presente memoria. Constituye un objetivo adicional de la presente invención proporcionar sondas que sean aptas para los procedimientos de detección rápida. Constituye asimismo un objetivo de la presente invención proporcionar un procedimiento para un procedimiento eficiente, fiable y/o rápido para la detección de secuencias de nucleótidos elegidas como objetivo, preferentemente realizando ensayos de ligación de oligonucleótidos.
Los presentes inventores se han propuesto eliminar o por lo menos disminuir los problemas existentes en la técnica anterior, tratando de mantener al mismo tiempo los muchos aspectos ventajosos de la misma, y continuar mejorando la tecnología. Otros problemas de la técnica anterior y las soluciones proporcionadas a los mismos por la presente invención se pondrán de manifiesto a lo largo de la descripción, las figuras y las diversas formas de realización descritas en la presente memoria.
SUMARIO DE LA INVENCIÓN
Los presentes inventores han podido combinar tecnologías de secuenciación rápida novedosas con la versatilidad de los ensayos basados en la ligación de oligonucleótidos. En particular, la presente invención se refiere a un procedimiento de detección rápida de secuencias de nucleótidos elegidas como objetivo basándose en ensayos de ligación de oligonucleótidos, en el que las sondas utilizadas en los ensayos de ligación se modifican de tal modo que se puede utilizar un procedimiento de secuenciación rápida para revelar inequívocamente la presente ausencia de la cantidad de las una o más secuencias de nucleótidos elegidas como objetivo.... [Seguir leyendo]
Reivindicaciones:
1. Procedimiento para la detección rápida de por lo menos 100 secuencias de nucleótidos elegidas como objetivo en una pluralidad de muestras, comprendiendo el procedimiento las etapas de:
(a) proporcionar para cada secuencia de nucleótidos elegida como objetivo en cada una de las muestras:
I. una primera sonda y una segunda sonda, comprendiendo la primera sonda una sección específica elegida como objetivo en su extremo 3' y una primera sección etiquetada que no es complementaria a la secuencia de nucleótidos elegida como objetivo y que comprende una primera secuencia de enlace con un cebador, comprendiendo la segunda sonda una segunda sección específica elegida como objetivo en su extremo 5' y una segunda sección etiquetada que no es complementaria a la secuencia de nucleótidos elegida como objetivo y que comprende una segunda secuencia de enlace con el cebador, comprendiendo la primera o la segunda sección etiquetada (s) , o ambas, una secuencia identificadora que se encuentra entre la secuencia primera o segunda correspondiente de enlace con el cebador y la sección específica elegida como objetivo primera o segunda correspondiente,
o II. una sonda circularizable, en la que las sondas primera y segunda tal como se definen en la alternativa I se combinan en una sonda; o III. una primera y una segunda sonda tal como se definen en la alternativa I, comprendiendo además la primera sonda una sección de sujeción que puede hibridarse con una sección de sujeción complementaria que se encuentra en la segunda sonda, por lo que las secciones de sujeción son sustancialmente no complementarias a la secuencia elegida como objetivo.
o IV. una primera y una segunda sonda tal como se definen en la alternativa I, excepto en que la primera sonda no comprende una primera secuencia de enlace con el cebador y en que se proporciona una sonda del compuesto que comprende una sección que puede hibridarse con una parte de la primera sección específica elegida como objetivo la primera sonda y comprende además una sección de enlace con el cebador, y en el que el alargamiento de la sonda del compuesto proporciona una sonda del compuesto alargada amplificable,
(b) permitir que la primera y la segunda sección específica elegidas como objetivo de las sondas primera y segunda correspondientes o la sonda circularizable para hibridarse con la secuencia elegida como objetivo,
(c) ligar las sondas primera y segunda o la sonda circularizable cuando las secciones específicas elegidas como objetivo correspondientes de las sondas o la sonda circularizable se hibridan con secciones sustancialmente adyacentes en la secuencia elegida como objetivo para proporcionar sondas ligadas,
(d) opcionalmente, amplificar las sondas ligadas con un primer y un segundo cebador para proporcionar amplicones,
(e) someter las sondas ligadas o amplicones a la tecnología de secuenciación rápida para determinar por lo menos una parte de la secuencia de nucleótidos, comprendiendo por lo menos la (s) secuencia (s) identificadora (s) , de las sondas ligadas o los amplicones,
(f) identificar la presencia de la secuencia de nucleótidos elegida como objetivo en la muestra determinando la presencia o ausencia de la secuencia identificadora en la secuencia de nucleótidos de la etapa (e) .
2. Procedimiento según la reivindicación 1, en el que las sondas primera y segunda se emparejan con secciones no adyacentes en la secuencia elegida como objetivo y se proporcionan una polimerasa y nucleótidos de tal modo que se llena la distancia entre las sondas primera y segunda.
3. Procedimiento según cualquiera de las reivindicaciones 1 a 2, en el que la secuenciación se realiza mediante secuenciación rápida sobre un soporte sólido.
4. Método según la reivindicación 1, en el que la secuenciación rápida comprende las etapas de:
- emparejar las sondas ligadas o amplicones con perlas, emparejándose cada perla con una única sonda ligada o amplicón;
-emulsionar las perlas en microrreactores, comprendiendo cada microrreactor de agua en aceite una única perla;
- realizar la PCR en emulsión para amplificar sondas ligadas o amplicones en la superficie de las perlas; - opcionalmente, seleccionar / enriquecer las perlas que comprenden fragmentos amplificados ligados adaptadores;
- cargar las perlas en pocillos, comprendiendo cada pocillo una única perla; y
- generar una señal de pirofosfato.
5. Método según la reivindicación 1, en el que la secuenciación rápida comprende las etapas de:
- emparejar las sondas ligadas o amplicones con perlas, emparejándose cada perla con una única sonda ligada o amplicón;
-emulsionar las perlas en microrreactores, comprendiendo cada microrreactor de agua en aceite una única perla;
- realizar la PCR en emulsión para amplificar sondas ligadas o amplicones en la superficie de las perlas; - opcionalmente, seleccionar / enriquecer las perlas que comprenden fragmentos amplificados ligados adaptadores;
- cargar las perlas en pocillos, comprendiendo cada pocillo una única perla; y
- determinar la secuencia de nucleótidos de las sondas ligadas amplificadas o amplicones utilizando nucleótidos de terminación reversibles etiquetados.
6. Método según la reivindicación 1, en el que la secuenciación rápida comprende las etapas de:
- emparejar las sondas ligadas o amplicones con una superficie que comprende los cebadores primero y segundo o las secuencias de enlace con el cebador primera y segunda, respectivamente,
- realizar la amplificación en puente para proporcionar grupos de sondas amplificadas o amplicones,
- determinar la secuencia de nucleótidos de las sondas ligadas amplificadas o amplicones utilizando nucleótidos de terminación reversibles etiquetados.
7. Método según la reivindicación 1, en el que la secuenciación rápida comprende las etapas de:
- emparejar las sondas ligadas o amplicones con una superficie que comprende los cebadores primero y segundo o las secuencias de enlace con el cebador primera y segunda, respectivamente,
- realizar la amplificación en puente para proporcionar grupos de sondas amplificadas o amplicones,
- generar una señal de pirofosfato.
8. Procedimiento según la reivindicación 1, en el que el identificador es un identificador de muestras y/o un identificador combinado de locus / alelos.
9. Procedimiento según la reivindicación 1, en el que las sondas ligadas se amplifican con un primer y un segundo cebador para proporcionar amplicones.
10. Procedimiento según la reivindicación 9, en el que el cebador comprende una secuencia de anclaje 3'.
11. Procedimiento según la reivindicación 1, en el que para dos o más muestras o para dos o más combinaciones de locus / alelo, las secuencias identificadoras se utilizan para determinar el genotipo de la (s) muestra (s) para una o más secuencias y/o polimorfismos, tales como SNP y/o indeles.
12. Uso de la secuenciación rápida en la determinación de la presencia o ausencia de por lo menos 100 secuencias elegidas como objetivo en una pluralidad de muestras utilizando un ensayo basado en la ligación, en el que para la pluralidad de secuencias elegidas como objetivo se proporcionan una primera sonda y una segunda sonda, comprendiendo la primera sonda una sección específica de como objetivo en su extremo 3' y una primera sección etiquetada que no es complementaria a la secuencia de nucleótidos elegida como objetivo y que comprende una primera secuencia de enlace con un cebador, comprendiendo la segunda sonda una segunda sección específica elegida como objetivo en su extremo 5' y una segunda sección etiquetada que no es complementaria a la secuencia de nucleótidos elegida como objetivo y que comprende una segunda secuencia de enlace con el cebador, comprendiendo la primera o la segunda sección etiquetada (s) , o ambas, una secuencia identificadora que se encuentra entre la secuencia primera o segunda correspondiente de enlace con el cebador y la sección específica elegida como objetivo primera o segunda correspondiente, en el que el identificador no comprende 2 o más bases consecutivas idénticas y en el que todos los identificadores difieren entre sí en por lo menos dos bases, y en el que las sondas primera y segunda se ligan cuando las secciones específicas elegidas como objetivo correspondientes de las sondas se hibridan con secciones sustancialmente adyacentes de la secuencia elegida como objetivo para proporcionar sondas ligadas y las sondas ligadas se someten a secuenciación rápida para determinar por lo menos una parte de la secuencia de nucleótidos, comprendiendo por lo menos la (s) secuencia (s) identificador (as) , de las sondas ligadas.
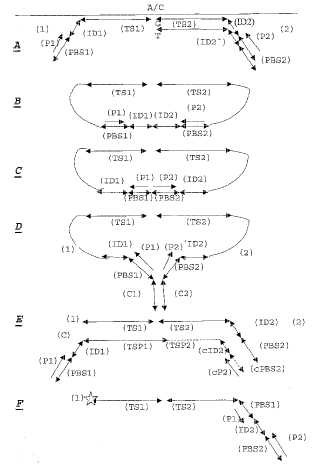
Secuenciación unidireccional o bidireccional por síntesis Extensión específica del alelo antes de la ligación Sujeción
Grupo de bloqueo (opcional)
Zona de fijación Zona de fijación
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]