Sondas, bibliotecas y kits para análisis de mezclas de ácidos nucleicos y procedimientos para construirlos.
Una biblioteca de sondas oligonucleotídicas para un transcriptoma dado en la que cada sonda en la bibliotecaconsiste en una marca de la secuencia de reconocimiento que tiene una longitud de 6 a 12 nucleótidos y unresto de detección en el que al menos un monómero en cada sonda oligonucleotídica es un análogo delmonómero modificado,
lo que incrementa la afinidad de unión por la secuencia diana complementariarespecto al correspondiente oligodesoxirribonucleótido sin modificar, de modo que las sondas de la bibliotecatienen suficiente estabilidad para la unión específica de secuencia y la detección de al menos un 70 %,preferentemente al menos un 90 % de todos los ácidos nucleicos diana diferentes en un transcriptoma de unaespecie y en la que el número de diferentes secuencias de reconocimiento comprende menos del 10 % detodas las marcas de secuencia posibles de una(s) longitud(es) dada(s).
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/DK2004/000429.
Solicitante: EXIQON A/S.
Nacionalidad solicitante: Dinamarca.
Dirección: SKELSTEDET 16 2950 VEDBAEK DINAMARCA.
Inventor/es: RAMSING,NIELS B, MOURITZEN,Peter, ECHWALD,Sören Morgenthaler, TOLSTRUP,Niels.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07H21/04 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con desoxirribosilo como radical sacárido.
- C12Q1/68 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
- G06F19/00
PDF original: ES-2385828_T3.pdf
Fragmento de la descripción:
Sondas, bibliotecas y kits para análisis de mezclas de ácidos nucleicos y procedimientos para construirlos Campo de la invención La invención se refiere a sondas de ácido nucleico, bibliotecas de sondas de ácido nucleico y kits para detectar, clasificar o cuantificar componentes en una mezcla compleja de ácidos nucleicos, tal como un transcriptoma, y procedimientos de usarlos.
Antecedentes de la invención Con el advenimiento de las micromatrices para realizar el perfil de expresión de miles de genes, tales como matrices GeneChip™ (Affymetrix, Inc., Santa Clara, CA) se pueden identificar las correlaciones entre genes expresados y fenotipos celulares en una fracción con los costes y trabajo necesarios para los procedimientos tradicionales, tales como transferencia Northern o análisis de transferencia de puntos. Las micromatrices permiten el desarrollo de múltiples ensayos paralelos para identificar y validar biomarcadores de enfermedad y dianas farmacológicas que se pueden usar en el diagnóstico y tratamiento. También se pueden usar perfiles de expresión génica para estimar y predecir las consecuencias metabólicas y toxicológicas de la exposición a un agente (p. ej., tal como un fármaco, una posible toxina o carcinógeno etc.) o una condición (p. ej., temperatura, pH etc.) .
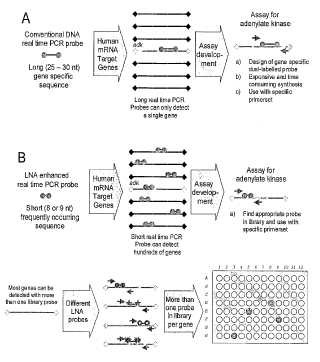
Los experimentos con micromatrices a menudo proporcionan datos redundantes, solo una fracción de los cuales tiene valor para el investigador. Adicionalmente, dado el formato muy paralelo de los ensayos basados en micromatrices, las condiciones pueden no ser óptimas para sondas de captura individuales. Por estos motivos, a los experimentos con micromatrices les suelen seguir, o suelen ser sustituidos por ellos, estudios confirmatorios usando ensayos homogéneos con un único gen, Estos son, con mayor frecuencia, procedimientos basados en PCR cuantitativa tal como el ensayo con 5’ nucleasa u otros tipos de ensayos cuantitativos con sondas con marcaje doble. No obstante, estos ensayos siguen requiriendo tiempo y son ensayos de una sola reacción que están dificultados por costes elevados y procedimientos de diseño de sondas que requieren mucho tiempo. Además, las sondas para el ensayo de 5' nucleasa son relativamente largas (p. ej., de 15-30 nucleótidos) . Por tanto, las limitaciones en los sistemas de ensayo homogéneo que se conocen actualmente crean un cuello de botella en la validación de los hallazgos de micromatrices y en los procedimientos de validación de dianas enfocadas.
Un enfoque para evitar este cuello de botella es omitir las caras sondas indicadoras con marcaje doble usadas en los procedimientos del ensayo de la 5’ nucleasa y balizas moleculares y, en su lugar, usar pigmentos que se intercalan en el ADN no específicos de secuencia, tales como el Verde SYBR que brilla cuando se une al ADN bicatenario pero no al monocatenario. Usando dichos pigmentos es posible detectar de forma universal cualquier secuencia amplificada en tiempo real. No obstante, esta tecnología se ve dificultada por varios problemas. Por ejemplo, el cebado inespecífico durante el procedimiento de amplificación por PCR puede generar amplicones no diana accidentales que contribuirán al proceso de cuantificación. Además, son frecuentes las interacciones entre los cebadores para PCR en la reacción que forman “cebador-dímeros". Debido a la elevada concentración de cebadores de uso habitual en una reacción de PCR, esto puede conducir a cantidades significativas de amplicones no diana bicatenarios cortos a los que también se unen los pigmentos de intercalación. Por tanto, el procedimiento preferido de cuantificación de ARNm mediante PCR en tiempo real usa sondas de detección específicas de secuencia.
Un enfoque para evitar el problema de la amplificación aleatoria y la formación de cebador-dímeros es usar sondas de detección genéricas que se pueden usar para detectar un gran número de tipos diferentes de moléculas de ácido nucleico, mientras que la retención de alguna especificidad de secuencia ha sido descrita por Simeonov, y col. (Nucleic Acid Research 30 (17) : 91, 2002; U.S. Patent Publication 20020197630) e implica el uso de una biblioteca de sondas que comprende más del 10 % de todas las posibles secuencias de una longitud (o longitudes) dada. La biblioteca puede incluir varias nucleobases no naturales y otras modificaciones para estabilizar la unión de sondas/cebadorres en la biblioteca a una secuencia diana. Incluso en este caso, para la mayoría de las secuencias se requiere una longitud mínima de al menos 8 bases para alcanzar un grado de estabilidad que sea compatible con la mayoría de las condiciones de ensayo relevantes para aplicaciones tales como la PCR en tiempo real. Dado que una biblioteca universal de todos los posibles octámeros contiene 65.536 secuencias diferentes, incluso la biblioteca más pequeña considerada anteriormente por Simeonov, y col. contiene más del 10 % de todas las posibilidades, es decir al menos 6554 secuencias, cuya manipulación no es práctica y es muy caro de construir.
Desde un punto de vista práctico, varios factores limitan la facilidad de uso y la accesibilidad de las aplicaciones de ensayos homogéneos contemporáneos. Los problemas que se encuentran los usuarios de las tecnologías de ensayo convencionales incluyen:
• costes prohibitivamente elevados al intentar detectar muchos genes diferentes en pocas muestras, ya que el precio de adquisición de una sonda para cada tránscrito es alto.
• la síntesis de sondas marcadas requiere tiempo y el tiempo desde el pedido hasta la recepción desde el fabricante suele ser superior a una semana.
• Los kits diseñados pueden no funcionar la primera vez y los kits validados son caros por ensayo. 2
• Es difícil analizar rápidamente una diana nueva o mejora iterativamente un diseño de una sonda.
• La secuencia exacta de la sonda de las sondas validadas comerciales puede ser desconocida para el cliente, lo que da lugar a problemas con la evaluación de los resultados y la idoneidad para publicación científica.
• Cuando las condiciones o los componentes de los ensayos son oscuros, puede ser imposible pedir reactivos de una fuente alternativa.
La invención descrita aborda estos problemas prácticos y está dirigida a asegurar un desarrollo de ensayo rápido y barato de ensayos precisos y específicos para la cuantificación de tránscritos génicos.
Sumario de la invención Es deseable poder cuantificar la expresión de la mayoría de los genes (p. ej., > 98 %) en, por ejemplo, el transcriptoma humano, usando un número limitado de sondas de detección oligonucleotídicas en un sistema de ensayo homogéneo. La presente invención resuelve los problemas con los que se encuentran los enfoque contemporáneos con los ensayos homogéneos destacados anteriormente proporcionando un procedimiento para la construcción de multi-sondas genéricas con suficiente especificidad de secuencia, de modo que es improbable que detecten un fragmento de secuencia amplificada alatoriamente o cebador-dímeros, pero todavía son capaces de detectar muchas secuencias diana diferentes cada una. Dichas sondas se pueden usar en diferentes ensayos y se pueden combinar en pequeñas bibliotecas de sondas (50 a 500 sondas) que se pueden usar para detectar y/o cuantificar componentes individuales en mezclas complejas compuestas por miles de ácidos nucleicos diferentes (p. ej., detección de tránscritos individuales en el transcriptoma humano compuesto por > 30.000 ácidos nucleicos diferentes) cuando se combinan con un conjunto de cebadores específicos de diana.
Cada multisonda comprende dos elementos: 1) un elemento de detección o resto de detección que consiste en uno o más marcadores parta detectar la unión de la sonda a la diana; y 2) un elementos de reconocimiento o marcador de secuencia de reconocimiento que garantiza la unión a la (s) diana (s) específica (s) de interés. El elemento de detección puede ser cualquiera de varios principios de detección usados en ensayos homogéneos. La detección de la unión es directa mediante un cambio mensurable en las propiedades de uno o más de los marcadores tras la unión a la diana (p. ej., un ensayo de tipo baliza molecular con o sin estructura troncal) o indirecta mediante una reacción posterior tras la unión (p. ej. escisión meciante actividad 5' nucleasa de la ADN polimerasa en ensayos de 5' nucleasa) .
El elemento de reconocimiento es un nuevo componente de la presente invención. Comprende un resto oligonucleotídico corto cuya secuencia se ha seleccionado para permitir la detección de una gran suboblación de nucleótidos... [Seguir leyendo]
Reivindicaciones:
1. Una biblioteca de sondas oligonucleotídicas para un transcriptoma dado en la que cada sonda en la biblioteca consiste en una marca de la secuencia de reconocimiento que tiene una longitud de 6 a 12 nucleótidos y un resto de detección en el que al menos un monómero en cada sonda oligonucleotídica es un análogo del monómero modificado, lo que incrementa la afinidad de unión por la secuencia diana complementaria respecto al correspondiente oligodesoxirribonucleótido sin modificar, de modo que las sondas de la biblioteca tienen suficiente estabilidad para la unión específica de secuencia y la detección de al menos un 70 %, preferentemente al menos un 90 % de todos los ácidos nucleicos diana diferentes en un transcriptoma de una especie y en la que el número de diferentes secuencias de reconocimiento comprende menos del 10 % de todas las marcas de secuencia posibles de una (s) longitud (es) dada (s) .
2. La biblioteca de acuerdo con la reivindicación 1, en la que las sondas de la biblioteca tienen una estabilidad suficiente para la unión específica de secuencia y la detección de al menos el 70 % de todos los ácidos nucleicos diana diferentes en cualquier transcriptoma eucariótico diana.
3. La biblioteca de acuerdo con la reivindicación 2, en la que el transcriptoma eucariótico está seleccionado del 15 grupo compuesto por un transcriptoma de hongos, de plantas y de un animal, tal como un mamífero.
4. La biblioteca de acuerdo con la reivindicación 1, en la que las sondas de la biblioteca tienen una estabilidad suficiente para la unión específica de secuencia y la detección de al menos el 90 % de todos los ácidos nucleicos diana diferentes en el transcriptoma de una especie seleccionada de un ser humano, un ratón, una rata y un mono.
5. Una biblioteca de sondas oligonucleotídicas de acuerdo con cualquiera de las reivindicaciones precedentes, en la que el segmento marca de la secuencia de reconocimiento de las sondas en la biblioteca se ha modificado de al menos uno de los modos siguientes:
i) sustitución con al menos un nucleótido no natural ii) sustitución con al menos un resto químico para incrementar la estabilidad de la sonda.
6. Una biblioteca de sondas oligonucleotídicas de acuerdo con la reivindicación 1 o 5, en la que la marca de la secuencia de reconocimiento tiene una longitud de 8 o 9 nucleótidos.
7. Una biblioteca de sondas oligonucleotídicas de acuerdo con la reivindicación 6, en la que las marcas de la secuencia de reconocimiento están sustituidas por nucleótidos LNA.
8. Una biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones
precedentes, en la que más del 90 % de las sondas oligonucleotídicas se pueden unir y detectar al menos dos secuencias diana en una población de ácido nucleico.
9. Una biblioteca de acuerdo con la reivindicación 8, en la que la marca de la secuencia de reconocimiento es complementaria a al menos dos secuencias diana en la población de ácido nucleico.
10. Una biblioteca de sondas oligonucleotídicas de 8 y 9 nucleótidos de longitud, que comprende una mezcla de
subconjuntos de sondas oligonucleotídicas definidas en una cualquiera de las reivindicaciones 1-9 de modo que las sondas oligonucleotídicas tienen una estabilidad suficiente para la unión específica de secuencia y la detección de al menos el 70 % de todos los ácidos nucleicos diana en un transcriptoma.
11. Una biblioteca de sondas oligonucleotídicas de una cualquiera de las reivindicaciones precedentes, en la que el número de diferentes secuencias diana en una población de ácido nucleico es al menos 100.
12. Una biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que al menos un nucleótido en cada sonda oligonucleotídica está sustituido con un análogo nucleotídico no natural, un análogo de desoxirribosa o de ribosa o un enlace internucleotídico distinto a un enlace fosfodiéster.
13. Una biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones 45 precedentes, en la que el resto de detección es un ligando de unión al surco menor unido de forma covalente o no covalente o un intercalante seleccionado del grupo que comprende pigmentos de cianina asimétricos, DAPI, SYBR Verde I, SYBR Verde II, SYBR Oro, PicoGreen, naranja tiazol, Hoechst 33342, bromuro de etidio, 1-O- (1-pirenilmetil) glicerol y Hoechst 33258.
14. La biblioteca de sondas oligonucleotídicas de acuerdo con la reivindicación 12 o 13, en la que el enlace 50 internucleotídico distinto al enlace fosfodiéster es un enlace internucleotídico sn fosfato.
15. La biblioteca de sondas oligonucleotídicas de acuerdo con la reivindicación 14, en la que el enlace internucleotídico está seleccionado del grupo que consiste en fosfonato de alquilo, fosforoamidita, alquilfosfotriéster, fosforotioato y enlaces fosforoditioato.
16. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que dichas sondas oligonucleotídicas contienen nucleótidos no naturales, tales como 2’-Ometilo, diaminapurina, 2-tiouracilo, 5-nitroindol, bases universales o degeneradas, ácidos nucleicos intercalantes o ligandos de unión a surco menor, para potenciar su unión a una secuencia de ácido nucleico complementaria.
17. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que dichas secuencias de reconocimiento diferentes comprenden menos del 1 % de todos los posibles oligonucleótidos de una longitud dada.
18. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones
precedentes, en la que cada sonda se puede detectar usando una marca doble mediante el principio de ensayo de baliza molecular.
19. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones 1-17, en la que cada sonda se puede detectar usando una marca doble mediante el principio de ensayo de 5’ nucleasa.
20. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones
precedentes, en la que cada sonda contiene un único resto de detección que se puede detectar mediante el principio de ensayo de baliza molecular.
21. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que la población de ácido nucleico diana es una muestra de ARNm, una muestra de ADNc
o una muestra de ADN genómico.
22. La biblioteca de sondas oligonucleotídicas de acuerdo con la reivindicación 21, en la que dicha población de ARNm diana o de ADNc diana se origina de los transcriptomas de ser humano, ratón o rata.
23. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que dichas secuencias diana de la sonda se producen al menos una vez dentro de más del 4 % de diferentes ácidos nucleico diana en una población de ácido nucleico diana.
24. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que las secuencias de la sonda autocomplementarias se han omitido de dicha biblioteca.
25. La biblioteca de sondas oligonucleotídicas de acuerdo con la reivindicación 24, en la que se ha eliminado la selección de dichas secuencias autocomplementarias.
26. La biblioteca de sondas oligonucleotídicas de acuerdo con la reivindicación 24, en la que sdichas secuencias
autocomplementarias se han eliminado mediante modificaciones específicas de secuencia, tal como nucleótidos no estándar, nucleótidos con nucleobases SBC, 2’-O-metilo, diamina purina, 2-tio uracilo, bases universales o degeneradas o ligandos de unión al surco menor.
27. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que la temperatura de fusión Tm de cada sonda está ajustada para que sea adecuada para ensayos basados en PCR mediante sustitución con modificaciones no naturales, tales como LNA, modificado opcionalmente con nucleobases de SBC, 2’-O-metilo, diamina purina, 2-tio-uracilo, 5-nitroindol, bases universales o degeneradas, ácidos nucleicos intercalantes o ligandos de unión a surco menor, para potenciar su unión a una secuencia de ácido nucleico complementaria.
28. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones 40 precedentes, en la que la temperatura de fusión (Tm) de cada sonda es al menos 50 ºC.
29. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que cada sonda tiene un nucleótido de ADN en el extremo 5’.
30. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que cada sonda contiene un par de fluoróforo-inactivador para detección.
45 31. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que cada sonda se puede detectar mediante el principio de ensayo de baliza molecular.
32. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que cada sonda está unida a un fluoróforo intercalante o ligando de unión a surco menor, que, tras la unión a un ADN bicatenario o a un heterodúplex de ADN-ARN diana, altera la fluorescencia.
50 33. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones precedentes, en la que la población diana es el transcriptoma humano.
34. La biblioteca de sondas oligonucleotídicas de acuerdo con una cualquiera de las reivindicaciones
precedentes, en la que cada sonda oligonucleotídica detecta el mayor número posible de diferentes ácidos nucleicos diana resultante en una cobertura máxima para una población de ácido nucleico diana mediante dicha biblioteca.
35. La biblioteca de sondas oligonucleotídicas en la TABLA Ia capaz de detectar las secuencias complementarias 5 en cualquier población de ácidos nucleicos dada.
36. La biblioteca de acuerdo con una cualquiera de las reivindicaciones precedentes, que comprende sondas, cada una ellas con un elemento de reconocimiento enumerado en la TABLA Ia en la memoria y/o que comprende sondas, teniendo cada una un elemento de reconocimiento complementario a los elementos de reconocimiento enumerado en la TABLA 1.
37. Un procedimiento de seleccionar secuencias oligonucleotídicas útiles en la biblioteca de acuerdo con una cualquiera de las reivindicaciones precedentes, que comprende a) proporcionar una primera lista de todos los oligonucleótidos posibles de un número predefinido de nucleótidos, N, teniendo dichos oligonucleótidos una temperatura de fusión, Tm, de al menos 50 ºC.
b) proporcionar una segunda lista de secuencias de ácido nucleico, c) identificar y almacenar para cada miembro de dicha primera lista, el número de miembros de dicha segunda lista, que incluye una secuencia complementaria de cada miembro citado, d) seleccionar un miembro de dicha primera lista, que en la identificación en la etapa c coincide con el número máximo, identificado en la etapa c, de miembros de dicha segunda lista, e) añadir el miembro seleccionado en la etapa d a una tercera lista consistente en los oligonucleótidos 20 seleccionados útiles en la biblioteca de acuerdo con una cualquiera de las reivindicaciones precedentes.
f) restar el miembro seleccionado en la etapa d de dicha primera lista para proporcionar una primera lista revisada, m) repetir las etapas de f hasta que dicha tercera lista consiste en miembros que, juntos, serán complementarios de al menos el 70 % de todos los ácidos nucleicos diana diferentes en el transcriptoma 25 dada o de al menos 90 % de todos los ácidos nucleicos diana diferentes en un transcriptoma de una especie.
38. El procedimiento de acuerdo con la reivindicación 37, en el que la Tm es al menos 60 ºC.
39. El procedimiento de acuerdo con cualquiera de las reivindicaciones 37 o 38, en el que la primera lista de oligonucleótidos solo incluye oligonucleótidos incapaces de autohibridación.
40. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 39, que, después de la etapa f y antes de la etapa m, comprende las etapas siguientes:
g) restar todos los miembros de dicha segunda lista, que incluye una secuencia complementaria al miembro seleccionado en la etapa d para obtener una segunda lista revisada, h) identificar y almacenar para cada miembro de dicha primera lista revisada, el número de miembros de 35 dicha segunda lista revisada, que incluye una secuencia complementaria de cada miembro citado, i) seleccionar un miembro de dicha primera lista, que en la identificación en la etapa h coincide con el número máximo, identificado en la etapa h, de miembros de dicha segunda lista, o seleccionar un miembro de dicha primera lista proporciona el número máximo obtenido multiplicando el número identificado en la etapa h con el número identificado en la etapa c, j) añadir el miembro seleccionado en la etapa i a dicha tercera lista, k) restar del miembro seleccionado en la etapa i de dicha primera lista revisada, y j) restar todos los miembros de dicha segunda lista revisada, que incluye una secuencia complementaria al miembro seleccionado en la etapa i.
41. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 40, en el que la repetición en la 45 etapa m continua hasta que dicha tercera lista consiste en miembros que, juntos, serán complementarios a al menos 85 % de los miembros de la lista de las secuencias de ácido nucleico diana de la etapa b.
42. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 41, en el que, tras la selección del primer miembro de dicha tercera lista, la selección en la etapa d después de la etapa c está precedida por la identificación de los miembros de dicha primera lista que hibrida con más de un porcentaje seleccionado del 50 número máximo de miembros de dicha segunda lista, de modo que solo dichos miembros identificados de
este modo se someten a la selección en la etapa d.
43. El procedimiento de acuerdo con la reivindicación 42, en el que el porcentaje seleccionado es 80 %.
44. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 43, en el que N es un número entero seleccionado de 6, 7, 8, 9, 10, 11 y 12.
45. El procedimiento de acuerdo con la reivindicación 46, en el que N es 8 o 9.
46. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 47, en el que dicha segunda lista de la etapa b comprende secuencias de ácido nucleico diana como se definen en la reivindicación 21 o 22.
47. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 46, realizado esencialmente como se establece en la Fig. 2.
48. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 47, en la que dichas primera, segunda y tercera listas se almacenan en la memoria de un sistema informático, preferentemente en una base de datos.
49. Un programa informático que proporciona instrucciones para implementar el procedimiento de acuerdo con una cualquiera de las reivindicacione.
3. 48 incorporadas en un medio legible por ordenador.
50. Un sistema que comprende una base de datos de secuencias diana y un programa de aplicación para ejecutar el programa informático de la reivindicación 49.
51. Un procedimiento para identificar una sonda para la detección de y cebadores que amplificarán, un ácido nucleico diana, en el que el procedimiento comprende A) introducir, en un sistema informático, datos que identifican de forma única la secuencia de ácido nucleico de dicho ácido nucleico diana, en el que dicho sistema informático comprende una base de datos que contiene información de la composición de al menos una biblioteca de sondas de ácido nucleico de acuerdo con una cualquiera de las reivindicaciones 1-36, y en el que el sistema informático comprende además una base de datos de secuencias de ácido nucleico diana para cada sonda de dicha al menos una biblioteca y/o comprende además medios para adquirir y comparar datos de las secuencias de ácido nucleico, B) Identificar, en el sistema informático, una sonda de al menos una biblioteca, en el que la secuencia de la sonda existe en la secuencia del ácido nucleido diana o una secuencia complementaria a la secuencia del ácido nucleico diana C) Identificar, en el sistema informático, el cebador que amplificará la secuencia del ácido nucleico diana D) proporcionar, como identificación de un medio específico para la detección, una salida que destaca la 30 sonda identificada en la etapa B y las secuencias de los cebadores identificadas en le etapa C.
52. El procedimiento de acuerdo con la reivindicación 51, en el que la etapa A comprende también introducir en el sistema informático los datos que identifica la al menos una biblioteca de ácidos nucleicos a partir de la cual se desea seleccionar un miembro para usar en el medio de detección específico.
53. El procedimiento de acuerdo con la reivindicación 52, en el que el dato que identifica la composición de al 35 menos una biblioteca es un código de producto.
54. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
5. 53, en el que la entrada en la etapa A se realiza a través de una interfaz de web en Internet.
55. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
5. 54, en el que los cebadores
identificados en la etapa C se escogen de un modo tal que se minimiza la posibilidad de amplificar ácidos 40 nucleicos genómicos en una reacción de PCR.
56. El procedimiento de acuerdo la reivindicación 55, en el que al menos uno de los cebadores se selecciona de modo que induzca una secuencia de nucleótidos que en el ADN genómico está interrumpida por un intrón,
57. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
5. 56, en el que los cebadores
seleccionados en la etapa C se escogen de un modo tal que se minimiza la longitud de amplicones obtenidos 45 de PCR realizada en la secuencia de ácido nucleico diana.
58. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
5. 57, en el que los cebadores seleccionados en la etapa C se escogen de un modo tal que se optimiza el contenido en GC para realizar la PCR.
59. Un programa informático que proporciona instrucciones para implementar el procedimiento de acuerdo con 50 una cualquiera de las reivindicacione.
5. 58 incorporadas en un medio legible por ordenador.
60. Un sistema que comprende una base de datos de sondas de ácido nucleico como se definen en una cualquiera de las reivindicaciones 1-36 y un programa de aplicación para ejecutar el programa informático de la reivindicación 59.
61. Un procedimiento para realizar una pluralidad de secuencias diana, que comprende poner en contacto una
muestra de secuencias diana con una biblioteca de acuertdo con una cualquiera de las reivindicaciones 1-36 y detectar, caracterizar o cuantificar las secuencias de la sonda que se unen a las secuencias diana.
62. El procedimiento de acuerdo con la reivindicación 61, que proporciona la detección de una secuencia de ácico nucleico que está presente en menos del 10 % de la pluralidad de secuencias que están unidas por las secuencias multi-sondas.
63. El procedimiento de acuerdo con la reivindicación 62, en el que las secuencias de ARNm diana o las secuencias de ADNc comprenden un transcriptoma.
64. El procedimiento de acuerdo con la reivindicación 63, en el que el transcriptoma es un transcriptoma humano.
65. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
6. 64, en el que la biblioteca de sondas está acoplada covalentemente a un soporte sólido.
66. El procedimiento de acuerdo con la reivindicación 65, en el que el soporte sólido comprende una placa de microtitulación y cada pocillo de la placa de microtitulación comprende una sonda de la biblioteca diferente.
67. El procedimiento de acuerdo con una cualquiera de las reivindicacione.
6. 66, en el que la etapa de detectar se realiza amplificando una secuencia de ácido nucleico diana que contiene una secuencia de reconocimiento complementaria a una sonda de la biblioteca.
68. El procedimiento de acuerdo con la reivindicación 67, en el que la amplificación del ácido nucleico diana se lleva a cabo usando un par de sondas oligonucleotídicas que flanquean la secuencia de reconocimiento complementaria a una sonda de la biblioteca.
69. El procedimiento de acuerdo con la reivindicació.
6. 68, en el que la presencia o el nivel de expresión de una o más secuencias de ácido nucleico diana se correlaciona con el fenotipo de una especie.
70. El procedimiento de la reivindicación 69, en el que el fenotipo es una enfermedad.
71. Un procedimiento de análisis de una mezcla de ácidos nucleicos que usa una biblioteca de acuerdo con una cualquiera de las reivindicaciones 1-36 que comprende las etapas de (a) poner en contacto un ácido nucleico diana con una biblioteca de sondas oligonucleotídicas marcadas, teniendo cada una de dichas sondas oligonucleotíticas una secuencia conocida y estando unidas a un soporte sólido en una posición conocida, para hibridar dicho ácido nucleico diana con al menos un miembro de dicha biblioteca de sondas, de modo que se forma una biblioteca hibridada;
(b) o bien (i) poner en contacto dicha biblioteca hibridada con una nucleasa capaz de escindir los oligonucleótidos bicatenarios para liberar de dicha biblioteca hibridada una porción de dichas sondas oligonucleotídicas marcadas o fragmentos de las mismas; o (ii) identificar dichas posiciones de dicha biblioteca hibridada en la que las sondas marcadas o fragmentos de las mismas que han hibridado, para determinar la secuencia de dicho ácido nucleico diana; y (c) identificar dichas posiciones de dicha biblioteca hibridada de la que se han eliminado las sondas marcadas, o fragmentos de las mismas, para determinar la secuencia de dicho ácido nucleico diana sin marcar.
Figura 17 (cont.)
66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149
Figura 17 (cont.)
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]