AMPLIFICACIÓN DE FRAGMENTOS DE ADN.
Un procedimiento para la amplificación selectiva de una muestra de una molécula de ácido nucleico que tiene una secuencia diana adyacente a un extremo 3',
comprendiendo el procedimiento (i) poner en contacto la muestra con un oligonucleótido molde que tiene (a) una región 3' sustancialmente complementaria a la secuencia diana adyacente al extremo 3' de una molécula de ácido nucleico; (b) una cola 5' que comprende una secuencia de ácido nucleico tal que se forma una cola 5' libre cuando el oligonucleótido molde hibrida con la secuencia diana localizada adyacente a un extremo 3', proporcionando la cola 5' un molde para la extensión del extremo 3' de la secuencia diana que incorpora la secuencia complementaria a la cola 5' del oligonucleótido molde provocando la adición de la secuencia complementaria a la cola 5' a la secuencia diana; y (c) una modificación en la región 3' que retarda la extensión 3' de dicho oligonucleótido molde; (ii) poner en contacto la muestra con un segundo oligonucleótido para cebar en una dirección inversa el oligonucleótido molde, y opcionalmente un tercer oligonucleótido que comparte la secuencia de nucleótidos con la cola 5' del oligonucleótido molde; (iii) realizar la amplificación de la muestra en la que (a) la hibridación del oligonucleótido molde con la secuencia diana adyacente a un extremo 3' se estabiliza por copiado de la cola 5' del oligonucleótido molde por extensión desde el extremo 3' de la secuencia diana en presencia de extensión 3' retardada de dicho oligonucleótido; y (b) en la que la consecuente hibridación del oligonucleótido molde estabilizada con la secuencia diana adyacente a un extremo 3' potencia la eficacia de la extensión 3' del oligonucleótido molde, y (c) en la que la amplificación sucede usando el oligonucleótido molde y/o el tercer oligonucleótido en combinación con el segundo oligonucleótido, provocando la amplificación selectiva de la secuencia diana adyacente a un extremo 3'
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/AU2007/000389.
Solicitante: COMMONWEALTH SCIENTIFIC AND INDUSTRIAL RESEARCH ORGANISATION.
Nacionalidad solicitante: Australia.
Dirección: LIMESTONE AVENUE CAMPBELL, ACT 2612 AUSTRALIA.
Inventor/es: MOLLOY,PETER,LAURENCE, RAND,Keith.
Fecha de Publicación: .
Fecha Solicitud PCT: 28 de Marzo de 2007.
Clasificación Internacional de Patentes:
- C12Q1/68D2G
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia.
PDF original: ES-2357746_T3.pdf




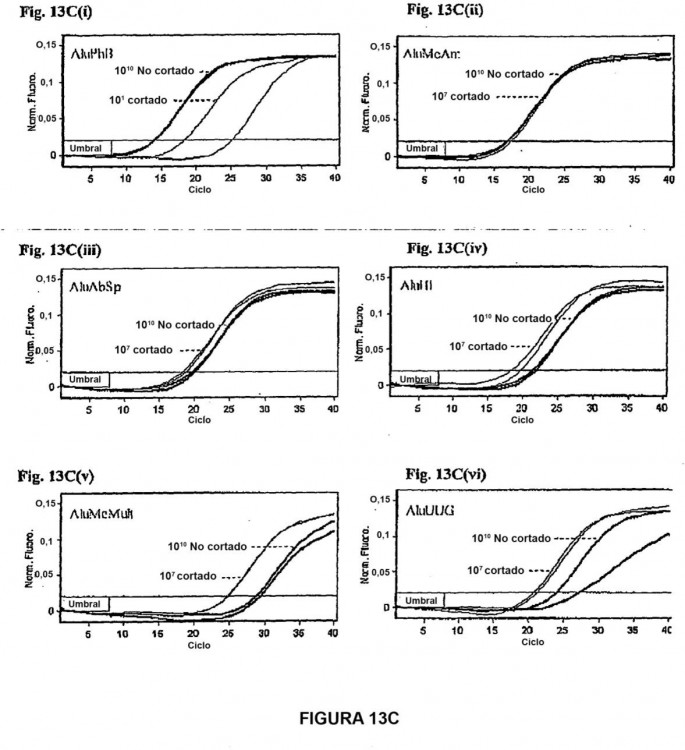
Fragmento de la descripción:
CAMPO DE LA INVENCIÓN
La invención se refiere a moléculas de ácido nucleico de diferenciación que tienen una secuencia específica adyacente a un extremo 3' de moléculas de ácido nucleico en las que la misma secuencia está incluida dentro de la molécula o está ausente. La invención también se refiere a moléculas de ácido nucleico de diferenciación que difieren 5 en la secuencia adyacente a un extremo 3'. En particular, la invención se refiere a un procedimiento para la amplificación selectiva de ADN escindido en presencia de ADN no escindido que comparte la misma secuencia diana.
ANTECEDENTES DE LA INVENCIÓN
La reacción en cadena de la polimerasa (PCR) se basa en ciclos repetidos de desnaturalización de ADN bicatenario, seguido de hibridación del cebador oligonucleotídico con el molde de ADN, y extensión de cebador por una 10 ADN polimerasa (por ejemplo, véase, Mullis y col. patentes de Estados Unidos Nº 4.683.195, 4.683.202 y 4.800.159). Los cebadores oligonucleotídicos usados en PCR están diseñados para hibridar con cadenas opuestas del ADN, y se colocan de tal modo que el producto de la extensión catalizada por la ADN polimerasa de un cebador pueda servir como cadena molde para el otro cebador. El procedimiento de amplificación por PCR produce el aumento exponencial de ADN discreto cuya longitud está definida por los extremos 5' de los cebadores oligonucleotídicos. 15
En su aplicación convencional, se eligen cebadores que aparean con secuencias dentro del genoma diana y flanquean la región de ADN que tiene que amplificarse. Además, se han descrito varias variaciones de PCR en las que se ligan engarces en los extremos de fragmentos de ADN y después las secuencias de los engarces se usan para cebar la amplificación del ADN. La PCR mediada por ligamiento se aplicó por primera vez para la producción de huella genética de ADN y reacciones de secuenciación donde los extremos de ADN se generaron por digestión con DNasa I o 20 escisión química (Mueller y Wold, 1989 y Pfeifer y col. 1989) y se extendieron hasta los extremos formados por digestión de restricción (Steigerwald y col. 1990). La especificidad se consigue combinando cebadores para una región diana específica con cebadores dirigidos al engarce añadido. Las variaciones de la técnica han encontrado una variedad de usos en la secuenciación de genomas y análisis de metilación del ADN, paseo cromosómico, identificación de sitios de integración o recombinación en el cromosoma, estudio de puntos de ruptura de mutaciones y extremos del ARNm. La 25 PCR mediada por ligamiento también puede usarse para la amplificación del genoma completo, donde se amplifica la población entera de moléculas con extremos ligados (Schumaker y col., 2006).
A pesar de la utilidad de gran alcance de la PCR mediada por ligamiento, existe una necesidad en curso de metodologías basadas en PCR mejoradas que permitan la amplificación selectiva de ADN que tengan extremos 3' y que proporcionen ventajas de simplicidad, especificidad y conveniencia. 30
SUMARIO DE LA INVENCIÓN
La presente invención proporciona procedimientos para diferenciar una molécula de ácido nucleico que tiene una secuencia específica adyacente a un extremo 3' de moléculas de ácido nucleico en las que está presente la misma secuencia diana pero incluida dentro de la molécula, o está ausente. Los extremos 3' pueden ser el resultado de, por ejemplo, escisión con enzimas de restricción u otra escisión enzimática o química específica, o los extremos pueden ser 35 los extremos libres de amplicones generados por PCR, los extremos libres de un ADN de origen natural tal como de un cromosoma, virus o fago, o los extremos pueden producirse por transcripción inversa a partir de un molde de ARN, o producirse por cualquier otro medio que genere ácidos nucleicos que tienen un extremo 3'.
De acuerdo con una realización de la invención, también mencionada en este documento como 'PCR específica de extremo' o ES-PCR, se utilizan las secuencias de nucleótidos localizadas adyacentes a un extremo 3' de 40 una molécula de ácido nucleico para cebar las reacciones de amplificación que proceden solamente cuando estas secuencias se encuentran adyacentes a un extremo 3', en lugar de cuando estas secuencias están incluidas dentro de las moléculas de ácido nucleico.
Para conseguir esta diferenciación, se utiliza un oligonucleótido (a partir de ahora en este documento "oligonucleótido molde") que tiene una parte 3' complementaria a la secuencia diana y una parte 5' que forma una cola 45 5' cuando el oligonucleótido hibrida con el ácido nucleico. Cuando el oligonucleótido molde hibrida con la secuencia diana localizada adyacente a un extremo 3', puede tener lugar el copiado de la cola 5' en presencia de una polimerasa adecuada por extensión del extremo 3' libre. Cuando el oligonucleótido molde hibrida con la secuencia diana que está incluida dentro de una molécula de ácido nucleico, no puede suceder el copiado de la cola 5', debido a la ausencia de un extremo 3' libre. El oligonucleótido actualmente descrito se menciona en este documento como oligonucleótido molde 50 debido a su cola 5' que sirve como molde para la extensión 3' de la secuencia diana localizada adyacente a un extremo 3'.
El copiado de la cola 5' cuando la secuencia diana aparece adyacente a un extremo 3' provoca la adición de una nueva secuencia en el extremo 3' de la molécula de ácido nucleico marcada como diana, que puede utilizarse en una posterior amplificación selectiva de moléculas de ácido nucleico que tienen la secuencia diana adyacente al 55 extremo 3'.
Por ejemplo, cuando un extremo 3' es un producto de escisión con enzimas de restricción, sucederá la amplificación selectiva del ADN escindido sobre el ADN no escindido.
En una realización, el oligonucleótido molde incorpora una modificación que retarda la extensión 3' del oligonucleótido, mientras el copiado de su cola 5' por extensión de la secuencia diana procede sin impedimentos. Por consiguiente, la secuencia de ácido nucleico de la región 3' del oligonucleótido molde puede elegirse de modo que la 5 hibridación de la secuencia diana sea inestable en las condiciones usadas para que en ausencia de copiado de la cola 5', se inhiba la hibridación del oligonucleótido molde con la secuencia diana incluida dentro de una molécula de ácido nucleico. El copiado de la cola 5' potencia la hibridación del oligonucleótido molde con la secuencia diana adyacente a un extremo 3' en comparación con la hibridación del oligonucleótido molde con la secuencia diana incluida dentro de una molécula de ácido nucleico, en la que no sucede el copiado de la cola 5'. 10
La hibridación potenciada del oligonucleótido molde a su vez aumenta la eficacia con la que sucede la extensión 3' del oligonucleótido molde, posibilitando de este modo que proceda la amplificación de la secuencia diana, ya sea en combinación con otro oligonucleótido molde que ceba en la dirección inversa, o con un cebador inverso complementario a secuencias en otras partes dentro de la molécula de ácido nucleico marcada como diana, para permitir la detección o amplificación específica de la molécula deseada, es decir, la que tiene la secuencia diana 15 adyacente a un extremo 3'.
La secuencia recién incorporada en el extremo 3' de una molécula de ácido nucleico marcada como diana (por copiado de la cola 5' del oligonucleótido) también puede utilizarse para amplificar la molécula de ácido nucleico marcada como diana.
Por lo tanto, en una realización de la invención, se incuba una muestra con un oligonucleótido molde cuya parte 20 3' contiene secuencias sustancialmente complementarias a la secuencia diana adyacente al extremo 3' de la molécula de ácido nucleico marcada como diana, y formando la parte 5' una cola 5'. El oligonucleótido molde incorpora una modificación que retarda la extensión desde su extremo 3'. En presencia de una polimerasa adecuada, la cola 5' del oligonucleótido molde se copia por extensión de la secuencia diana a partir de su extremo 3'. Como resultado de la extensión 3' de la secuencia diana, la hibridación del oligonucleótido molde se potencia en comparación con su 25 hibridación a la secuencia diana incluida dentro de una molécula de ácido nucleico, promoviendo por lo tanto la extensión 3' del oligonucleótido molde cuando está hibridado con la secuencia diana adyacente a un extremo 3'. El propio oligonucleótido molde o la secuencia recién incorporada (mencionada... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento para la amplificación selectiva de una muestra de una molécula de ácido nucleico que tiene una secuencia diana adyacente a un extremo 3', comprendiendo el procedimiento
(i) poner en contacto la muestra con un oligonucleótido molde que tiene
(a) una región 3' sustancialmente complementaria a la secuencia diana adyacente al extremo 3' de 5 una molécula de ácido nucleico;
(b) una cola 5' que comprende una secuencia de ácido nucleico tal que se forma una cola 5' libre cuando el oligonucleótido molde hibrida con la secuencia diana localizada adyacente a un extremo 3', proporcionando la cola 5' un molde para la extensión del extremo 3' de la secuencia diana que incorpora la secuencia complementaria a la cola 5' del oligonucleótido molde provocando la adición de la secuencia 10 complementaria a la cola 5' a la secuencia diana; y
(c) una modificación en la región 3' que retarda la extensión 3' de dicho oligonucleótido molde;
(ii) poner en contacto la muestra con un segundo oligonucleótido para cebar en una dirección inversa el oligonucleótido molde, y opcionalmente un tercer oligonucleótido que comparte la secuencia de nucleótidos con la cola 5' del oligonucleótido molde; 15
(iii) realizar la amplificación de la muestra en la que
(a) la hibridación del oligonucleótido molde con la secuencia diana adyacente a un extremo 3' se estabiliza por copiado de la cola 5' del oligonucleótido molde por extensión desde el extremo 3' de la secuencia diana en presencia de extensión 3' retardada de dicho oligonucleótido; y
(b) en la que la consecuente hibridación del oligonucleótido molde estabilizada con la secuencia diana 20 adyacente a un extremo 3' potencia la eficacia de la extensión 3' del oligonucleótido molde, y
(c) en la que la amplificación sucede usando el oligonucleótido molde y/o el tercer oligonucleótido en combinación con el segundo oligonucleótido, provocando la amplificación selectiva de la secuencia diana adyacente a un extremo 3'.
2. El procedimiento de la reivindicación 1, en el que la amplificación selectiva es de una muestra de una molécula 25 de ácido nucleico que tiene una secuencia diana adyacente a un extremo 3' en presencia de moléculas que comprenden la secuencia diana incluida dentro de la molécula, donde la hibridación del oligonucleótido molde con la secuencia diana adyacente a un extremo 3' está estabilizada en comparación con la hibridación de dicho oligonucleótido con la secuencia diana incluida dentro de la molécula de modo que la eficacia de la extensión 3' del oligonucleótido molde cuando hibrida con la secuencia diana adyacente a un extremo 3' está potenciada en comparación con la 30 extensión del oligonucleótido molde hibridado con la secuencia diana incluida dentro de la molécula.
3. El procedimiento de la reivindicación 1, en el que la amplificación selectiva es de una muestra de una molécula de ácido nucleico que tiene una secuencia diana adyacente a un extremo 3' en presencia de una población mixta de moléculas que tienen diferentes extremos 3', donde la hibridación del oligonucleótido molde con la secuencia diana adyacente a un extremo 3' está estabilizada en comparación con la hibridación de dicho oligonucleótido con una 35 secuencia no complementaria adyacente a un extremo 3' de modo que la eficacia de la extensión 3' del molde oligonucleotídico cuando hibrida con la secuencia diana adyacente a un extremo 3' está potenciada en comparación con la extensión del oligonucleótido molde hibridado con la secuencia no complementaria adyacente a un extremo 3'.
4. El procedimiento de la reivindicación 2 ó 3, en el que la molécula de ácido nucleico que tiene una secuencia diana adyacente a un extremo 3' es el resultado de la escisión de la molécula, donde la escisión se debe opcionalmente 40 a una endonucleasa de restricción específica de secuencia.
5. El procedimiento de acuerdo con una cualquiera de las reivindicaciones 1-4, en el que una modificación en la región 3' del oligonucleótido molde que retarda la extensión 3' del oligonucleótido se selecciona entre el grupo constituido por
(i) la incorporación de un desapareamiento de nucleótido 3' terminal, 45
(ii) la incorporación de uno o más desapareamientos de nucleótidos en la región 3' del oligonucleótido molde cerca del extremo 3',
(iii) la incorporación de una deleción en la región 3' del oligonucleótido molde cerca del extremo 3',
(iv) la incorporación de una inserción en la región 3' del oligonucleótido molde cerca del extremo 3', y
(v) cualquier combinación de modificaciones (i)-(iv). 50
6. Un procedimiento para la amplificación selectiva de una muestra de una molécula de ácido nucleico que tiene
una secuencia diana adyacente a un extremo 3', comprendiendo el procedimiento
(i) poner en contacto la muestra con un oligonucleótido molde que tiene
(a) una región 3' sustancialmente complementaria a la secuencia diana adyacente al extremo 3' a una molécula de ácido nucleico; y
(b) una cola 5' que comprende una secuencia de ácido nucleico tal que se forma una cola 5' libre 5 cuando el oligonucleótido molde hibrida con la secuencia diana localizada adyacente a un extremo 3', proporcionando la cola 5' un molde para la extensión del extremo 3' de la secuencia diana que incorpora la secuencia complementaria a la cola 5' del oligonucleótido molde provocando la adición de la secuencia complementaria a la cola 5' a la secuencia diana; y
(c) una modificación en la región 3' que bloquea la extensión 3' de dicho oligonucleótido molde, o una 10 modificación en la región de hibridación del oligonucleótido molde que permite la extensión 3' pero impide o bloquea el copiado en la región 3' de dicho oligonucleótido;
(ii) poner en contacto la muestra con un segundo oligonucleótido para el cebado en una dirección inversa del oligonucleótido molde;
(iii) poner en contacto la muestra con un tercer oligonucleótido que comparte la secuencia de nucleótidos con la 15 cola 5' del oligonucleótido molde y a partir del cual la extensión 3' procede sin impedimentos;
(iv) realizar la amplificación de la muestra en la que
(a) la hibridación del oligonucleótido molde con las moléculas de ácido nucleico en la muestra está seguida por el copiado de la cola 5' del oligonucleótido molde cuando el oligonucleótido molde hibrida con la secuencia diana adyacente a un extremo 3' pero no en presencia de extensión 3' bloqueada del oligonucleótido 20 molde, o en presencia de extensión 3' no impedida de un oligonucleótido molde a partir del cual el posterior copiado está impedido o bloqueado; y
(b) en la que la amplificación procede con el segundo y tercer oligonucleótidos, amplificando de forma selectiva la secuencia diana localizada adyacente a un extremo 3' debido a la cola 5' copiada.
7. El procedimiento de la reivindicación 6, en el que la amplificación selectiva es de una muestra de una molécula 25 de ácido nucleico que tiene una secuencia diana adyacente a un extremo 3' en presencia de moléculas que comprenden la secuencia diana incluida dentro de la molécula, donde el copiado de la cola 5' del oligonucleótido molde no sucede cuando el oligonucleótido molde hibrida con la secuencia diana incluida dentro de la molécula de modo que no procede la amplificación de la secuencia diana incluida dentro de la molécula.
8. El procedimiento de la reivindicación 6, en el que la amplificación selectiva es de una muestra de una molécula 30 de ácido nucleico que tiene una secuencia diana adyacente a un extremo 3' en presencia de una población mixta de extremos 3', donde el copiado de la cola 5' del oligonucleótido molde no sucede cuando el oligonucleótido molde hibrida con una secuencia no complementaria adyacente a un extremo 3', de modo que no procede la amplificación de la secuencia no complementaria adyacente a un extremo 3'.
9. El procedimiento de la reivindicación 7 u 8, en el que la molécula de ácido nucleico que tiene una secuencia 35 diana adyacente a un extremo 3' es el resultado de la escisión de la molécula, donde la escisión se debe opcionalmente a una endonucleasa de restricción específica de secuencia.
10. El procedimiento de acuerdo con una cualquiera de las reivindicaciones 6-9, en el que la modificación en la región 3' del oligonucleótido molde que bloquea la extensión 3' del oligonucleótido molde se selecciona entre el grupo que consiste en 40
(i) la incorporación de uno o más restos o análogos de nucleótidos no extensibles en su extremo 3',
(ii) la incorporación de una combinación de un resto o análogo de nucleótido no extensible 3' terminal y uno o más desapareamientos de nucleótidos en la región 3' del oligonucleótido molde cerca de su extremo 3',
(iii) la incorporación de uno o más sitios abásicos en la región 3' del oligonucleótido molde cerca de su extremo 3', y 45
(iv) la incorporación de una combinación de uno o más sitios abásicos y uno o más desapareamientos de nucleótidos en la región 3' del oligonucleótido molde cerca de su extremo 3'.
11. El procedimiento de acuerdo con la reivindicación 10, en el que el uno o más restos o análogos de nucleótidos no extensibles se seleccionan entre el grupo que consiste en
(i) un 2', 3' didesoxinucleótido, 50
(ii) un espaciados 3' C3, C18 o de otra longitud,
(iii) un nucleótido 3' fosforilado,
(iv) una base de ácido peptidonucleico,
(v) un engarce amina,
(vi) uno o más uracilos tratados con uracil ADN glucosilasa,
(vii) ARN, 5
(viii) uno o más restos de 2' O-metil ARN, y
(ix) cualquier combinación de (i)-(viii).
12. El procedimiento de acuerdo con una cualquiera de las reivindicaciones 6-9, en el que una modificación en la región 3' que permite la extensión 3' pero impide o bloquea el copiado en la región 3' de dicho oligonucleótido se selecciona entre el grupo que consiste en 10
(i) la inserción dentro de la región 3' del oligonucleótido molde de uno o más análogos de bases,
(ii) la inserción de uno o más nucleótidos de ARN,
(iii) la inserción de uno o más sitios abásicos, y
(iii) cualquier combinación de (i) - (iii).
13. El procedimiento de acuerdo con la reivindicación 8, en el que los extremos 3' de las moléculas de ácido 15 nucleico en la muestra que incluye el extremo 3' de la molécula de ácido nucleico marcada como diana, son el resultado de la digestión de la muestra con una endonucleasa de restricción cortadora flanqueante, y opcionalmente donde la modificación en la región 3' que bloquea la extensión 3' de dicho oligonucleótido molde es la incorporación de uno o más análogos de bases, uno o más sitios abásicos o cualquier combinación de éstas en su extremo 3'.
14. El procedimiento de acuerdo con la reivindicación 13, en el que el oligonucleótido molde incorpora 20 adicionalmente una modificación en la región 3' que bloquea la extensión 3' de la secuencia diana.
15. El procedimiento de acuerdo con la reivindicación 14, en el que la modificación es la incorporación en la región 3' del oligonucleótido molde de uno o más análogos de bases.
16. El procedimiento de acuerdo con la reivindicación 15, en el que el oligonucleótido molde incorpora un desapareamiento de nucleótido 3' terminal, uno o más sitios abásicos y la incorporación de uno o más análogos de 25 bases.
17. El procedimiento de acuerdo con una cualquiera de las reivindicaciones 1-16, en el que el segundo oligonucleótido es un oligonucleótido molde adicional.
18. Un kit para la amplificación selectiva de ADN escindido de una muestra de ADN que comprende ADN escindido y no escindido, comprendiendo el kit (i) un oligonucleótido molde, (ii) un segundo oligonucleótido, y (iii) un tercer 30 oligonucleótido; definido de acuerdo con el procedimiento de una cualquiera de las reivindicaciones 1-17.
19. El procedimiento de acuerdo con la reivindicación 4 ó 9, en el que la endonucleasa de restricción es una endonucleasa de restricción sensible a metilación específica de secuencia, seleccionada entre el grupo que consiste en HpaII, HhaI, BstuI, NotI, SmaI, SacII, GlaI y BisI.
20. El procedimiento de acuerdo con la reivindicación 3 o reivindicación 8, para detectar la presencia o ausencia de 35 una deleción o inserción en un ácido nucleico en una muestra,
en el que la molécula de ácido nucleico en la muestra se digiere con una enzima de restricción que tiene un sitio de escisión fuera de su sitio de reconocimiento,
y en el que la deleción o inserción sucede entre el sitio de reconocimiento y el sitio de escisión de la enzima de restricción. 40
21. El procedimiento de acuerdo con la reivindicación 4 ó 9, en el que la endonucleasa de restricción se elige para distinguir entre la presencia o ausencia de un sitio de restricción en las moléculas de ácido nucleico de la muestra.
22. El procedimiento de una cualquiera de las reivindicaciones 1 a 21 usado para la detección de un polimorfismo o mutación de un único nucleótido, para la detección de inestabilidad en repeticiones de nucleótidos simples, o para la determinación del estado de metilación de una molécula de ácido nucleico. 45
Patentes similares o relacionadas:
DETERMINACIÓN DEL GRADO DE METILACIÓN DE ADN, del 23 de Noviembre de 2011, de HEINRICH-HEINE-UNIVERSITÄT DÜSSELDORF: Procedimiento para determinar el grado de metilación de ADN normalizado que comprende las etapas: a) determinación cuantitativa de la presencia de un transposón […]
PROCEDIMIENTO DE OBTENCION DE UN DISPOSITIVO PARA DIAGNOSTICO DE MUTACIONES FRECUENTES DE LA PROTEINA LRRK2 BASADO EN PCR EN TIEMPO REAL, del 1 de Julio de 2011, de UNIVERSIDAD DE EXTREMADURA FUNDESALUD: La presente invención se refiere a un procedimiento de obtención de un dispositivo para diagnóstico de mutaciones frecuentes de la proteína Irrk2 […]
CONJUNTO DE CEBADORES, SONDAS, PROCEDIMIENTO Y KIT PARA EL GENOTIPADODEL POLIMORFISMO GENETICO -250G/A DEL GEN LIPC, del 24 de Junio de 2011, de UNIVERSIDAD DE MALAGA: Conjunto de cebadores, sondas, procedimiento y kit para el genotipado del polimorfismo genético -250G/A del gen LIPC.La presente invención se refiere a un conjunto de cebadores, […]
 MÉTODOS PARA EL RÁPIDO ANÁLISIS FORENSE DE ADN MITOCONDRIAL Y CARATERIZACIÓN HETEROPLASMIA DE ADN MITOCONDRIAL, del 16 de Febrero de 2011, de IBIS BIOSCIENCES, INC: Un método forense que comprende los pasos de: (i) determinar una primera masa molecular de un primer producto de amplificación de un Cebador amplicón identificador […]
MÉTODOS PARA EL RÁPIDO ANÁLISIS FORENSE DE ADN MITOCONDRIAL Y CARATERIZACIÓN HETEROPLASMIA DE ADN MITOCONDRIAL, del 16 de Febrero de 2011, de IBIS BIOSCIENCES, INC: Un método forense que comprende los pasos de: (i) determinar una primera masa molecular de un primer producto de amplificación de un Cebador amplicón identificador […]
CONJUNTO DE CEBADORES, SONDAS, PROCEDIMIENTO Y KIT PARA EL GENOTIPADODEL POLIMORFISMO GENETICO S447X DEL GEN LPL, del 27 de Enero de 2011, de UNIVERSIDAD DE MALAGA: Conjunto de cebadores, sondas, procedimiento y kit para el genotipado del polimorfismo genético S447X del gen LPL.La presente invención se refiere a un conjunto de […]
GENERACION DE MUESTRAS PARA GENOTIPAR MEDIANTE ESPECTROMETRIA DE MASAS, del 29 de Octubre de 2010, de CENTRE NATIONAL DE GENOTYPE: Método para el genotipado de polimorfismos de un nucleótido único (SNP) mediante espectrometría de masas, que comprende las etapas siguientes: a. […]
 DETECCION DE LA UNION DE RECOMBINACION DE ADN, del 6 de Octubre de 2010, de CEPHEID: Un método para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión en una secuencia polinucleotídica […]
DETECCION DE LA UNION DE RECOMBINACION DE ADN, del 6 de Octubre de 2010, de CEPHEID: Un método para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión en una secuencia polinucleotídica […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]