Procedimiento para identificar un contenido porcino en un alimento.
Un procedimiento para identificar un contenido porcino en un alimento,
en el que el procedimiento incluye las etapas de:
a) extraer el ácido desoxirribonucleico (ADN) de una muestra de carne de cerdo;
b) diseñar un cebador directo y de un cebador inverso en base al gen mitocondrial ND5 de la carne de cerdo; y
c) realizar una prueba de reacción en cadena de la polimerasa (PCR) en los cebadores directo e inverso;
caracterizado por que:
la secuencia del cebador directo es SUS-FWD: 5'-AGC TGC ACT AGA AGC AAT CC-3')
y la secuencia del cebador inverso es SUS-RVS: 5'-ATG CGT TTG AGT GGG TTA GG-3'.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/MY2009/000047.
Solicitante: Universiti Putra Malaysia.
Nacionalidad solicitante: Malasia.
Dirección: UPM SERDANG SELANGOR DARUL EHSAN 43400 MALASIA.
Inventor/es: B. CHE MAN,YAAKOB, MUSTAFA,SHUHAIMI, KHALID,FARIHAH LIYANA, AZMI,AIDA AZRINA, SAZILI,AWIS QURNI, ABDUL RAHIM,RAHA.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/06 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Células animales.
- C12N15/10 C12N 15/00 […] › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
PDF original: ES-2448585_T3.pdf
Fragmento de la descripción:
Procedimiento para identificar un contenido porcino en un alimento.
CAMPO DE LA INVENCIÓN
La invención se refiere a un procedimiento para diseñar cebadores para la identificación de ingredientes alimentarios, especialmente alimento procesado basado en carne. En particular, el procedimiento sirve para identificar la presencia de cerdo (Sus scrofa) en un alimento procesado para la autentificación Halal.
ANTECEDENTES DE LA INVENCIÓN
La adulteración de alimentos es una cuestión común a nivel mundial. Por ejemplo, se usaron carnes más baratas como un sustituto para carnes más costosas. Más frecuentemente, se ha usado carne de cerdo para sustituir otros tipos de carne en productos alimentarios. Por lo tanto, la identificación de especies animales, especialmente cerdo en productos alimentarios, se está convirtiendo en una cuestión importante para los consumidores. La incidencia de alteración de la etiquetación de alimentos puede ser mucho más importante con respecto a la presencia de alimento potencialmente no Halal. Por esta razón, se han desarrollado varios procedimientos para identificar la especie de origen de la carne fresca y de los productos cárnicos. Se han empleado en la industria alimentaria numerosos procedimientos basados en análisis de ADN para controlar adulteraciones de los productos alimentarios. Los procedimientos establecidos para especiación animal se basan principalmente en lípidos, proteínas y ADN. Sin embargo, los procedimientos basados en ADN son particularmente más fiables puesto que el ADN es más estable en condiciones asociadas a las altas temperaturas, presiones y tratamiento químico usado en el procesamiento de alimentos.
La identificación de especies de tejidos animales en productos cárnicos es una cuestión importante para proteger al consumidor de la adulteración ilegal o indeseable; por razones económicas, religiosas y de salud. Para este fin, se han desarrollado numerosos procedimientos analíticos basados en análisis de proteínas y ADN. Entre los procedimientos basados en ADN, que se desarrollan en gran medida para la identificación de especies están PCR convencional específica de la especie y PCR en tiempo real. Entre los fragmentos génicos diana desarrollados para PCR específica de especie de cerdo están aquellos obtenidos a partir de 12S de ARNr ND5, bucle de desplazamiento mitocondrial (D-Loop) y el receptor 1 de Melanocortina Nuclear (MCIR) .
Aunque se demostró que el procedimiento que utiliza PCR convencional fue un éxito, requiere una manipulación post-PCR que prolonga el tiempo de análisis y la manipulación de productos químicos peligrosos puede provocar contaminación en el laboratorio. Por otra parte, los procedimientos de PCR en tiempo real poseen un gran potencial para reemplazar la PCR convencional. Esto es principalmente debido a que los procedimientos por PCR en tiempo real son rápidos, sensibles, específicos, de alto grado de automatización y de cuantificación de diana (Heid y col., 1996) .
RESUMEN DE LA INVENCIÓN
La presente invención se refiere a un procedimiento para identificar un contenido de cerdo en un alimento, donde el procedimiento incluye las etapas de extraer el ácido desoxirribonucleico (ADN) de una muestra de cerdo y diseñar un cebador directo y un cebador inverso en base al gen mitocondrial ND5 del cerdo realizando una prueba de reacción en cadena de la polimerasa (PCR) en los cebadores directo e inverso, caracterizado porque la secuencia del cebador directo es SUS-FWD: 5'-AGC TGC ACT ACA AGC AAT CC-3') y la secuencia del cebador inverso es SUS-RVS: 5'-ATG CGT TTG AGT GGG TTA GG-3'.
BREVE DESCRIPCIÓN DEL DIBUJO
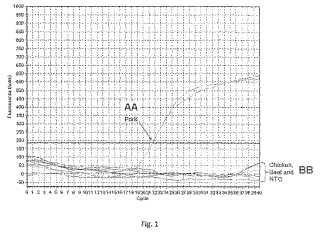
La figura 1 muestra la prueba de especificidad en el cebador de carne de cerdo diseñada contra otras especies de carne (ternera y pollo) .
La figura 2 muestra la prueba de sensibilidad del cebador de carne de cerdo con diluciones en serie de 10 veces.
La figura 3 muestra la optimización de la concentración del cebador.
La figura 4 muestra la optimización de la temperatura de atemperado del cebador.
DESCRIPCIÓN DETALLADA DE LA INVENCIÓN
La invención describe el desarrollo y aplicación de un ensayo por PCR en tiempo real específico para cerdo para la autentificación Halal. Se diseñan cebadores para amplificar un amplicón de 89 pb del gen mitocondrial de ND5 (ND5) de cerdo y se desparejaron de especies comerciales de pollo y ternera. El ensayo es altamente sensible y detectó la presencia de 0, 001 ng de ADN de plantilla de cerdo cuando se valoró usando diluciones de ADN en agua. El conjunto de cebadores desarrollado para la verificación del producto Halal se basa en el gen mitocondrial ND5 de cerdo y la secuencia de los cebadores es como se indica a continuación:
Cebador directo (SUS-FWD: 5'-AGC TGC ACT ACA AGC AAT CC-3')
Cebador inverso (SUS-RVS: 5'-ATG CGT TTG AGT GGG TTA GG-3')
La condiciones PCR en la detección de ADN de carne de cerdo se hacen por la amplificación en el Mastercycler ep (Eppendorf AG, Hamburgo, Alemania) . Cada tubo de reacción contiene 20 !l de mezcla de reacción que consiste en 10 !l de 2x Quantitect SYBR Green PCR Master Mix (Qiagen, Hilden, Alemania) , 1 !l de cebador directo, 1 !l de cebador inverso, 3 !l de dH2O y 5 !l de muestra de ADN (20 ng/!l) . El programa de ciclo de amplificación de 3 etapas es como se indica a continuación: activación inicial a 95 ºC durante 15 min, desnaturalización a 94 ºC durante 15 s, atemperado a 58 ºC durante 30 s y extensión a 72 ºC durante 30 s. La etapa de activación inicial sirve para activar la Polimerasa de ADN HotStarTaq presente en la mezcla de reacción. El ciclo se repite 40 veces y se realizó un análisis de curva de fusión para verificar la especificidad y la identidad del ADN amplificado.
Muestras [0009] Las tres especies que se usan en este estudio son carne de cerdo, de ternera y de pollo. Las muestras de carne se adquieren en un mercado local, el mercado al por mayor Selangor. Se almacenan a -20 ºC hasta su uso para la extracción de ADN.
Extracción de ADN
Se extrae el ADN de las muestras de carne usando DNeasy® Blood and Tissue Kit (Qiagen, Hilden, Alemania) . Se pesan 25 mg de muestra en un tubo de microcentrífuga de 1, 5 ml. Se añaden 180 !l de Tampón ATL y 20 !l de K proteinasa. La mezcla se agita vorticialmente y después se incuba durante una noche a 56 ºC en baño de agua para lisis. Cuando las muestras se lisan completamente, se añaden 4 !l de RNasa A (100 mg/ml) , se mezclan y se incuban a temperatura ambiente durante 2 min. La mezcla se agita vorticialmente antes de añadir 200 !l de Tampón AL y después se agita vorticialmente de nuevo vigorosamente. Después, se añaden 200 !l de etanol (96-100%) y se mezclan vorticialmente para producir una solución homogénea. La mezcla se pipetea en el conjunto de columna giratoria DNeasy Mini y se centrifuga a 8.000 rpm durante 1 min. El flujo saliente y el tubo de recogida se desechan. Después, la columna giratoria DNeasy Mini se coloca en un nuevo tubo de recogida de 2 ml. Se añaden 500 !l de Tampón AW2 y se centrifuga a 14.000 rpm durante 3 minutos para asegurar que la columna está seca y no se presente arrastre de etanol. La columna giratoria DNeasy Mini se coloca entonces en un nuevo tubo de microcentrífuga de 1, 5 ml y se añaden 100 !l de Tampón AF para la elución. El tubo se incuba durante 1 min a temperatura ambiente y después se centrifuga durante 1 min a 8.000 rpm. El sobrenadante que contiene el ADN extraído se almacena a 4 ºC antes del siguiente uso.
Diseño del cebador
Se utiliza el software Primer3 (http://frodo.wi.mit.edulcgi-bin/primer3/primer3_www.cgi) para diseñar el conjunto de cebadores usado en el análisis por PCR en tiempo real. Las secuencias del gen ND5 de la carne de cerdo (NC_000845) , la ternera (NC_006853) y el pollo (NC_001323) obtenidas a partir de la base de datos del NCBI GenBank (http://www.ncbi.nlm.nih.gov) , se alinean y se comparan. Se sintetizó un conjunto de cebadores (SUS-FWD: 5'-AGC TGC ACT ACA AGC AAT CC-3' y SUS-RVS: 5'-ATG CGT TTG AGT GGG TTA GG-3') para amplificar expresamente un fragmento de 89 pb del gen ND5 de carne de cerdo.
Análisis por PCR en tiempo real
La detección de ADN de la carne de cerdo se hace por la amplificación en el Mastercycler ep (Eppendorf AG, Hamburgo, Alemania) . Cada tubo de reacción contiene 20 !l de mezcla de reacción que consiste en 10 !l de 2x Quantitect SYBR Green PCR Master Mix (Qiagen, Hilden, Alemania) , 1 !l de cebador directo, 1 !l de cebador inverso, 3 !l de dH2O y 5 !l de muestra de ADN. El programa de ciclo de amplificación de 3 etapas es como se indica a continuación: activación inicial a 95 ºC durante 15 min, desnaturalización... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento para identificar un contenido porcino en un alimento, en el que el procedimiento incluye las etapas de:
a) extraer el ácido desoxirribonucleico (ADN) de una muestra de carne de cerdo; b) diseñar un cebador directo y de un cebador inverso en base al gen mitocondrial ND5 de la carne de cerdo; y
c) realizar una prueba de reacción en cadena de la polimerasa (PCR) en los cebadores directo e inverso; caracterizado por que: la secuencia del cebador directo es SUS-FWD.
5. AGC TGC ACT AGA AGC AAT CC-3') y la secuencia del cebador inverso es SUS-RVS.
5. ATG CGT TTG AGT GGG TTA GG-3'.
2. El procedimiento como se ha indicado en la reivindicación 1, en el que la concentración de los cebadores directo e inverso está comprendida entre 0, 3 y 0, 9 !m.
3. El procedimiento como se ha indicado en la reivindicación 1, en el que la temperatura de reacción está comprendida entre 50 ºC y 70 ºC.
4. Un procedimiento para la identificación de un contenido porcino en un alimento, en el que el procedimiento incluye las etapas de: a) diseñar un cebador directo y un cebador inverso en base al gen mitocondrial ND5 de la carne de cerdo; y b) realizar una prueba de reacción en cadena de la polimerasa (PCR) en los cebadores directo e inverso;
caracterizado por que: la secuencia del cebador directo es SUS-FWD.
5. AGC TGC ACT ACA AGC AAT CC-3') y la secuencia del cebador inverso es de SUS-RVS.
5. ATG CGT TTG AGT GGG TTA GG-3'.
5. Procedimiento como se ha indicado en la reivindicación 4, en el que la concentración de los cebadores directo e inverso está comprendida entre 0, 3 y 0, 9 !m.
6. Procedimiento como se ha indicado en la reivindicación 4, en el que la temperatura de reacción está comprendida entre 50 ºC y 70 ºC.
Patentes similares o relacionadas:
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Método para romper un ácido nucleico y añadir un adaptador por medio de transposasa y reactivo, del 1 de Julio de 2020, de MGI Tech Co., Ltd: Un metodo para romper un acido nucleico y anadir un adaptador por medio de una transposasa, que comprende las siguientes etapas: interrumpir […]
Vacuna subunitaria contra Mycoplasma spp., del 1 de Julio de 2020, de Agricultural Technology Research Institute: Una composición para prevenir una infección por Mycoplasma spp., que comprende: un principio activo, que comprende una proteína de PdhA; y un adyuvante […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Preparación de bibliotecas de ácido nucleico marcado usando protocolo de adición en un solo tubo, del 13 de Mayo de 2020, de ILLUMINA, INC: Un método para preparar una biblioteca de fragmentos de ácido nucleico marcados que comprende: (a) poner en contacto una célula individual […]
Proteínas de captura de la superficie celular recombinantes, del 13 de Mayo de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para detectar y aislar células que producen altos niveles de una proteína heterodimérica que tiene una primera subunidad y una segunda subunidad, […]
Etiquetado y evaluación de una secuencia diana, del 13 de Mayo de 2020, de RhoDx, Inc: Un método para modificar un ácido nucleico, que comprende: (a) poner en contacto un ácido nucleico de cadena sencilla con una actividad de transferasa […]